手把手教学!XGBoost如何调整最优参数预测生存期
点击蓝字 关注我们
小伙伴们大家好啊,我是大海哥,前几天有小伙伴询问,可不可以出一个XGBoost预测生存期的教学,然后教一下调参啊!
安排~
今天大海哥将使用基因表达数据结合临床数据,来一期XGBoost调整最优参数并预测生存期的教学。首先分析之前,我们先给不太了解这个算法的小伙伴们科普一下,什么是XGBoost?这个算法有什么过人之处?
XGBoost(eXtreme Gradient Boosting)是一种集成学习算法,主要用于解决分类和回归问题。它是基于梯度提升树(Gradient Boosting Tree)的机器学习算法,在各种数据科学竞赛和实际应用中都表现出色。以下是其主要特点:
梯度提升树(Gradient Boosting Tree)基础:XGBoost是建立在梯度提升树算法之上的。梯度提升树通过迭代地训练多个弱学习器(通常是决策树),每个弱学习器都试图纠正前一个弱学习器的错误。
正则化:XGBoost在模型训练过程中引入了正则化技术,防止过拟合。通过控制模型的复杂度,XGBoost能够在训练数据上取得很好的性能,并且在未见过的数据上也能表现出色。
优化目标函数:XGBoost采用自定义的目标函数,该目标函数包括预测误差和正则化项,以便在训练过程中更好地优化模型。
处理缺失值:XGBoost能够自动处理缺失值,在树的构建过程中,会自动考虑缺失值所在的分支。
不仅如此,该算法还支持并行加速哦!多线程欸!简直完美
好了,介绍完毕,让我们开始代码部分吧!
#首先导入我们需要的R包library(xgboost)library(survival)#然后就是读取数据ml<-read.csv("ML_data.csv",header=T,row.names=1,check.names=F)
#定义我们的数据Dtrain <- xgb.DMatrix(data = as.matrix(ml[,c(3:79)]), label = ml$time)#然后我们需要定义一个参数范围,让模型挑选出其中最优的值max_depth. <- c(3:11)min_child_weight. <- c(3:18)#先随便定义一个参数列表best_param=list(objective = "survival:cox",eval_metric = "cox-nloglik",max_depth = 5,eta = 0.01,gamma = 0,subsample = 0.8,colsample_bytree = 0.8,min_child_weight = 2,lambda=1,alpha=0)
看一下数据的样子
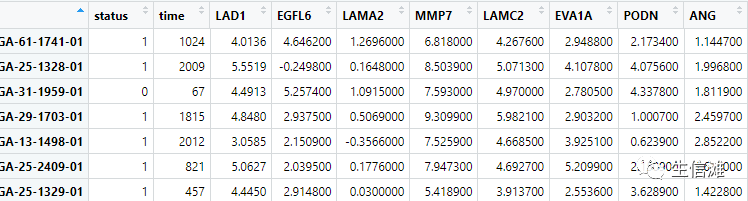
#然后就可以开始迭代啦!#在深度和子节点的范围内迭代for (a in max_depth.){for (b in min_child_weight.){print("max_depth, min_child_weight")print(a)print(b)#定义一个参数列表,一会就拿来和自己定义的最优参数列表比较param <- list(objective = "survival:cox",eval_metric = "cox-nloglik",max_depth = a,eta = 0.01,gamma = 0,subsample = 0.8,colsample_bytree = 0.8,min_child_weight = b,lambda=1,alpha=0)
#定义交叉验证的参数cv.nround = 5000cv.nfold = 5#设置一个种子随机数set.seed(seed.number)#开始交叉验证啦,这一步就是来测试参数哦!mdcv <- xgb.cv(data=Dtrain,params = param,nthread=10,nfold=cv.nfold,nrounds=cv.nround,watchlist,verbose = T,early_stopping_rounds=30,maximize=FALSE )if("try-error" %in% class(mdcv)){next}
#开始判断阈值并保留较优结果min_loss = min(mdcv$evaluation_log[,'test_cox_nloglik_mean'])min_loss_index = which.min(as.numeric(unlist(mdcv$evaluation_log[,'test_cox_nloglik_mean'])))#保存最终的最优结果if (min_loss < best_loss) {best_loss = min_lossbest_loss_index = min_loss_indexbest_param = paramwrite.table(best_loss, file = "best_loss.txt", append=F)write.table(best_loss_index, file = "best_loss_index.txt", append=F)write.table(best_param, file = "best_param.txt", append=F)}write.table(a,file='max_depth.txt',append=FALSE)write.table(b,file='min_child_weight.txt',append=FALSE)}}
开始交叉验证,这一步时间有点久哦!
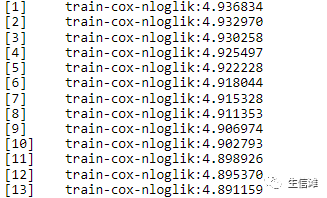
至此,我们就得到了最优参数
来看看大海哥得到的最优结果吧!

再看一下具体都是什么类型的吧

细心的小伙伴发现了,怎么只是挑选了最优的两个参数,其他的参数呢?
是的,其他的参数也需要我们来不断迭代测试,方式和上面的一样哦!只需要修改一下变量就好啦!
#然后我们就可以使用最优参数来构建模型啦model_xgb <- xgboost(data=Dtrain, params=best_param, nrounds=i, nthread=10)result <- list()#这里就要选择测试集了,定义方式和训练集一样哦!xgb_preds <- predict(model_xgb, newdata = Dtest, type = "survival")result$pred = xgb_predspred_xgb <- as.numeric(xgb_preds)result$best_param = best_paramresult$nround = nroundresult$model = model_xgb#最后可以获取我们的C1指数啦,大家也可以使用其他方法获取别的指标哦!c1=survConcordance(Surv(ml$time, ml$status) ~ pred_xgb)
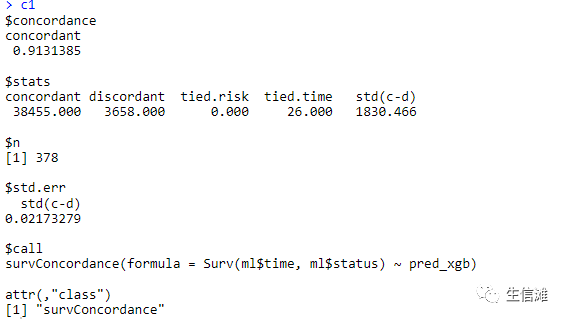
结果不错,大家也可以试试看哦!说不定会有更好的结果哦!

点击“阅读原文”进入网址
