还不知道TCGA数据如何下载处理?快跟上小果步伐去学习!!
 想要分析肿瘤数据的小伙伴,都少不了TCGA数据的使用,这个数据库呢全名是癌症基因组图谱计划。.作为目前最大的癌症基因信息数据库,包含了许多肿瘤的数据,所以小伙伴都很想用TCGA数据去做分析,但是刚入手不知道从哪里下载,小果这里教大家如何去下载并使用R语言去处理数据。
想要分析肿瘤数据的小伙伴,都少不了TCGA数据的使用,这个数据库呢全名是癌症基因组图谱计划。.作为目前最大的癌症基因信息数据库,包含了许多肿瘤的数据,所以小伙伴都很想用TCGA数据去做分析,但是刚入手不知道从哪里下载,小果这里教大家如何去下载并使用R语言去处理数据。
这里小果使用UCSC xena数据库 这个数据库包含又TCGA数据库肿瘤信息,并且数据已经整理好,最重要的是他是开源的!!来看一下:#xena官网
#https://xenabrowser.net/datapages/ 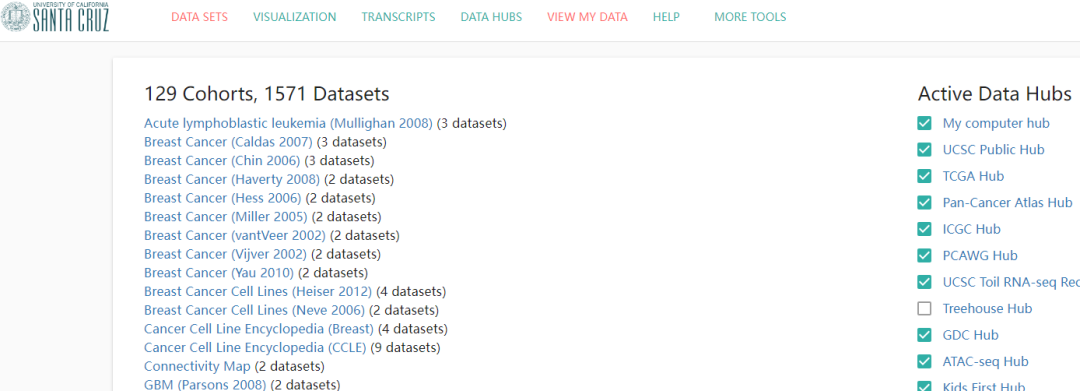
这是他的主页面 这里小果使用宫颈癌示例数据去演示,宫颈癌在TCGA数据库中是CESC我们找到这个 点击进入
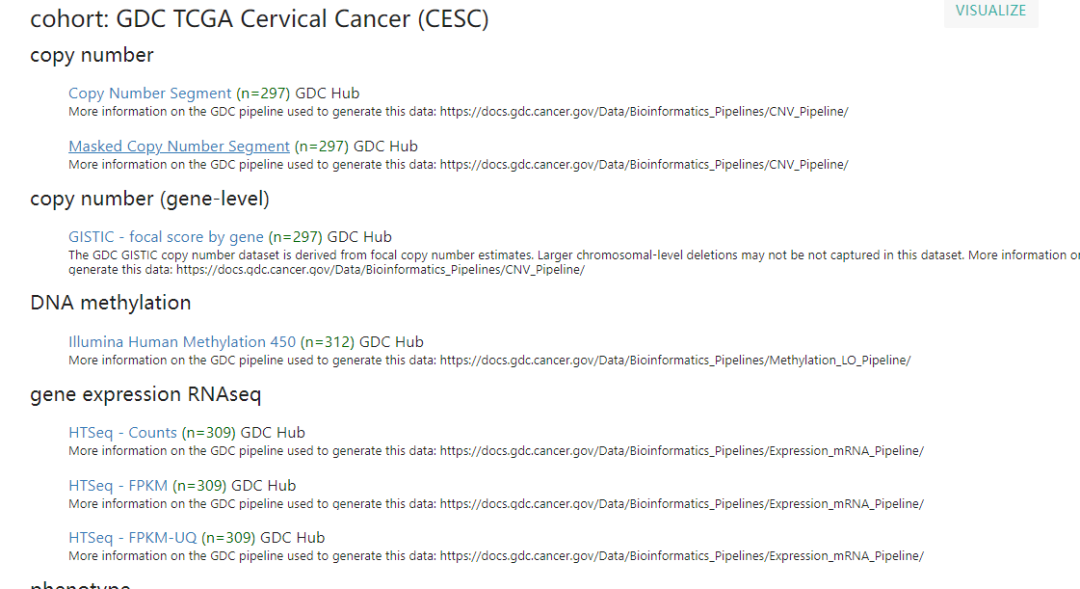
这里面就是他的数据了,我们主要使用的呢就是counts数据还以FPKMS数据,这里就展示Counts数据的处理,FPKM数据和counts数据处理形式是一样的,
 我们点击进入
我们点击进入
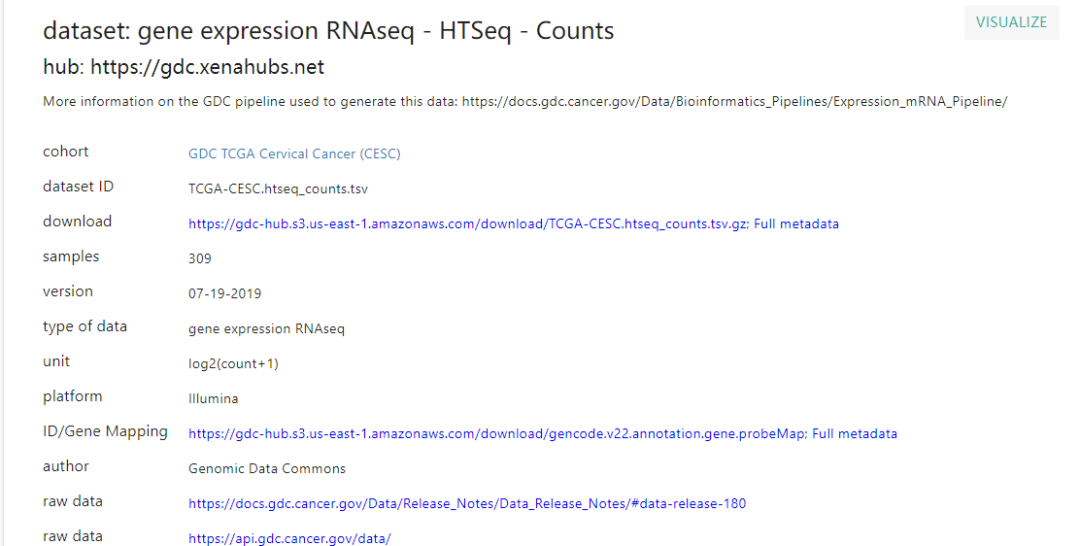
这里就会有他的信息,我们下载就会得到


这样一个表达文件。下面我们就去处理这个数据吧!

公众号回复111获取代码,代码编号:231107
#install.packages("tidyverse")library(tidyverse)#每次重新打开R都要library一下##文件的读取#读取tsv文件counts1 = read.table(file = 'TCGA-CESC.htseq_counts.tsv', sep = 't', header = TRUE)
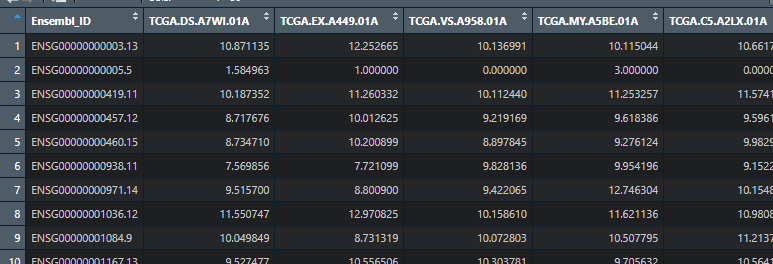
得到一下的矩阵
我们需要对矩阵进行处理,将第一列放在文本框中
rownames(counts1) <- counts1[,1] #Alt <-#x <- counts1[,1:3]counts1 = counts1[,-1]
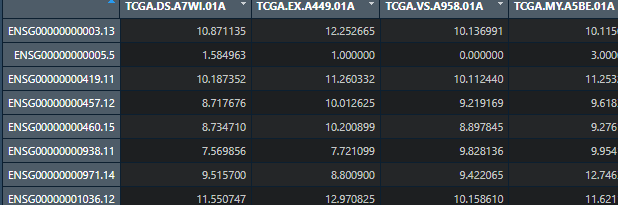
接下来就是数据处理,我们要得到肿瘤的样本还以正常的样本数据,含有01A的是肿瘤样本,11A的为正常,因此我们使用下面table函数去寻找并计算,
#table函数table(substr(colnames(counts1),14,16))#c("01A","11A")#%in%符号用于判断是否属于counts1 <- counts1[,substr(colnames(counts1),14,16)%in% c("01A","11A")]table(substr(colnames(counts1),14,16))#保留列名前15位rownames(counts1) <- substr(rownames(counts1),1,15)ceiling(1.2)ceiling(3.8)counts <- ceiling(2^(counts1)-1)
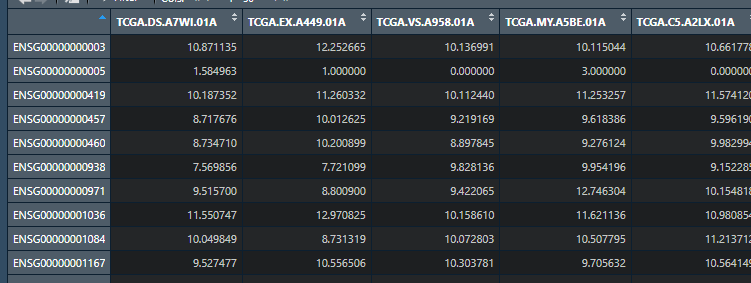
这样一来列的名称中含有小数点的数字没有了,这样方便我们去对照寻找基因名
我们将
write.table(counts,"counts.txt",sep = "t",row.names = T,col.names = NA,quote = F)那么我们有了数据,肯定要做差异表达,下面小果带大家去学习对上述矩阵进行差异表达分析。counts <- read.table("counts.txt",sep = "t",row.names = 1,check.names = F,stringsAsFactors = F,header = T)Ginfo_0 <- read.table("gene_length_Table.txt",sep = "t",check.names = F,stringsAsFactors = F,header = T,row.names = 1)
就是上述注释文件,包含我们想要的基因信息
我们只要RNA信息,并且找到Gene名替换矩阵前面的第一列信息:
Ginfo <- Ginfo_0[which(Ginfo_0$genetype == "protein_coding"),] #只要编码RNA#美元符号代表提取列#取行名交集intersectcomgene <- intersect(rownames(counts),rownames(Ginfo))counts <- counts[comgene,]#class(counts)#判断数据类型#class(comgene)Ginfo <- Ginfo[comgene,]a <- rownames(counts)b <- rownames(Ginfo)identical(a,b)#判断是否相同counts$Gene <- as.character(Ginfo$genename) #新增Gene Symbolcounts <- counts[!duplicated(counts$Gene),] #去重复rownames(counts) <- counts$Gene #将行名变为Gene Symbolcounts <- counts[,-ncol(counts)] #去除最后一列write.table(counts, file = "CESC_counts_mRNA_all.txt",sep = "t",row.names = T,col.names = NA,quote = F)这里我们#保存癌症患者的countstumor <- colnames(counts)[substr(colnames(counts),14,16) == "01A"]counts_01A <- counts[,tumor]#并且找到只要肿瘤的矩阵,为了我们后续差异分析用write.table(counts_01A, file = "CESC_counts_mRNA_01A.txt",sep = "t",row.names = T,col.names = NA,quote = F)
下面开始我们的差异分析吧!
#差异分析library(tidyverse)#安装BiocManager#if(!require(DESeq2))BiocManager::install('DESeq2')library(DESeq2)#这里我们用这个包DESeq2,做差异分析的包后面就跟着小果代码跑跑跑就行了counts = counts[apply(counts, 1, function(x) sum(x > 1) > 32), ]conditions=data.frame(sample=colnames(counts),group=factor(ifelse(substr(colnames(counts),14,16) == "01A","T","N"),levels = c("N","T"))) %>%column_to_rownames("sample")dds <- DESeqDataSetFromMatrix(countData = counts,colData = conditions,design = ~ group)dds <- DESeq(dds)#这一步有点慢,小伙伴多多等待不要动resultsNames(dds)res <- results(dds)save(res,file = "CESC_DEG.rda")#一定要保存!res_deseq2 <- as.data.frame(res)%>%arrange(padj) %>%dplyr::filter(abs(log2FoldChange) > 4, padj < 0.05)#根据自己需要

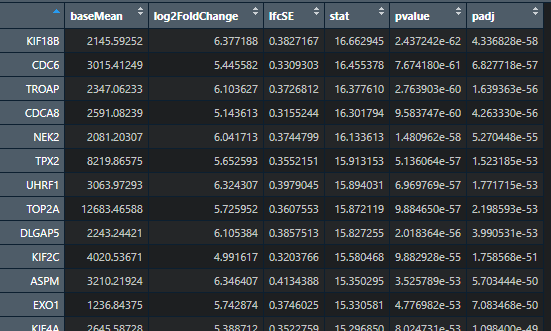
这样结果就跑完了,我们得到基因数1194,小伙伴觉得差异基因太少或者太多,自行去修改
##dplyr::filter(abs(log2FoldChange) > 4, padj < 0.05)#根据自己需要这里调整阈值的选择。一定在合理范围之内以上就是我们对TCGA数据的处理了,小伙伴有没有学会呢,多多理解代码的意义,不要跑错了
往期推荐

