干货!批量处理泛癌相关生存KM分析绘图
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

还在为如何入门生信而彷徨吗?还在为Linux系统而感到无助吗?快关注小果的微信公众号,小果手把手带你入门生信,没有门槛、没有难度,跟着效果走,啥问题都没有。
生存分析中的Kaplan-Meier曲线(也称为KM曲线)是一种常用的统计方法,用于估计不同组别之间的生存函数差异。KM曲线显示了在给定时间范围内生存下来的患者比例随时间的变化情况。
KM曲线通常用于以下情况:
比较不同治疗组或治疗方案的生存差异:例如,对于某种癌症,可以将患者分为接受不同治疗方案的组,并比较各组的生存情况。
评估预后因素对生存的影响:例如,研究特定基因变异与患者生存之间的关系,或者评估其他临床特征(如年龄、性别、肿瘤分期等)对生存的影响。
绘制KM曲线的步骤如下:
收集生存数据:包括每个个体的生存时间和事件状态(如生存或死亡)。
根据研究目的和变量特征,将个体分为不同的组别(例如治疗组和对照组)。
使用Kaplan-Meier方法计算每个组别中在给定时间点生存下来的患者比例。
绘制KM曲线:将不同组别在给定时间点的生存比例绘制在同一张图上。
进行统计检验:使用log-rank检验或其他合适的方法比较不同组别之间的生存差异。
library(limma)library(survival)library(survminer)library(data.table)library(tidyverse)library(ggsignif)library(RColorBrewer)
#首先,加载了一系列所需的R包files=dir("./DFI/")通过dir("./DFI/")获取"./DFI/"目录下的文件列表,这些文件包含了每种癌症类型的生存数据。pFilter=1 #pvalue ALL DATA,NO filter#if(pValue<pFilter){# if(pValue<0.001){# pValue="p<0.001"# }else{# pValue=paste0("p=",sprintf("%.03f",pValue))#}#for tumour types#通过一个循环遍历每个文件进行处理。在循环中,首先读取文件内容并转换为#数据框。然后对数据框进行处理,调整列名和数据格式。将存活时间(futime)#从天转换为年,将tpm转换为数值型数据。for ( i in 1:length(files) ){dirname=files[i]rt=fread(paste0("./DFI/",dirname),header=T,check.names=F)rt=as.data.frame.matrix(rt)
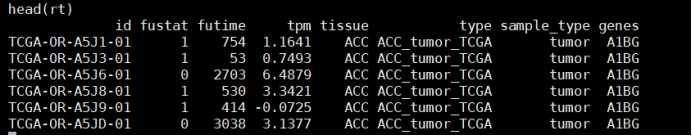
#rownames(rt)=rt[,1]rt=rt[,-1]rt=na.omit(rt)colnames(rt)[1:2]=c("fustat","futime")b=rt[,2]rt=cbind(b,rt)rt=rt[,-3]colnames(rt)[1]="futime"
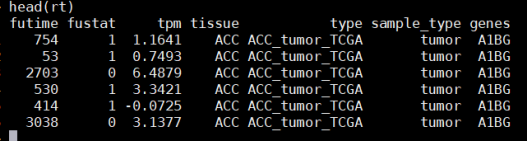
rt$futime=rt$futime/365 #Survival time in yearsrt$tpm=as.numeric(rt$tpm)#rt$tissue=as.factor(rt$tissue)
#######files namestmp<- strsplit(dirname,split=".",fixed=TRUE)dirnam<-unlist(lapply(tmp,head,1))tissue=c("ACC", "BLCA", "BRCA", "CESC", "CHOL", "COAD", "DLBC", "ESCA", "GBM", "HNSC", "KICH", "KIRC", "KIRP", "LGG", "LIHC", "LUAD", "LUSC", "MESO", "OV", "PAAD", "PCPG", "PRAD", "READ", "SARC", "SKCM", "STAD", "TGCT", "THCA", "THYM", "UCEC", "UCS", "UVM")for ( j in 1:length(tissue) ){#for(i in levels(rt[,"tissue"] )){id=rt[1,6]cancer=tissue[j]rt1=rt[(rt$tissue %in% cancer),] # by tumour typegroup=ifelse(rt1[,"tpm"]>median(rt1[,"tpm"]),"high","low")#根据癌症类型进行子集选择,得到特定癌症类型的数据框(rt1)。然后根据tpm的中位数将样本分为高表达组和低表达组,创建一个新的列group。通过判断是否同时包含TRUE和FALSE来排除表达水平相同的样本组。
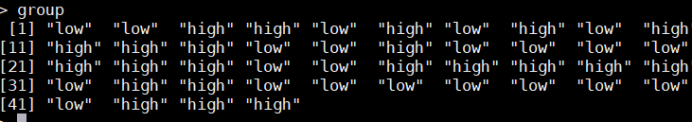
a=rt1[,"tpm"]>median(rt1[,"tpm"])#如果满足条件,接下来进行生存分析。首先使用survdiff函数进行组间生存差异的统计检验,并计算p值。然后使用survfit函数拟合生存曲线。if(length(table(group))==1) {next}if(TRUE & FALSE %in% a){diff=survdiff(Surv(futime,fustat)~ group,data = rt1) #Survival of rt1 data by groupspValue=1-pchisq(diff$chisq,df=1) #testfit <- survfit(Surv(futime,fustat) ~ group, data = rt1)#KM survival curves#接下来,使用ggsurvplot函数生成生存曲线图。这里设置了图形的标题、p值显示、图例、坐标轴标签等参数,并使用不同的颜色表示高表达组和低表达组。#最后,将生成的生存曲线图保存为PDF文件,命名方式为"文件名_癌症类型_p值_sur_DFI.pdf"。surPlot=ggsurvplot(fit,data=rt1,title=paste0("Cancer: ",cancer=tissue[j]),pval=pValue,pval.size=6,legend.labs=c("high","low"),legend.title=paste0(id," levels"),font.legend=12,xlab="Time(years)",ylab="Overall survival",break.time.by = 1,palette=c("red","blue"),conf.int=F,fontsize=4,risk.table=TRUE,risk.table.title="",risk.table.height=.25)pdf(paste0("./picture_DFI/",dirnam,"_",cancer=tissue[j],"_",pValue, "_sur_DFI.pdf"),width = 10, height = 7.5)#ggsave(paste0("./picture_expTime_TG_GTEX/",dirnam,"_",cancer=tissue[j],"_",pValue, "_sur_os.svg"), width = 14, height = 15)print(surPlot)dev.off()}}}#####
KM曲线图通常包括以下信息:时间轴:表示观察时间的横轴,通常以年、月、周等为单位。生存率轴:表示生存患者的比例,纵轴范围从0到1。曲线:每个组别的生存曲线,以实线或虚线表示。标题和图例:用于标识曲线对应的组别和其他重要信息。
通过绘制KM曲线,可以直观地比较不同组别之间的生存差异,帮助研究人员和临床医生了解某种因素对生存的影响,以指导治疗决策和预后评估。有问题可以联系小果。
来给大家展示展示结果图吧,使用firefox命令。
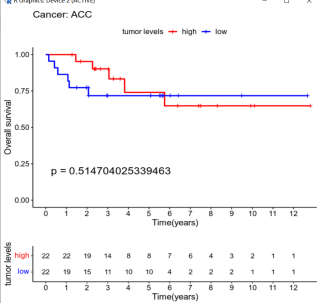
以上就是今天的内容啦!
下期将为你带来更多R语言的骚操作技巧,关注小果一起学习吧!
这里推荐一下小果新开发的零代码云生信分析工具平台包含超多零代码小工具,上传数据一键出图,感兴趣的小伙伴欢迎了解~
网址:http://www.biocloudservice.com/home.html
今天小果的分享就到这里,欢迎大家和小果一起讨论学习,下期再见哦!
小果友情推荐
好用又免费的工具安利


