一文搞定R数据集加载问题
点击蓝字
关注我们
大家好!我是小师妹!很高心跟大家见面啦~最近做数据分析时遇到了一些数据加载问题,在这里给大家分享总结一下。
首先小师妹带大家了解一下SeuratData,SeuratData是一种使用R的内部软件包和数据管理系统以Seurat对象形式分发数据集的机制。它为用户提供了一种访问Seurat小插曲中使用的数据集的简便方法。
我们对于SeuratData的安装可以通过devtools完成。
代码为:
devtools::install_github('satijalab/seurat-data')
选择1更新所有包。

上图显示安装完成。
加载SeuratData时,将显示所有可用数据集的列表(这与其他元数据包(如tidyverse)以及用于创建每个数据集的Seurat版本相似。
使用代码加载SeuratData:
library(SeuratData)
小师妹想要查看所有可用数据集的列表,使用AvailableData函数。
> AvailableData()
对于特定数据集的安装,例如pbmc3k,我们可以使用InstallData。此函数将接受数据集名称或相应的包名称。InstallData会自动附加已安装的数据集软件包,因此可以立即加载和使用数据集。
> InstallData("pbmc3k")我们可以使用data功能完成数据集的加载:
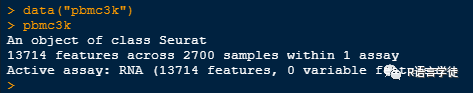
所提供的所有数据集均具有为其构建的帮助页面。可以使用标准help功能访问这些页面。
? pbmc3k小师妹想要在服务器上同样实现数据的加载,但使用InstallData遇到以下问题:

可以这样解决:
在本地Rstudio中下载数据集,得到数据集的网址,之后在服务器上下载,使用代码为
wget http://seurat.nygenome.org/src/contrib/stxBrain.SeuratData_0.1.1.tar.gz --no-check-certificate#--no-check-certificate不检查证书
然后在服务器上加载数据集:
install.packages('/media/desk16/iyun001/project/test/stxBrain.SeuratData_0.1.1.tar.gz',repos = NULL, type = "source")
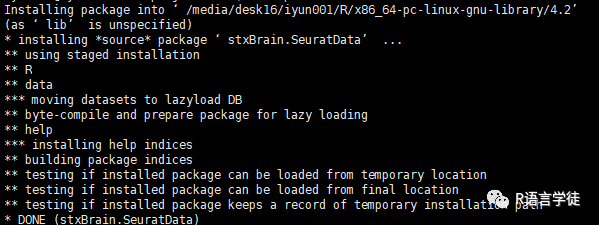
大功告成!再在服务器上加载数据集时就没有问题了。
小师妹的分享就到这里了,欢迎小伙伴们遇到问题来找我讨论啊。
这里小师妹还想推荐一个小工具:单细胞数据绘制小提琴图(http://www.biocloudservice.com/788/788.php),在线运行,可以使用加载的数据来实践哦。

欢迎宝子们跟小师妹一起交流哦~