还在思考预后模型需要那些图?小图告诉你还需要森林图,诺莫图,校准曲线以及DCA决策曲线!!!
点击蓝字
关注小图
不少做癌症肿瘤疾病方向的小伙伴都避免不了要去做预后分析,既然做预后分析,肯定少不了构建预后模型,而有的小伙伴仅仅只是把预后结果做出来,但是却没有绘制图,是因为小伙伴不知道该绘制那些图,去可视化自己结果,而往往就是森林图绘制完就草草了事,但是却忽略了模型的意义以及模型构建是好还是坏,所以,小图这里教小伙伴绘制森林图,诺莫图,校准曲线以及DCA决策曲线,去验证自己构建的预后模型,可视化自己预后模型的结果!下面跟着小图去学习吧!
library(tidyverse)library(regplot)library(rms)library(survminer)library(survival)#我们在这里要调用这几个包,有处理生存数据的,还有对数据形式进行分析的
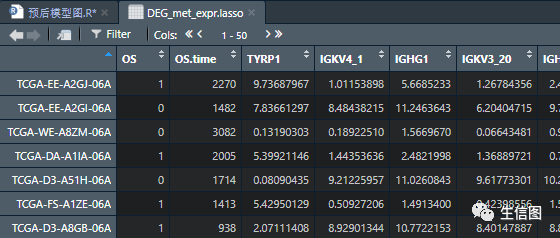
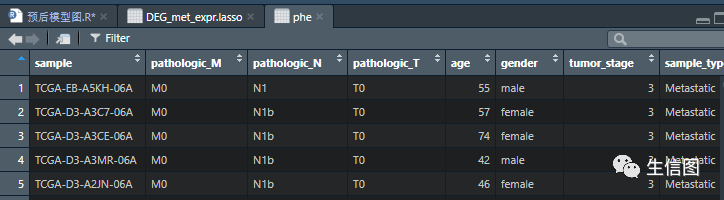
这里小图的示例数据是提前准备好的小伙伴们可以按照数据集形式自己去设置:
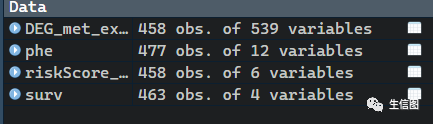
Phe是分期数据等
风险得分对应的生存状态等
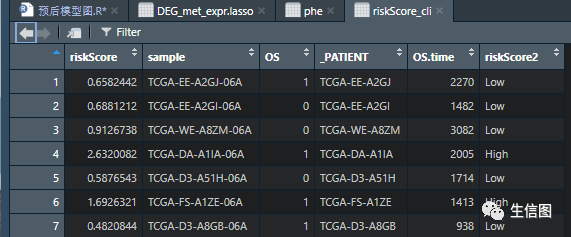
最后是样本对于的生存状态等
数据准备好了我们去绘制把!
我们先构建多因素的森林图
这里我们得到riskscore后,使用多因素COX模型验证其独立性 ,然后使用ggforest函数绘制森林图
riskScore_cli2 <- riskScore_cli %>%inner_join(phe)head(riskScore_cli2)#多因素森林图library(survminer)#构建多因素COXmultiCox <- coxph(Surv(OS.time, OS) ~ age + gender + tumor_stage +riskScore2,data = riskScore_cli2)multiCox#绘制森林图multicox_ggforest <- ggforest(multiCox,data = riskScore_cli2, #数据集main = 'Hazard ratio of multi cox', #标题cpositions = c(0.05, 0.15, 0.35), #前三列距离fontsize = 0.8, #字体大小refLabel = 'reference', #相对变量的数值标签,也可改为1noDigits = 3 #保留HR值以及95%CI的小数位数)multicox_ggforest
上述代码小图都有备注,小伙伴要仔细查看
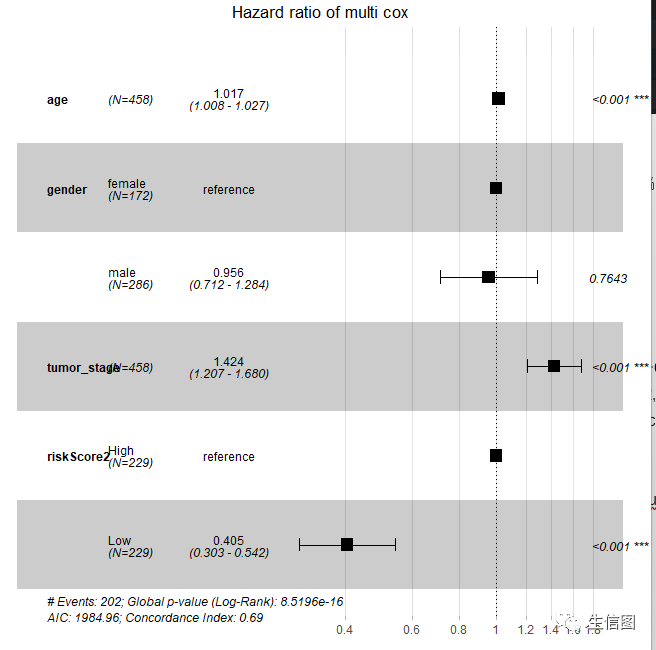
简单的森林图就绘制完成了。
后面就是诺莫图也就是Nomogram,通常也被称为列线图,常用于评估肿瘤学和医学的预后情况。
可将Cox回归的结果进行可视化。
dd=datadist(riskScore_cli2)options(datadist="dd")## 构建COX比例风险模型f <- psm(Surv(OS.time,OS) ~ age + gender + tumor_stage + riskScore2,data = riskScore_cli2,dist='lognormal')surv <- Survival(f) # 构建生存概率函数#nom <- nomogram(f, fun=list(function(x) surv(365, x),function(x) surv(1095, x),function(x) surv(1825, x) ),funlabel=c("1-year OS", "3-year OS","5-year OS"))plot(nom, xfrac=.2)

小伙伴看起来是不是有点单调,看似只有线条一样,
小图这里另一种绘制方法实验regplot绘制
library(regplot)Cox_nomo2 <- cph(Surv(OS.time, OS) ~ age + gender + tumor_stage + riskScore2,data = riskScore_cli2, x=T, y=T)regplot(Cox_nomo2,observation = riskScore_cli2[4,], #指定某一患者interval ="confidence",title="Nomogram",plots=c("violin", "boxes"),clickable = T,failtime = c(365,1095,1825)) #设置随访时间1年、3年和5年
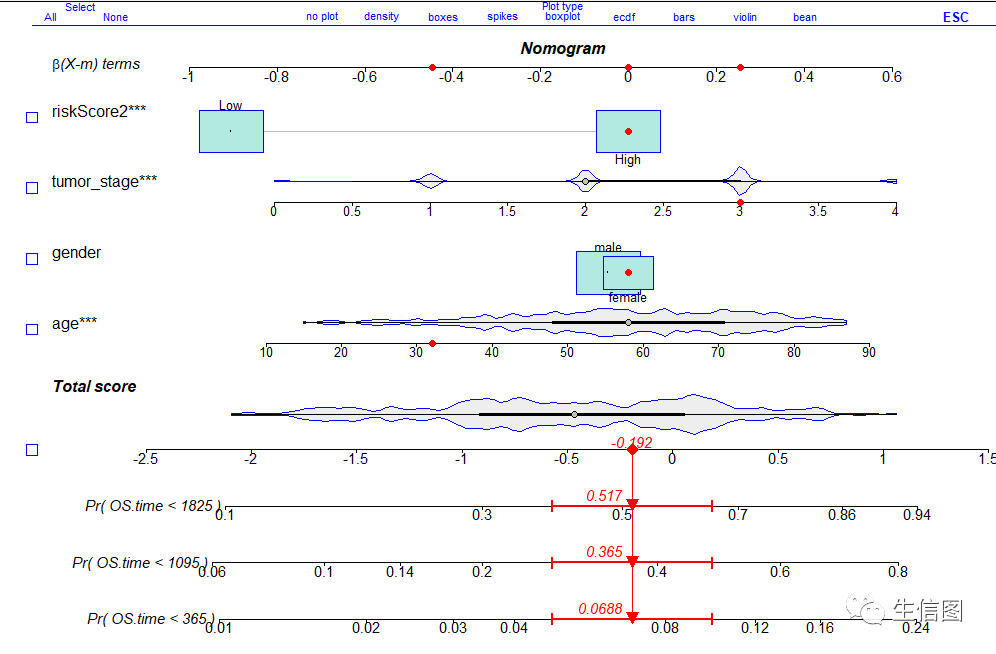
是不是立马不一样了 好看的不行,每一种特性都展示很清楚。
接下来我们去绘制校准曲线
一般来说,我们绘制1 ,3, 5年的校准曲线,小伙伴要注意m值根据样本数量选择,体现在图中多少errbar ,大概选择1/3 或者1/4即可。我们先计算一下
#1-yearcox1 <- cph(Surv(OS.time,OS) ~ age + gender + tumor_stage + riskScore2,surv=T,x=T, y=T,time.inc = 1*365,data=riskScore_cli2)cal <- calibrate(cox1, cmethod="KM", method="boot", u=1*365,m= 100, B=1000)#3-yearcox3 <- cph(Surv(OS.time,OS) ~ age + gender + tumor_stage + riskScore2,surv=T,x=T, y=T,time.inc = 1*365*3,data=riskScore_cli2)ca3 <- calibrate(cox3, cmethod="KM", method="boot", u=1*365*3,m= 100, B=1000)#5-yearcox5 <- cph(Surv(OS.time,OS) ~ age + gender + tumor_stage + riskScore2,surv=T,x=T, y=T,time.inc = 1*365*5,data=riskScore_cli2)ca5 <- calibrate(cox5, cmethod="KM", method="boot", u=1*365*5,m= 100, B=1000)
可视化结果
小伙伴可以根据选择去绘制单条还是双条这里,使用plot函数进行可视化,小伙伴如果想1,3,5年的校准曲线绘制在一张图中,使用add = T就可以,但是要注意调节xlim 和 ylim 的范围 。
#绘制单条plot(cal,lwd=2,lty=1,errbar.col="black",xlab ="Nomogram-Predicted Probability of 1-Year Survival",ylab="Actual 1-Year Survival",col="blue",sub=F)#多条plot(cal,lwd=2,lty=1,errbar.col="black",xlim = c(0.5,1) ,ylim = c(0.4,1),xlab ="Nomogram-Predicted Probability of 1,3,5Year Survival",ylab="Actual 1,3,5Year Survival",col="blue",sub=F)plot(ca3,add = T ,lwd=2,lty=1,errbar.col="black",col="red",sub=F)plot(ca5,add = T ,lwd=2,lty=1,errbar.col="black",col="green",sub=F)
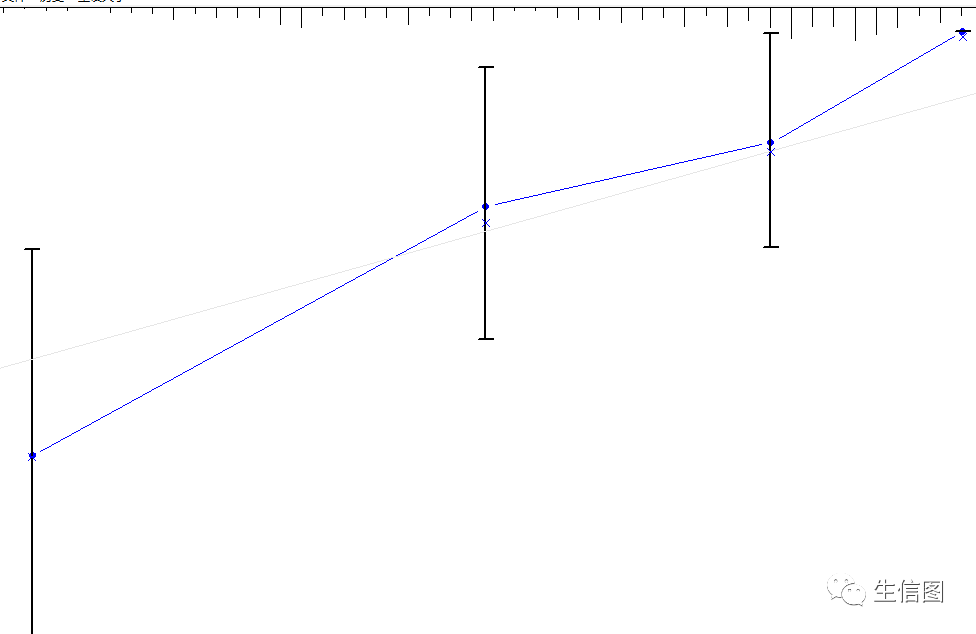
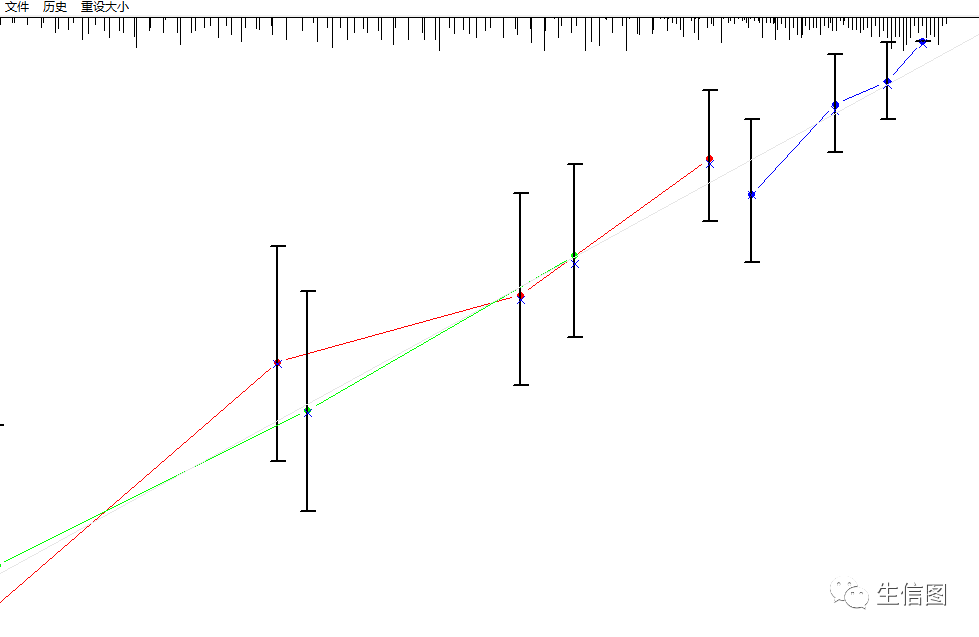
上述就是单条和多条,小伙伴自行选择
最后呢我们去绘制DCA决策曲线:
#DCA决策曲线library(caret)library(ggDCA)Cox_nomo2<-rms::cph(Surv(OS.time, OS) ~ age + gender + tumor_stage + riskScore2,data = riskScore_cli2)d_train <- dca(Cox_nomo2,times=1800)ggplot(d_train)
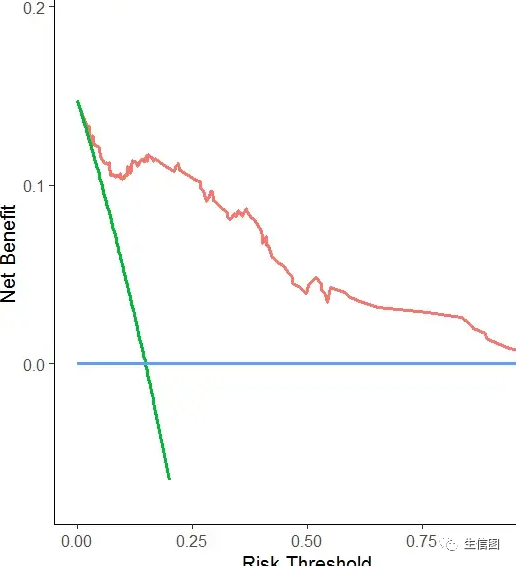

小伙伴可能会和小图一样,会出现ggDCA包下不下来,可以试试其他方式下来,有时候github进不去网络问题,多试几次,
上述就是预后模型大部分的绘制图了,小伙伴有没有学会呢,快拿自己数据去试试吧!
欢迎使用:云生信平台 ( http://www.biocloudservice.com/home.html)

|
往期推荐 |
|
|
|
|
|
|
👇点击阅读原文进入网址