小花教你使用R包corrr-对多个基因进行相关性分析和展示!!
点击蓝字 关注我们
小伙伴们在做相关性分析的时候绘制单基因的相关性分很简单,但是有的小伙伴遇见多个基因相关性分析,却不知道从何下手,小花在这里教小伙伴如何快速的方便的分析,然后高校的呈现。
今天小花主要介绍的市一个R包corrr,可以方便的做这个事情,而且小花认为这个包做的非常好。下面先介绍一下这个包:
他有一个主函数correlate可以快速的分析,是可以实现从A到B的转换。
不过小花觉得,这个包更重要的是,他还配置了两个可视化的函数,,一个是rplot可以绘制热图,一个是netword_plot绘制网络图。
小花注意到在corrr介绍中

包含的有7中函数
Rplot和network_plot()已经介绍过了,
上述中有一个fashion可以简洁化展示数据,去掉NA。
第一个框内的shave函数是把剃刀,可以去掉相关性结果上三角或者下三角并设置为NA
rearrange函数可以按照相关性系数聚类排序。
第二个框内的focus函数,类似于select函数,可以用来筛选想要查看的某行某列数据。
下面就跟着小花一一去学习吧:
首先我们需要谁知镜像以及安装R包:options("repos"=c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/"))install.packages("corrr")安装好之后我们去运行主函数看看结果:library(corrr)library(dplyr)x <- datasets::mtcars %>%correlate()
这里小花使用示例数据,小伙伴可以根据示例数据格式进行
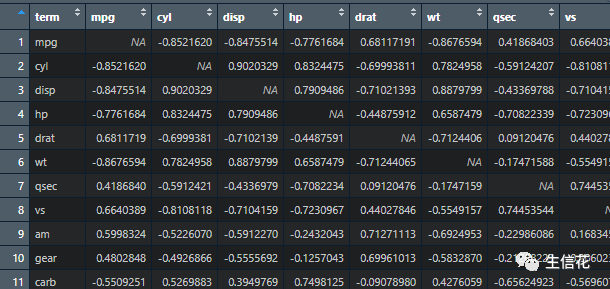
接着往下处理,focus选择要观察的数据,rearrange按照相关性系数排序,shave设置上三角的数据为NA
x <- datasets::mtcars %>%correlate() %>%focus(-cyl, -vs, mirror = TRUE) %>%rearrange() %>%shave()
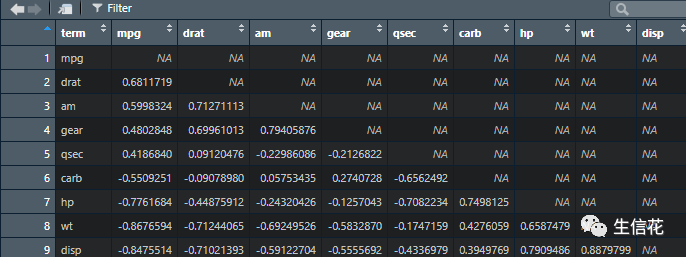
小伙伴会发现有很多的NA,我们可以使用fashion简化数据:
fashion(x)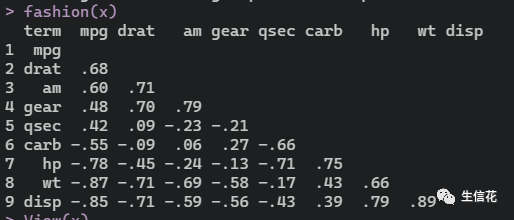
我们还可以使用rplot函数展示数据:
rplot(x)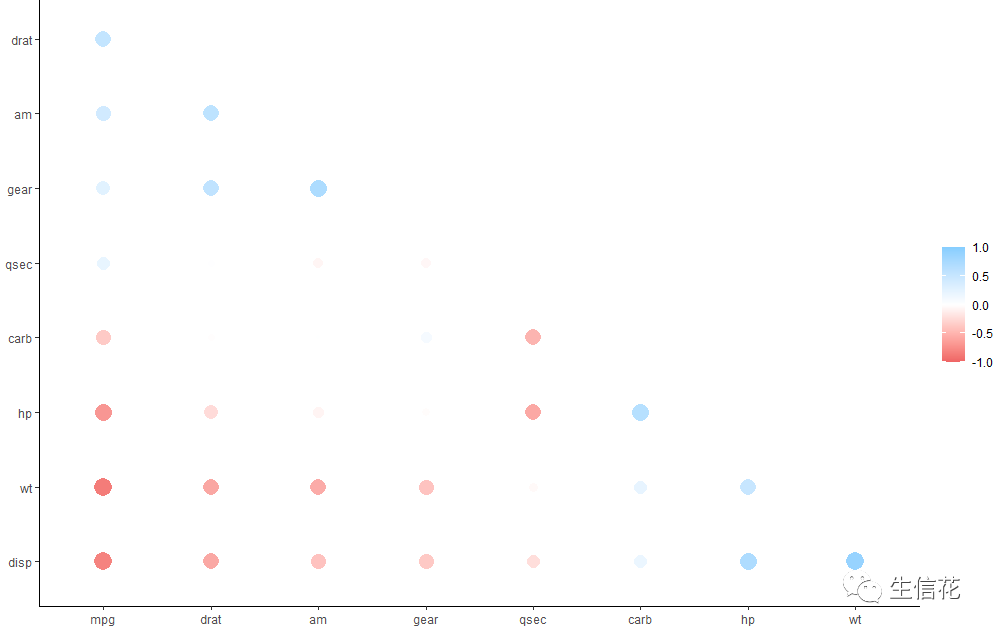
接下来就是有意思的图了,我们还可以用网络图来展示:
datasets::mtcars %>%correlate() %>%focus(-cyl, -vs, mirror = TRUE) %>%rearrange() %>%network_plot(min_cor = .2)

是不是很好看,这样形式的相关性图,放在文章中很是吸引。
最后呢我们还可以利用stretch函数实现数据的变换:
x <- datasets::mtcars %>%correlate() %>% # Create correlation data frame (cor_df)focus(-cyl, -vs, mirror = TRUE) %>% # Focus on cor_df without 'cyl' and 'vs'rearrange() %>%stretch()
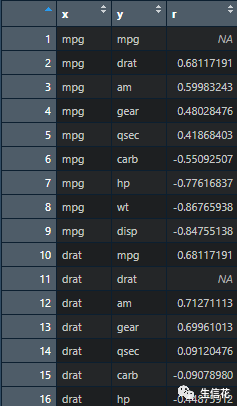
上述corrr的包已经学习完成,小伙伴是不是已经着亲自动手去绘制了呢,我们学习一个R包只是第一步,那么第二部就是想着如何用来展示自己的数据,小花这里有一个实例数据,亲自带小伙伴去实例一下:
首先导入我们的数据:
cc <- read.table('E:\生信花\data.txt',sep = "t",comment.char = "!", stringsAsFactors = F,header = T, fill=TRUE)#43931#先看看我们的数据
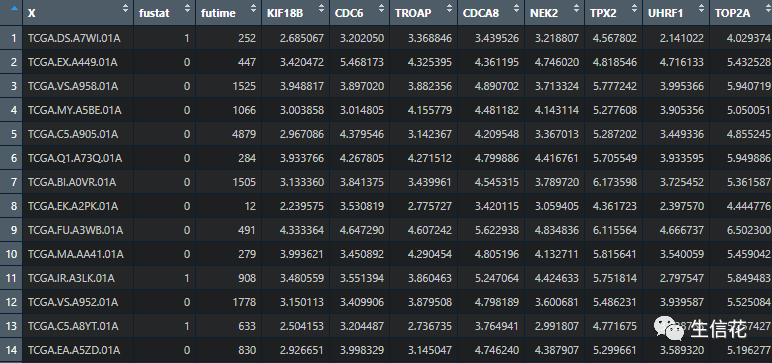
但是前两列不是我们需要的我们将它去除。
但是第一列是不在R软件中的列中我们先把他放进去
df<-ccrow.names(df) <- df[, 1]cc<- df[, -1]
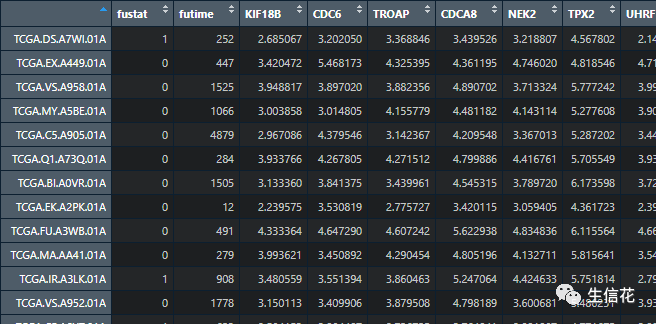
然后舍去前两列:
data <- cc[,-c(1:2)]
接下来就可以去绘制图片了,不要忘记调用R包:
library(corrr)library(dplyr)data %>%correlate() %>%rearrange() %>%network_plot()
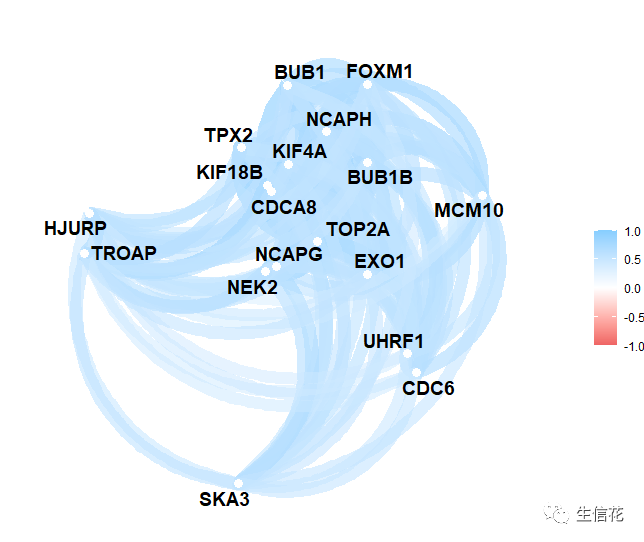
这个图看起来就有意思,这几个基因都有一定的相关性,并且都聚在一起,都是正相关,小伙伴可以使用自己的数据去绘制。
这样一来,多个基因的相关性分析就以网络图的形式展示出来了,小伙伴是不是心动了,不要往里多多理解上述函数的中意义,才能绘制出自己想要的图片。
欢迎使用:云生信 – 学生物信息学 (biocloudservice.com)
如果想用服务器可以联系微信:18502195490(快来联系我们使用吧!)

(点击阅读原文跳转)
![]() 点一下阅读原文了解更多资讯
点一下阅读原文了解更多资讯

