最新发布!细胞周期分析包tricycle!
 细胞周期(cell cycle),是指能持续分裂的真核细胞从一次有丝分裂结束后生长,再到下一次分裂结束的循环过程(准确来说只要有DNA复制,不管是不是有丝分裂它都有细胞周期。生殖细胞无细胞周期。)。细胞周期的长短反映了细胞所处状态,这是一个细胞物质积累与细胞分裂的循环过程。癌变的细胞以及特定阶段的胚胎细胞常常有异常的分裂周期。
细胞周期(cell cycle),是指能持续分裂的真核细胞从一次有丝分裂结束后生长,再到下一次分裂结束的循环过程(准确来说只要有DNA复制,不管是不是有丝分裂它都有细胞周期。生殖细胞无细胞周期。)。细胞周期的长短反映了细胞所处状态,这是一个细胞物质积累与细胞分裂的循环过程。癌变的细胞以及特定阶段的胚胎细胞常常有异常的分裂周期。
总的看来,细胞周期通常可划分为分裂间期(I期)和分裂期(M期),分裂间期是物质准备和积累阶段,分裂期则是细胞增殖的实施过程。整个周期表示为 I期→M期。其中分裂间期(I期)又常常可以划分为DNA合成前期(G1,gap 1),DNA合成期(S, synthesis)和DNA合成后期(G2, gap2)。在此期间的任务主要是完成染色质中的DNA复制和相关蛋白质的合成。将I期细分之后,整个细胞周期可以表示为:G1期→S期→G2期→M期。细胞进入G1期可能出现三种情况,其中暂不继续增殖,如骨髓干细胞和处于不利状态下的癌细胞,但在某些刺激下,这些细胞又可以继续生长分裂,因此有人把这种非增殖状态的G1期细胞称为G0期细胞。以区别处于增殖状态的G1期细胞。
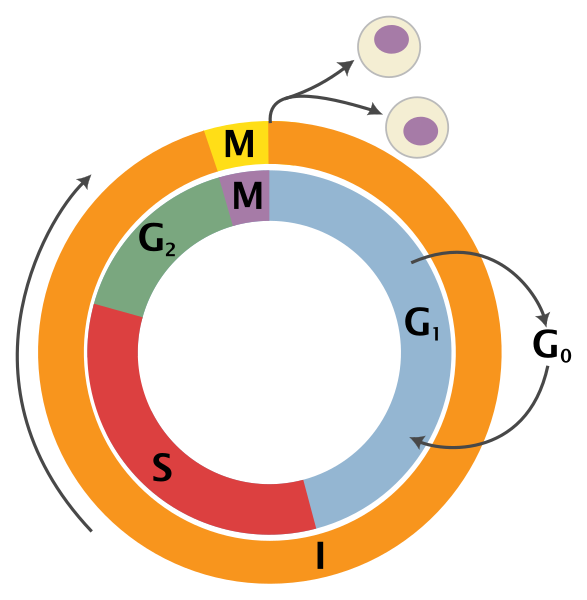
细胞周期是一个高度保守、连续的过程,控制着细胞的忠实复制和分裂。单细胞技术使得细胞周期的测量变得越来越精确,细胞周期既作为一种感兴趣的生物过程,又作为一个可能的混杂因素。尽管其有很大重要性和保守性,但还没有普遍适用的方法可以从单细胞 RNA-seq 数据中以高分辨率推断细胞周期中的位置。
目前有很多细胞周期分析软件,比如Seurat,Scanpy等。在这里,小果跟大家介绍下tricycle,一个全新发布的细胞周期分析 R/Bioconductor 包,通过利用细胞周期生物学的关键特征、周期函数主成分分析的数学特性以及迁移学习的使用来应对这一挑战。主要使用固定的参考数据集估计细胞周期嵌入,并将新数据投影到该参考嵌入中,这种方法克服了学习数据集相关嵌入的关键限制。然后,Tricycle 根据数据投影预测细胞周期中的细胞特定位置。好了,以上就是有关tricycle的前言介绍,话不多说,快来跟小果一起学习吧!
导入数据
library(tricycle)data(neurosphere_example)
将单细胞数据集投影到预先学习的细胞周期空间
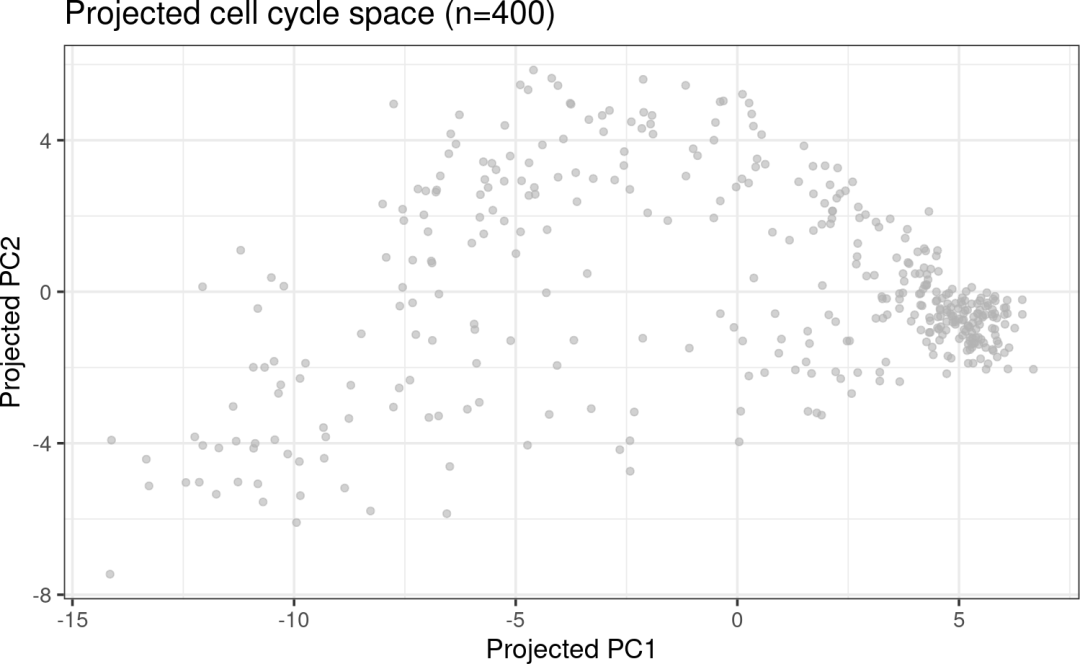
投影的细胞周期空间将存储在reduceDims中,名称为“tricycleEmbedding”(您可以设置其他嵌入名称)。
neurosphere_example<-project_cycle_space(neurosphere_example)neurosphere_examplelibrary(ggplot2)library(scattermore)library(scater)scater::plotReducedDim(neurosphere_example, dimred="tricycleEmbedding") +labs(x="Projected PC1", y="Projected PC2") +ggtitle(sprintf("Projected cell cycle space (n=%d)",ncol(neurosphere_example))) +theme_bw(base_size=14)
推断细胞周期位置
neurosphere_example<-estimate_cycle_position(neurosphere_example)names(colData(neurosphere_example))
推断细胞周期阶段
我们计算高表达的阶段特异性细胞周期标记基因的 z 分数,并将细胞分配到具有最大 z 分数的阶段。
neurosphere_example<-estimate_Schwabe_stage(neurosphere_example,gname.type='ENSEMBL',species='mouse')scater::plotReducedDim(neurosphere_example, dimred="tricycleEmbedding",colour_by="CCStage") +labs(x="Projected PC1", y="Projected PC2",title=paste0("Projected cell cycle space (n=", ncol(neurosphere_example), ")")) +theme_bw(base_size=14)

根据细胞周期位置绘制嵌入散点图
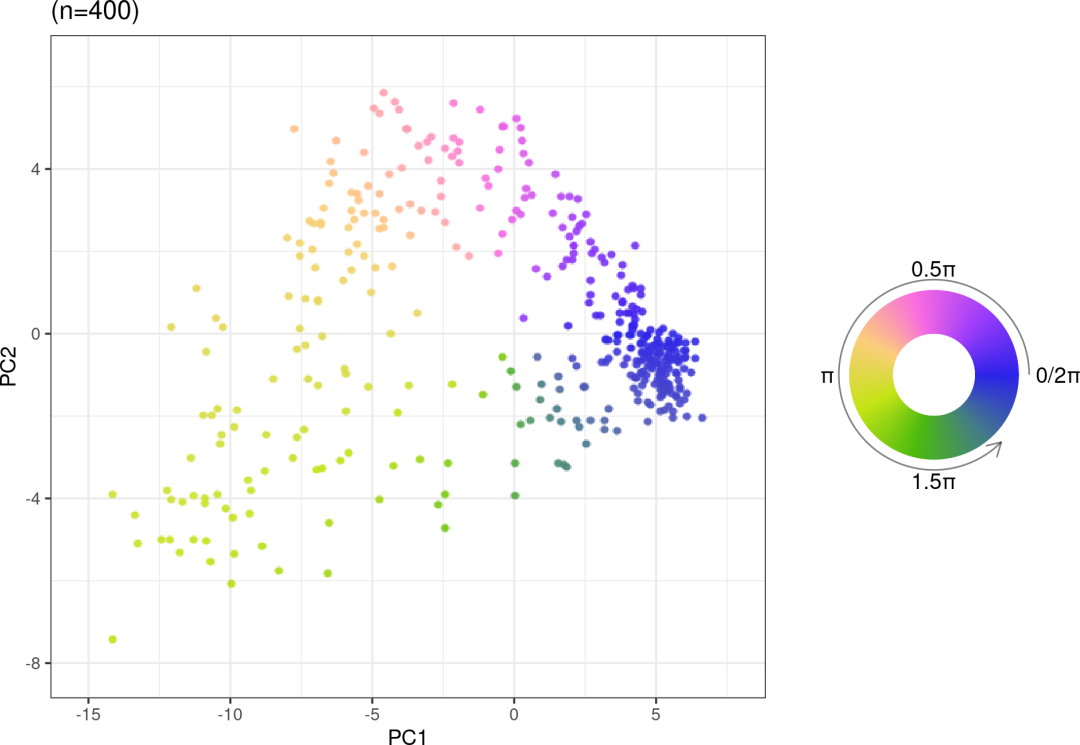
为了可视化任何嵌入上的细胞周期位置 θ,我们需要仔细选择循环调色板。因此,我们包含这样的函数来绘制具有循环变量的 SingleCellExperiment 对象的任何嵌入。还可以使用用于创建循环图例的配套辅助函数。
library(cowplot)p<-plot_emb_circle_scale(neurosphere_example, dimred=1,point.size=3.5, point.alpha=0.9) +theme_bw(base_size=14)legend<-circle_scale_legend(text.size=5, alpha=0.9)plot_grid(p, legend, ncol=2, rel_widths=c(1, 0.4))
好了,以上就是小果推荐的R包tricylce,是不是很简单?只需要简单的几行代码就可以绘制出如此精美的细胞周期图。还等什么呢?快来学习吧!关注生信果,一起来学习最近的生信工具! 

往期推荐