那些容易被人忽视的R包——基于caret的机器学习(二)
点击蓝字 关注我们
小伙伴们大家好啊,我是大海哥,今天我们要接着上次的分享,继续学习这个十分强大却低调的caret包,准备好了吗,让我们开始吧!
caret包的功能包含下面几种
数据拆分
数据预处理
特征选择
模型构建及优化
变量重要性评估
其他函数部分
并且已经详细介绍了数据拆分、数据预处理和模型构建及优化等部分,那么这一次,大海哥就接着继续介绍caret包的其他功能。
重新开始意味着要重新加载~
载入包和数据
library(caret)## 载入需要的程辑包:ggplot2## 载入需要的程辑包:latticedata(iris)iris <- na.omit(iris)
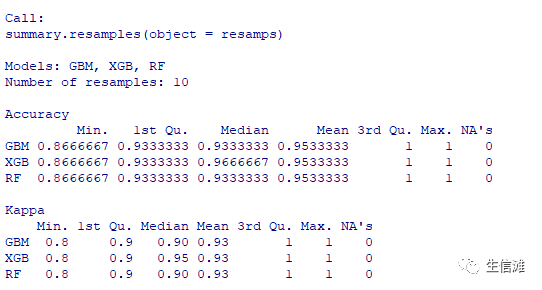
不同模型的比较
#设置重抽样方式fitControl <- trainControl(method = "cv",number = 10,allowParallel = TRUE)#建立gbm模型set.seed(1)gbmFit<- train(factor(Species) ~ .,data = iris,method = "gbm",trControl = fitControl,verbose = FALSE)#建立xgboost模型xgbFit <- train(factor(Species) ~ .,data = iris,method = "xgbTree",trControl = fitControl,verbose = FALSE,verbosity = 0)#建立随机森林模型rffit <- train(factor(Species) ~ .,data = iris,method = "rf",trControl = fitControl,verbose = FALSE)#这种模型构建方式是不是很简单!!#重采样测试进行结果对比resamps <- resamples(list(GBM = gbmFit,XGB =xgbFit,RF = rffit))
模型比较概况
summary(resamps)
可视化看看结果
bwplot(resamps)
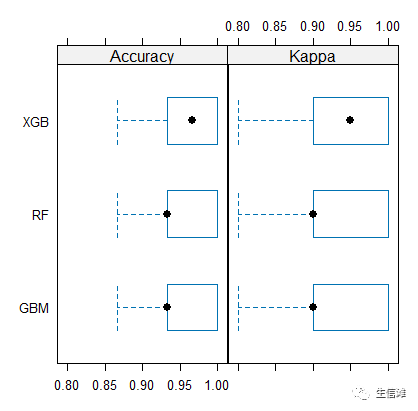
结果显示XGB最强,不愧是XGBoost!预测鸢尾花也不服输~
可以看出caret包构建模型的代码流程十分简单便捷,只需要提供一些关键参数即可,实乃萌新好助手!!
特征提取
1、过滤法
x<-iris[1:4] #变量特征y<-factor(iris$Species) #结果特征set.seed(10)filterCtrl <- sbfControl(functions = rfSBF,method = "repeatedcv", repeats = 5,saveDetails = TRUE)rfWithFilter <- sbf(y=y,x=x, data=iris, sbfControl = filterCtrl)
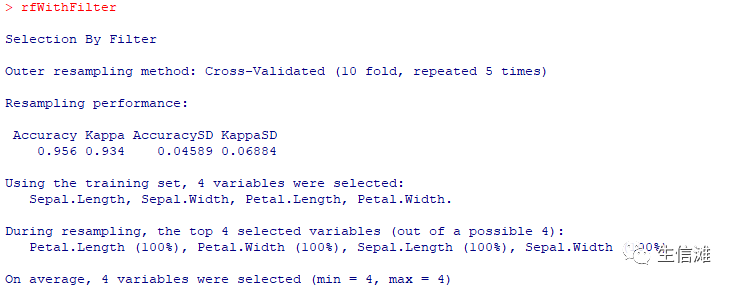
过滤法结果显示4个特征都被选出
2、递归特征消除(Recursive Feature Elimination,RFE)
subsets <- c(1:4)set.seed(10)ctrl <- rfeControl(functions = rfFuncs,method = "repeatedcv",repeats = 5,verbose = FALSE)rfProfile <- rfe(x, y,sizes = subsets,rfeControl = ctrl)rfProfile
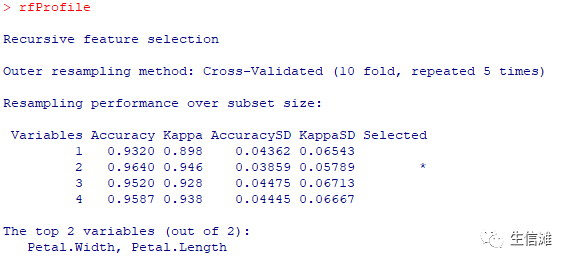
#RFE结果显示,Petal.Width和Petal.Length两个变量被选出#结果可视化plot(rfProfile, type = c("g", "o"))
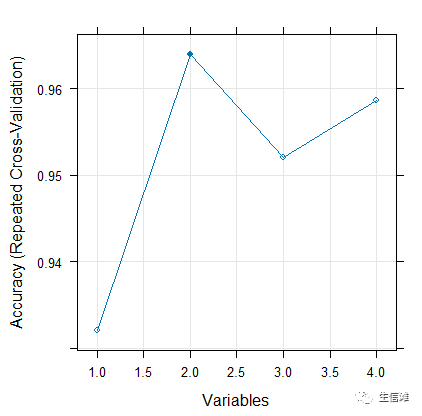
可以看到选择两个变量时,分类效果最好
3、基因算法特征选择
ga_ctrl <- gafsControl(functions = rfGA,method = "cv")set.seed(10)rf_ga <- gafs(x = x, y = factor(y),iters = 5,gafsControl = ga_ctrl)

可以看到基因算法选择的变量和RFE选择的变量一致。
4、模拟退火(Simulated Annealing)算法特征选择
sa_ctrl <- safsControl(functions = rfSA,method = "cv",improve= 50)set.seed(10)rf_sa <- safs(x = x, y = factor(y),iters = 50,safsControl = sa_ctrl)rf_sa$optVariables

#可以看出模拟退火算法选择的变量与其他几种均不相同#为了测试一下结果,我们通过lattice包的xyplot方法来看看数据分布情况library(lattice)#使用Petal.Length和Petal.Width两个特征维度来查看数据xyplot(Petal.Length ~ Petal.Width, data = iris,groups = Species,auto.key = list(corner=c(1, 0)))
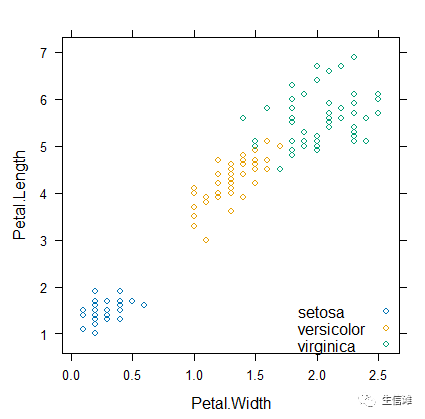
#可以看到,如果选择基因算法和RFE算法选择的变量Petal.Length和Petal.Width来查看数据,分类为setosa的数据可以被很好的区分开来,但是versicolor和virginica两类并不能被很好的区分,不过也没有完全交融在一起,表现还不错#我们再来试试模拟退火算法选择的特征结果xyplot(Sepal.Length ~ Petal.Length, data = iris,groups = Species,auto.key = list(corner=c(1, 0)))
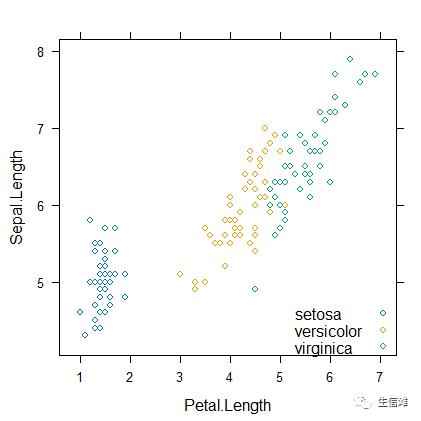
#同样的,可以看出setosa分类可以被很好的区分开来,但是versicolor和virginica两类并不能被很好的区分,并且有一些样本还被混在一起,#总结一下,基因算法和RFE算法以及模拟退火算法都实现了特征提取,减少了特征数量,并且基因算法和RFE算法提取结果表现优异。

点击“阅读原文”进入网址
