「付费」【代码分享-16】nature级别的基因表达与免疫通路相关性网络热图绘制教程

今天小果在一篇高分生信文章中看到了一个有趣的分析内容,将基因表达量与免疫相关通路关联起来,小果觉得是一个非常实用的分析,小伙伴是不是马上产生生了疑问,如何进行该分析,不急!小果为大家进行解答,首先利用GSVA包计算相关通路的富集分数,然后计算基因表达与通路富集分数的相关性,最后利用ggcor包绘制相关性网络热图,将二者形象的关联起来,通过小果的解释我想小伙伴们应该明白了吧!接下来跟着小果开始今天的实操哈!
1.准备需要的R包
#安装需要的R包install.packages("ggcor-master.tar.gz", repos = NULL, type = "source")install.packages("data.table")install.packages("GSVA")install.packages("ggplot2")#加载需要的R包library(data.table)library(GSVA)library(ggplot2)library(ggcor)
2.加载输入数据
# 加载TCGA-BLCA表达谱FPKM,行名为基因名,列名为样本名expr<-read.table("tcga_blca_fpkm.txt",sep = "t",row.names = 1,check.names = F,stringsAsFactors = F,header = T)
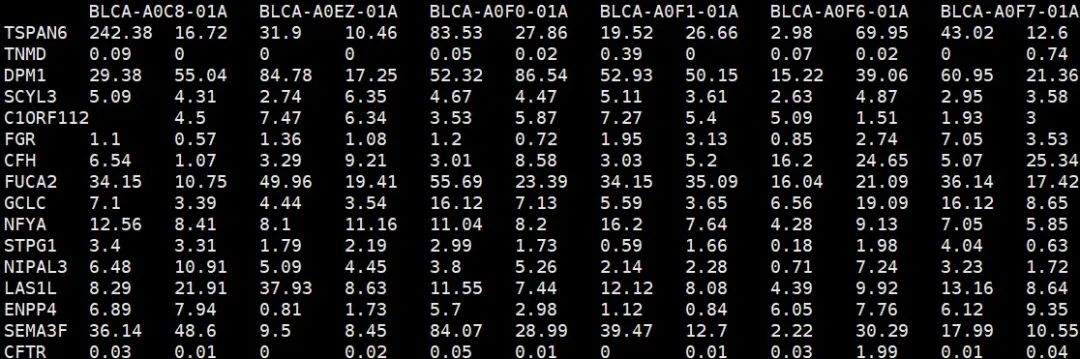
tumsam <- colnames(expr)[substr(colnames(expr),11,13) == "01A"] # 取出肿瘤样本# 取出目标基因SIGLEC15表达量siglec15 <- log2(expr["SIGLEC15",tumsam] + 1)#保存目标基因表达矩阵文件write.csv(siglec15, "easy_input_gene.csv")
结果文件
5.1 ggcor_plot_in_bottom_left.pdf
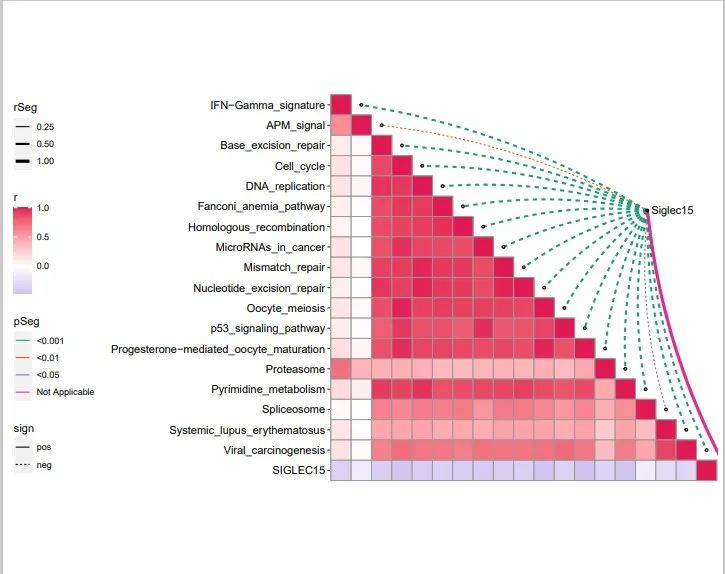
5.2 ggcor_plot_in_top_right.pdf
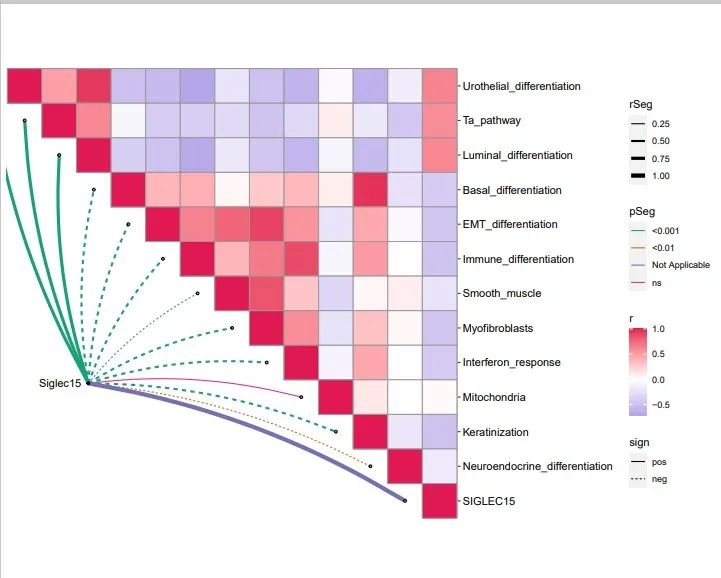
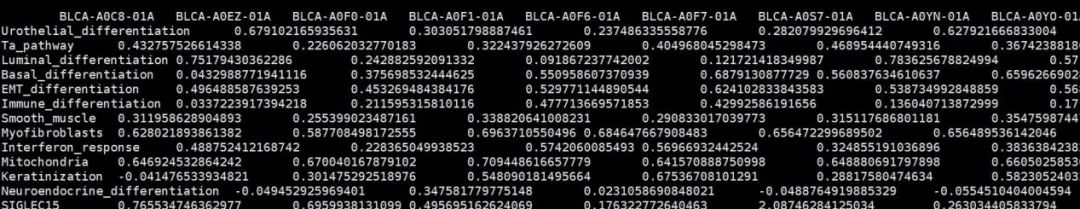
5.3 immPath.txt
该结果文件为计算的免疫通路富集分数结果文件,行名为通路名,列名为样本名。
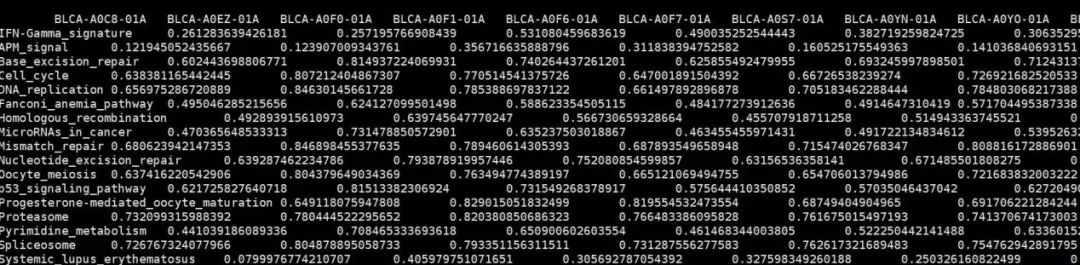
5.4 blcaPath.txt
该结果文件为计算的免疫通路富集分数结果文件,行名为通路名,列名为样本名
往期推荐
