实用小技巧——给火山图添加基因标签
点击蓝字 关注我们
小伙伴们大家好,我是大海哥,今天要给大家分享的不是什么很厉害的技术,也不是超前的理论科普,而是一个每个生信人都能用得上的小技巧,你说你用不上?难道你不做差异分析?只要你需要做差异分析,那么你肯定会绘制火山图,于是今天大海哥分享的技巧你就可以用上了。是的没错~大海哥今天分享的就是火山图绘制的小技巧,给基因散点加上基因名标签。
#先准备好R包if(!require(ggplot2))install.packages("ggplot2")if(!require(ggrepel))install.packages("ggrepel")if(!require(dplyr))install.packages("dplyr")library(ggplot2)library(ggrepel)library(dplyr)
#本次我们有3种方可以实现散点添加标签#方法一:利用空字符“”#这种方法是参照帮助文档里的一段代码:#下面代码来源于geom_text_repel的帮助文档p <- ggplot(mtcars,aes(wt, mpg, label = rownames(mtcars), colour = factor(cyl))) + geom_point()# Hide some of the labels, but repel from all data pointsmtcars$label <- rownames(mtcars)mtcars$label[1:15] <- ""p + geom_text_repel(data = mtcars, aes(wt, mpg, label = label))
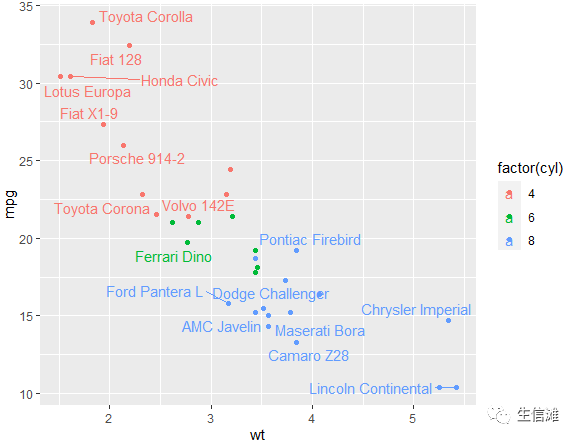
#这是结果图,可以看到,一些点有标签名,一些没有#于是大海哥学以致用#先画出火山图p <- ggplot(data = data, aes(x = log2(data$FC),y = -log10(data$p.value),colour=change,label = data$symbol)) +geom_point(alpha=0.4, size=3.5) + scale_color_manual(values=c("blue", "grey","red"))+xlim(c(-4.5, 4.5)) +geom_vline(xintercept=c(-1,1),lty=4,col="black",lwd=0.8) +geom_hline(yintercept = -log10(0.000001),lty=4,col="black",lwd=0.8) +labs(x="log2(fold change)",y="-log10 (p-value)",title="Differential metabolites") +theme_bw()+ theme(plot.title = element_text(hjust = 0.5), legend.position="right",legend.title = element_blank())p
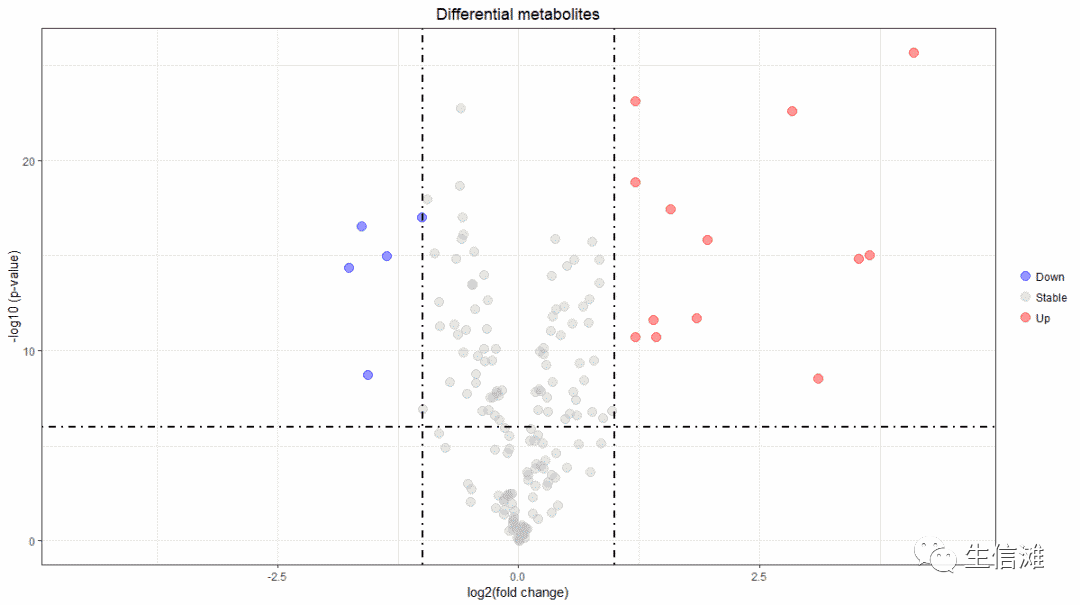
#然后我们可以筛选出部分基因#想在图上做修改,一半是调参数,一半是调数据。我们现在要做的就是调数据:要标记的,label=基因,无需标记的,label=“”。data$label=ifelse(data$p.value < 0.000001 & abs(log2(data$FC)) >= 1,data$symbol,"")#这就好了,然后把文字图层叠加上去#data为差异分析结果,大家自行修改哦!包含FC列和p.value以及基因名列就可以啦!p+geom_text_repel(data = data, aes(x = log2(data$FC), y = -log10(data$p.value), label = label),size = 3,box.padding = unit(0.5, "lines"),point.padding = unit(0.8, "lines"),segment.color = "black",show.legend = FALSE)
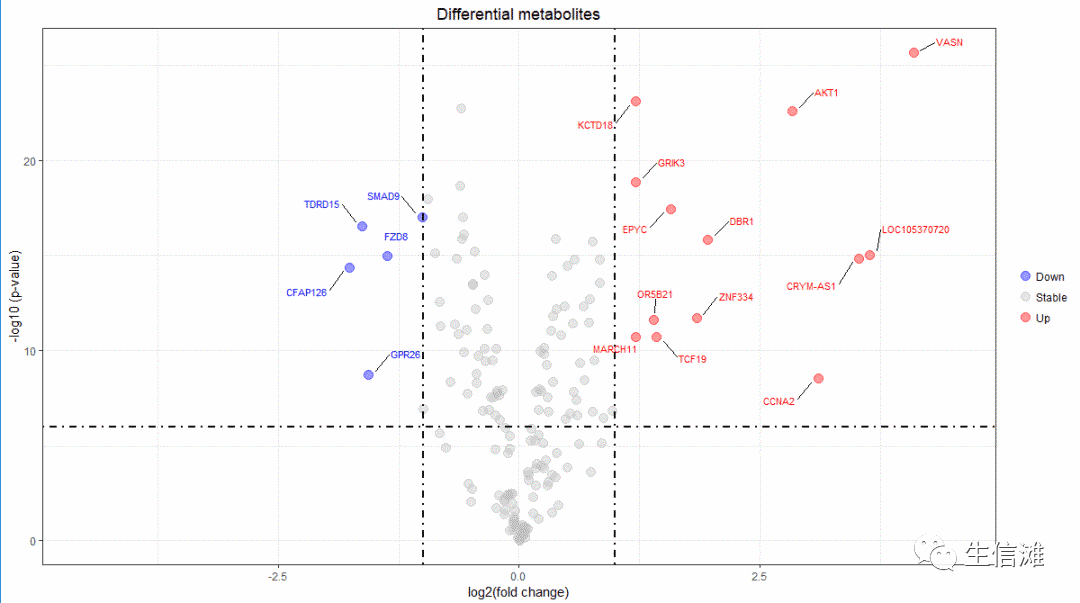
##完美实现!但是这种办法只适用于数据量较小的时候,数据量太大了对于设备的性能要求就会要求太高了!#方法二:图层叠加#这个方法是来自R数据科学笔记best_in_class <- mpg %>% group_by(class) %>% filter(row_number(desc(hwy)) == 1)ggplot(mpg, aes(displ, hwy)) + geom_point(aes(color = class)) + geom_point(size = 3, shape = 1, data = best_in_class) + ggrepel::geom_label_repel( aes(label = model), data = best_in_class )
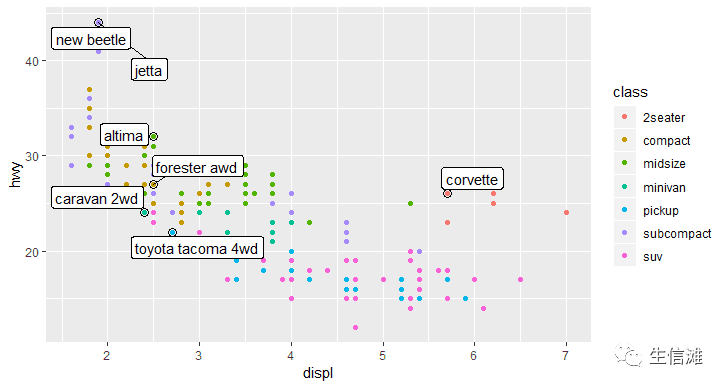
#这个方法适用于较大数据#先同样画出火山图p <- ggplot(data = test,aes(x = logFC,y = P.value)) + #test是差异分析结果,大家自行更换哦,包含logFC列和P.value以及基因名列就可以啦!geom_point(alpha=0.4, size=3.5,aes(color=change)) +scale_color_manual(values=c("blue", "grey","red"))+geom_vline(xintercept=c(-1,1),lty=4,col="black",lwd=0.8) +geom_hline(yintercept = -log10(0.01),lty=4,col="black",lwd=0.8) +theme_bw()p
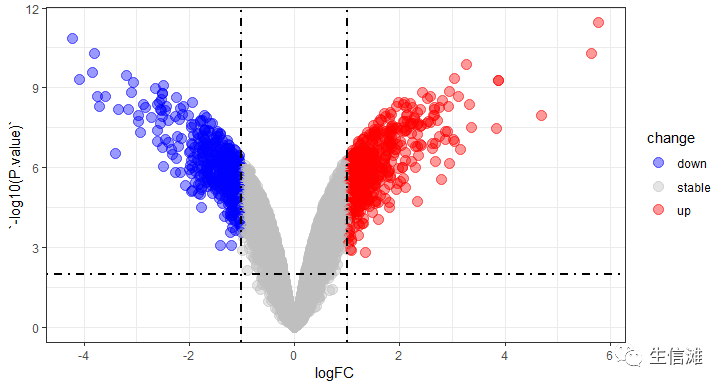
#然后选择想标记的基因,新建一个标签图层for_label <- test %>% filter(abs(logFC) >4& P.value> -log10(0.000001))#再把标签图层叠加上去p + geom_point(size = 3, shape = 1, data = for_label) +ggrepel::geom_label_repel(aes(label = symbol),data = for_label, color="black" )
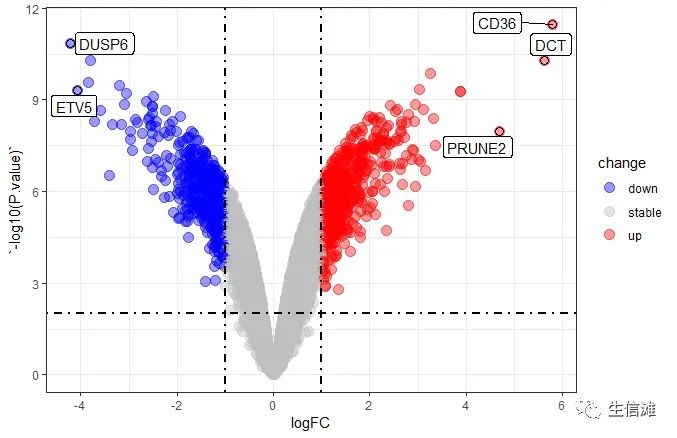
这个看着也是相当不错!
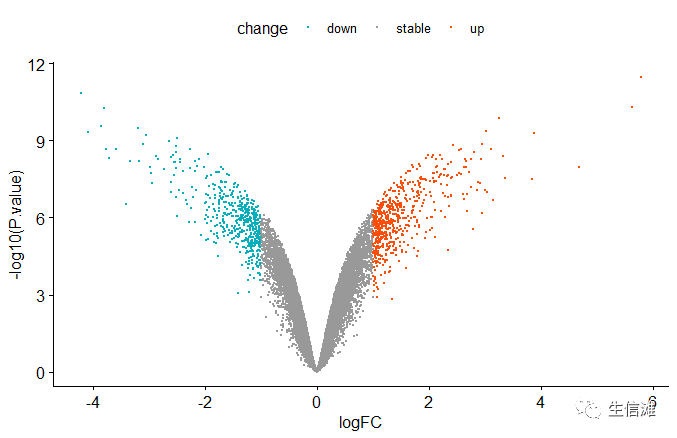
#还有方法三,也是最便捷的一种方式,因为ggpubr的函数有现成的参数library(ggpubr) colnames(test)[4] <- "v"ggscatter(test,x = "logFC",y ="P.value", ylab="-log10(P.value)",size=0.5,color = "change", palette = c("#00AFBB", "#999999", "#FC4E07"))#然后可以手动选择我们想要标记的基因,这里大海哥列举了这几个"CD36", "DUSP6", "DCT", "SPRY2", "MOXD1", "ETV4",大家自行修改。ggscatter(test, x = "logFC", y = "v", ylab="-log10(P.value)", color = "change", size = 0.5, label = "symbol", repel = T, palette = c("#00AFBB", "#999999", "#FC4E07") ,#就是这个label.select标签,直接加上去就行。#label.select = dat$symbol[1:30] , label.select = c("CD36", "DUSP6", "DCT", "SPRY2", "MOXD1", "ETV4" ) )#先看看原始的图
#看看加上后的图,我们这里重新选择logFC前30个基因ggscatter(test,x = "logFC",y = "v",ylab="-log10(P.value)",color = "change",size = 0.5,label = "symbol",repel = T,palette = c("#00AFBB", "#999999", "#FC4E07") ,label.select = test$symbol[1:30])
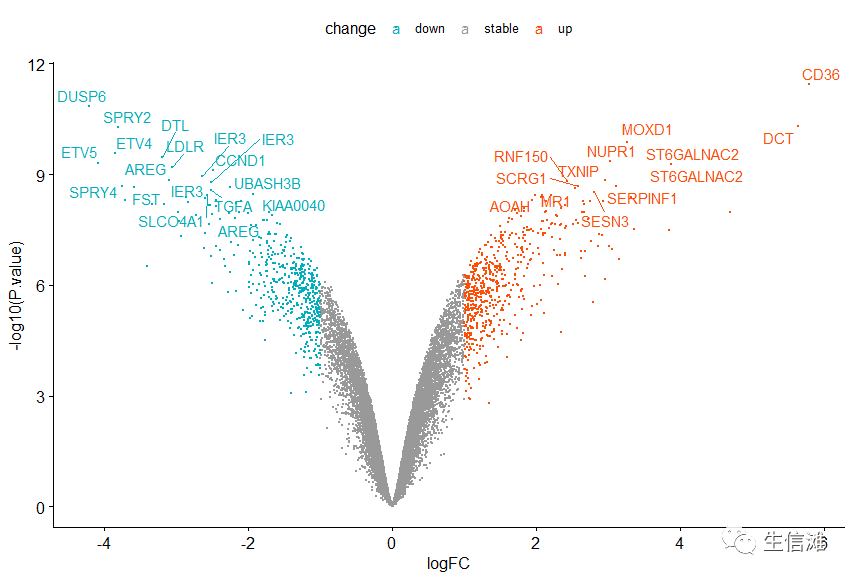
可以看到结果十分的方便美观!

点击“阅读原文”进入网址
