差异分析——从原理到火山图的实现
{ 点击蓝字,关注我们 }
什么是差异分析
差异分析,比较两组的差异性做相关分析。在比较的两组的数据过程中,涉及到“显著”的定义。怎样的差异才算是显著的呢,这就需要我们了解一些统计学的知识了。通常我们是对两组数据的差异倍数进行统计学上的检验,得到P value,认为达到一定的阈值(p<0.05)则为显著差异。我们经常做差异检验的时候看一些统计学的名词,例如:
FC(Fold Change),即为差异倍数。简单来说就是基因在一组样品中的表达值的均值除以其在另一组样品中的表达值的均值。如果规定FC>2为上调,那么FC<1/2为下调。在很多实际应用中,常常有人把FC值做log2转换,log2fc的值有正负值之分,很容易看出2个group之间的上下调关系。
Pvalue统计检验获得的是否统计差异显著的一个衡量值,约定成俗的P-value<0.05为统计检验显著的常规标准。
FDR (false discovery rate),即校正后的P值,中文一般译作错误发现率。
差异分析的方法
常见的差异性分析方法有,T检验、方差分析、卡方检验。根据你的数据类型选择合适的差异分析的方法,如果X和Y数据都是定类的数据类型就需要适应卡方检验,如果X是定类,Y是定量的数据类型,那可以使用T检验或者是方差检验。T检验适合于X组别是2的数据,而方差检验适用于2组或两组以上的。
差异分析的作图
一般差异分析可以用多种类型的图来展示,有箱线图、小提琴图、偏差图和火山图等等。今天我们用R包来探索火山图来展示差异分析的结果。
构建数据集
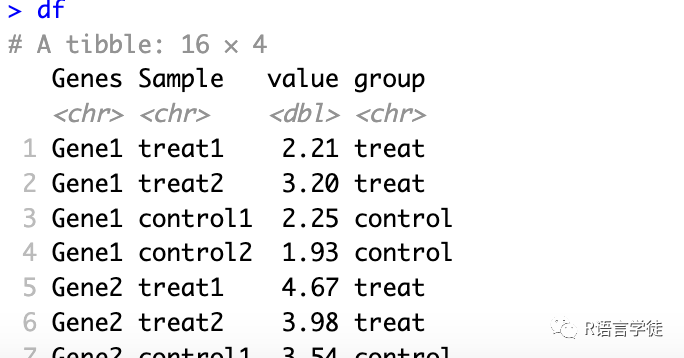
#set.seed(1234)mydata <- data.frame(Genes = c("Gene1","Gene2","Gene3","Gene4"),treat1 = rnorm(4,2)+1,treat2 = rnorm(4,2)+1,control1 = rnorm(4,1.5)+1,control1 = rnorm(4,1.5)+1)mydata #创建样本表达量信息

group <- data.frame(Sample = c("treat1", "treat2","control1","control2"),group = c(rep("treat",2),rep("control",2)))group#创建的样本分组信息

数据分析
# 表达量+分组 合并,整理install.packages("tidyverse")install.packages("rstatix")library(tidyverse)library(rstatix)#数据的整合df <- mydata %>% as_tibble() %>% pivot_longer(-1,names_to = "Sample",values_to = "value")#添加分组数据df <- left_join(df,group,by=c("Sample" = "Sample"))
绘制火山图
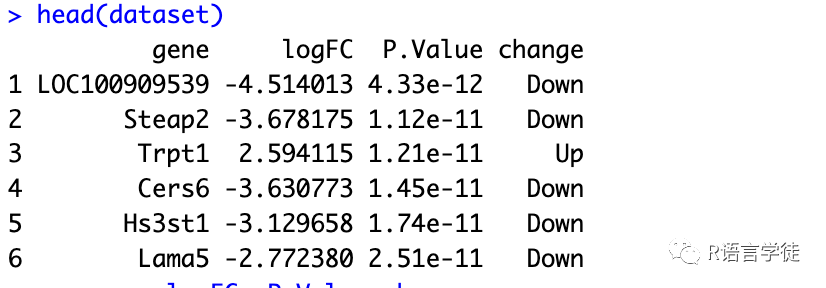
library(ggplot2)#这里把测试的样本放在附件中啦,供参考dataset <- read.table(file = "./dataset_volcano.txt", header = TRUE, sep = "")# 设置p_value和logFC的阈值cut_off_pvalue = 0.0000001 #统计显著性cut_off_logFC = 1.5 #差异倍数值# 根据阈值参数,上调基因设置为‘up’,下调基因设置为‘Down’,无差异设置为‘Stable’,并保存到change列中dataset$change = ifelse(dataset$P.Value < cut_off_pvalue & abs(dataset$logFC) >= cut_off_logFC,ifelse(dataset$logFC> cut_off_logFC ,'Up','Down'),'Stable')head(dataset)
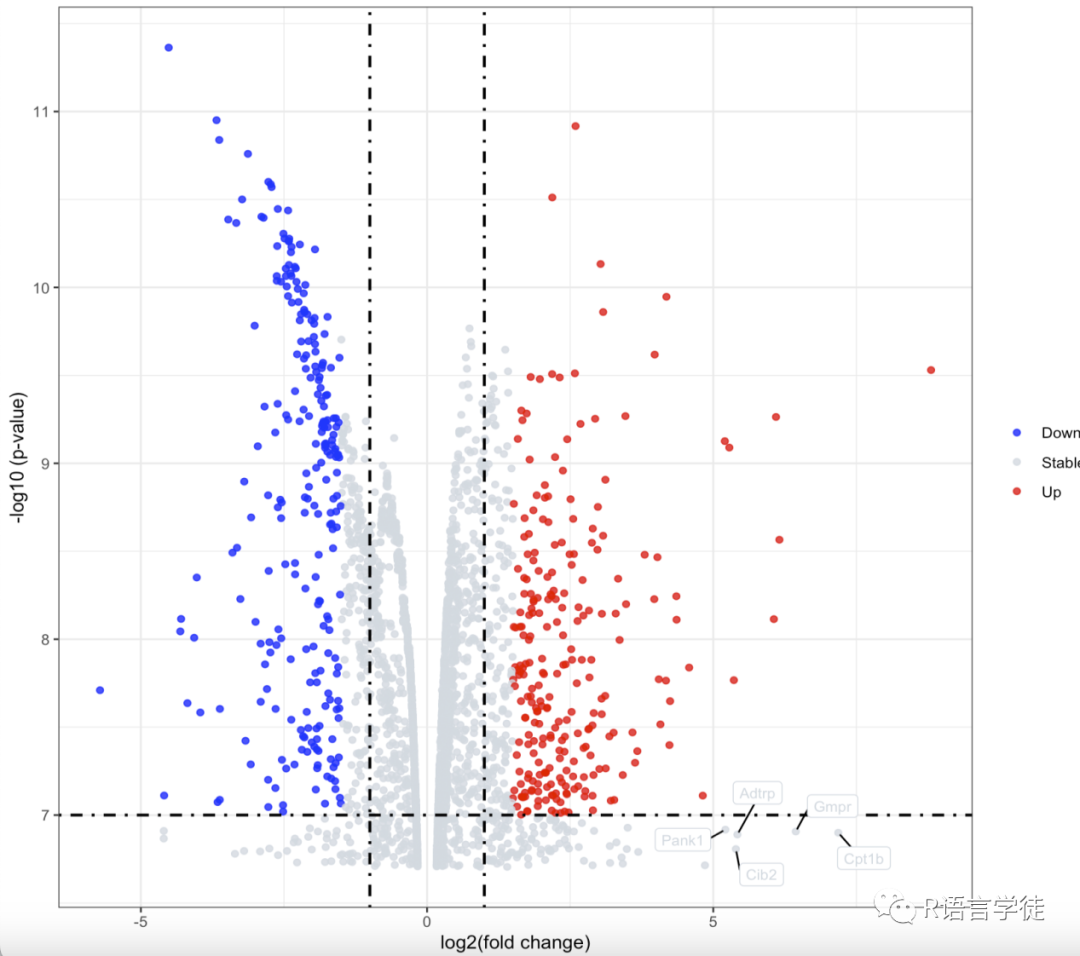
p <- ggplot(# 数据、映射、颜色dataset, aes(x = logFC, y = -log10(P.Value), colour=change)) +geom_point(alpha=0.8, size=1.5) +scale_color_manual(values=c("blue", "#d2dae2","red"))+# 辅助线geom_vline(xintercept=c(-1,1),lty=4,col="black",lwd=0.8) +geom_hline(yintercept = -log10(cut_off_pvalue),lty=4,col="black",lwd=0.8) +# 坐标轴labs(x="log2(fold change)",y="-log10 (p-value)")+theme_bw()+# 图例theme(plot.title = element_text(hjust = 0.5),legend.position="right",legend.title = element_blank())p#添加标签# 将需要标记的基因放置在label列(logFC >= 5)library(ggrepel)dataset$label <- ifelse(dataset$P.Value > cut_off_pvalue & abs(dataset$logFC) >= 5,as.character(dataset$gene), "")p + geom_label_repel(data = dataset, aes(x = dataset$logFC,y = -log10(dataset$P.Value),label = label),size = 3, box.padding = unit(0.5, "lines"),point.padding = unit(0.1, "lines"),segment.color = "black",show.legend = FALSE)
理解火山图
火山图是散点图的一种,它将统计测试中的统计显著性量度(如p value)和变化幅度相结合,从而能够帮助快速直观地识别那些变化幅度较大且具有统计学意义的数据点(基因等)。常应用于转录组研究,也能应用于基因组,蛋白质组,代谢组等统计数据。关注火山图,先理解每个点是什么(点代表基因、样品、通路或其它的,这个认识可以来自于常识,更准确的是看作者的描述),然后看横轴代表什么、纵轴代表什么,再看图例中展示的其他信息,如颜色、大小和形状分别代表什么。这些都理顺了,图理解就不难了。
例如我们做出的图中,
-
每个点代表一个检测到的基因。
-
横轴和纵轴用于固定点在空间的位置。
-
横轴是Log2(fold change),点越偏离中心,表示差异倍数越大。
-
纵轴是-Log 10 (adjusted P-value),点越靠图的顶部表示差异越显著。
-
点的大小和颜色也可以表示更多的属性,颜色标记其对应的基因是上调, 下调还是无差异。
这样我们的差异分析图就做好了,如果有问题可以与和小编进行讨论哦~
E
N
D
