教你如何在R中使用memisc语言包快速处理和管理各类型的数据
点击蓝字 关注我们
R语言是一种功能强大的统计分析和数据可视化工具,拥有许多强大的包,可以扩展其功能。其中,memisc(意为”Memory of the Social Sciences”)是一个非常有用的R语言包,旨在处理社会科学数据,并提供数据管理、数据转换和报告生成的功能。
memisc包提供了一组方便的函数,用于处理和管理各种类型的数据。它支持导入和导出多种数据格式,如CSV、SPSS和Stata等。这使得研究人员可以轻松地将各种数据源整合到一个统一的环境中进行分析。memisc还提供了一些用于数据清理和转换的函数,包括对缺失值的处理、重编码变量和创建衍生变量等。这些功能使得数据预处理变得更加高效和方便。
除了数据管理和转换功能,memisc还提供了用于报告生成的强大工具。使用该包,研究人员可以轻松地创建漂亮和可定制化的报告。memisc支持创建基于LaTeX的PDF报告,并提供了各种选项来控制报告的样式和格式。此外,该包还支持HTML和RTF格式的报告生成,使得研究人员可以在不同的平台上方便地共享和呈现结果。
memisc包的另一个重要特性是它对元数据的支持。元数据是描述数据集和变量的信息,包括变量名称、标签和缺失值定义等。memisc提供了一组函数,用于创建和管理元数据。通过使用元数据,研究人员可以更好地理解和解释数据,提高数据分析的可靠性和可重复性。
此外,memisc还支持用于描述和分析调查数据的复杂样本调查设计。它提供了一组函数,用于处理复杂样本调查数据的权重和聚集信息。这些函数使得在进行统计分析时能够考虑样本调查设计的特殊性,从而得到更准确和可靠的结果。
要使用memisc包,可以在R中使用以下命令进行安装和加载:> install.packages("memisc") #安装memisc语言包> library(memisc) #加载语言包
示例:
在R中,可以使用memisc包处理和管理生物信息数据。假设我们有一个包含基因表达量的数据集,我们可以使用memisc包的功能来处理和分析这些数据。首先,我们需要导入memisc包和我们的数据集。假设我们的数据集名为"gene_expression.csv",其中包含了基因表达量的信息。#安装语言包> install.packages("memisc")> library(memisc)# 导入数据集> gene_expression <- read.csv("gene_expression.csv", header = TRUE, sep = ","
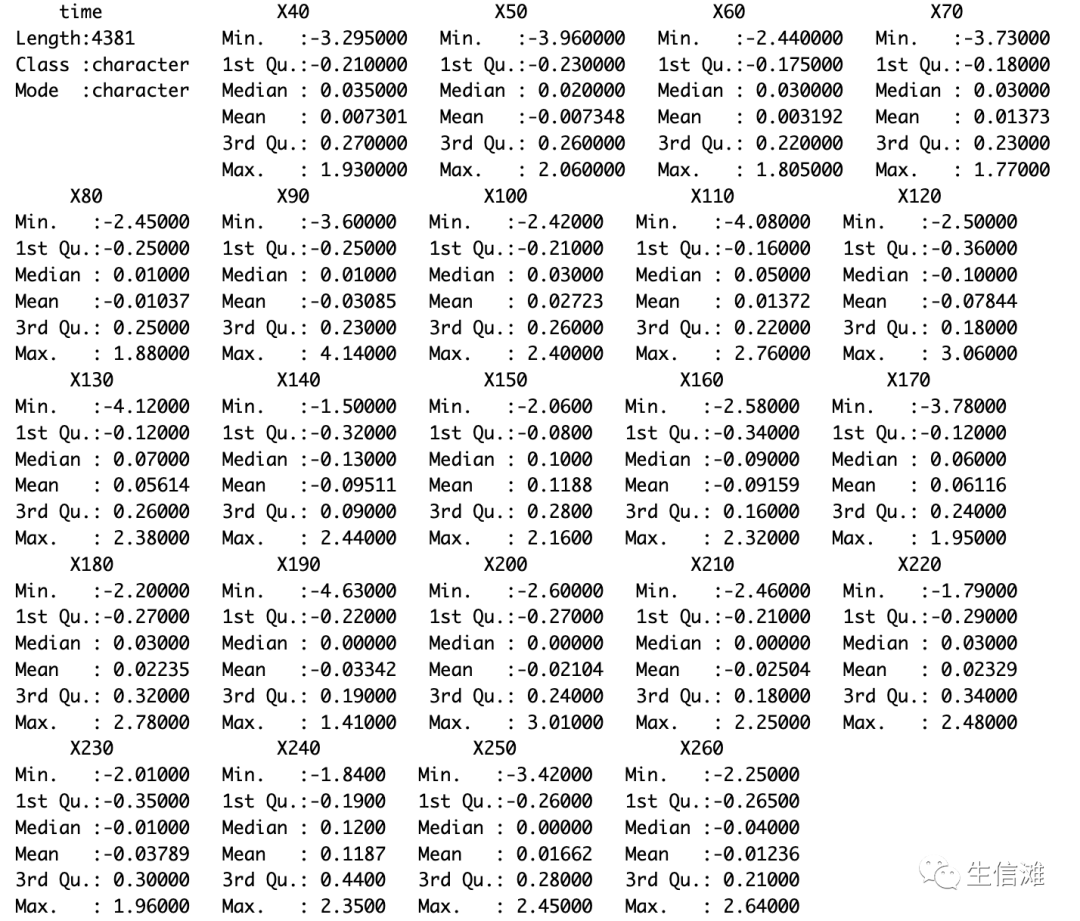
一旦我们导入了数据集,我们可以使用memisc包的各种函数来进行数据管理和分析。例如,我们可以查看数据集的结构和摘要统计信息# 查看数据集结构> dataStructure(gene_expression)# 查看摘要统计信息> summary(gene_expression)
接下来,我们可以使用memisc包的函数来进行数据转换和清理。假设我们想要计算每个基因的平均表达量,我们可以使用memisc包提供的函数来实现。
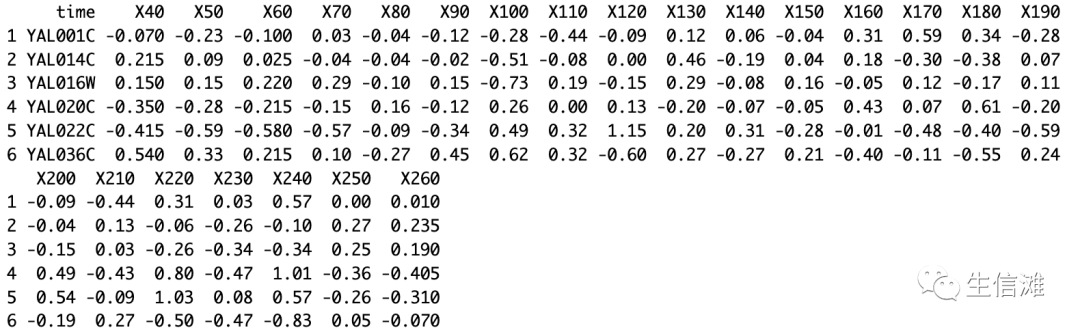
# 计算平均表达量> gene_expression$mean_expression <- rowMeans(gene_expression)# 查看新添加的变量> head(gene_expression)
除了数据转换,memisc包还支持创建和管理元数据。我们可以使用memisc包的函数为我们的数据集添加变量标签和缺失值定义等元数据信息。
# 创建变量标签> variable_labels(gene_expression) <- c(gene1 = "Gene 1 expression",gene2 = "Gene 2 expression",gene3 = "Gene 3 expression")# 创建缺失值定义> missing_values(gene_expression) <- list(gene1 = c(999, 888),gene2 = c(777),gene3 = c(666))# 查看变量标签和缺失值定义> variable_labels(gene_expression)> missing_values(gene_expression)
最后,我们可以使用memisc包的报告生成功能来创建漂亮和可定制化的报告。我们可以选择导出报告为PDF、HTML或RTF格式,具体取决于我们的需求。
# 创建PDF报告> report <- prepareCodebook(gene_expression)> writeCodebook(report, "gene_expression_codebook.pdf", format = "pdf")# 创建HTML报告> report <- prepareCodebook(gene_expression)> writeCodebook(report, "gene_expression_codebook.html", format = "html")# 创建RTF报告> report <- prepareCodebook(gene_expression)> writeCodebook(report, "gene_expression_codebook.rtf", format = "rtf")
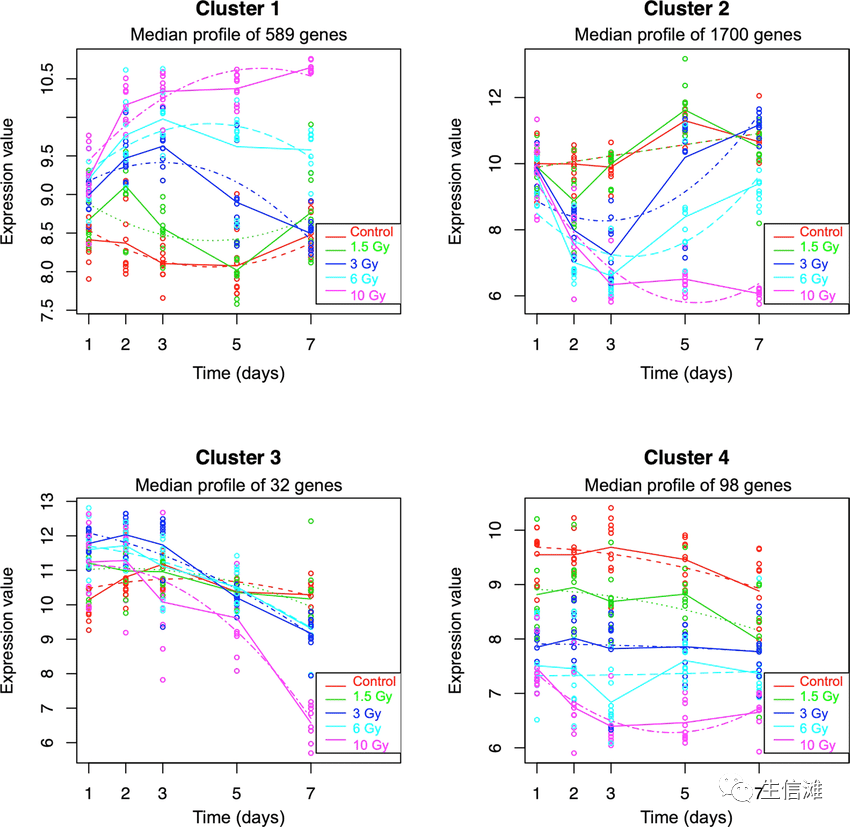
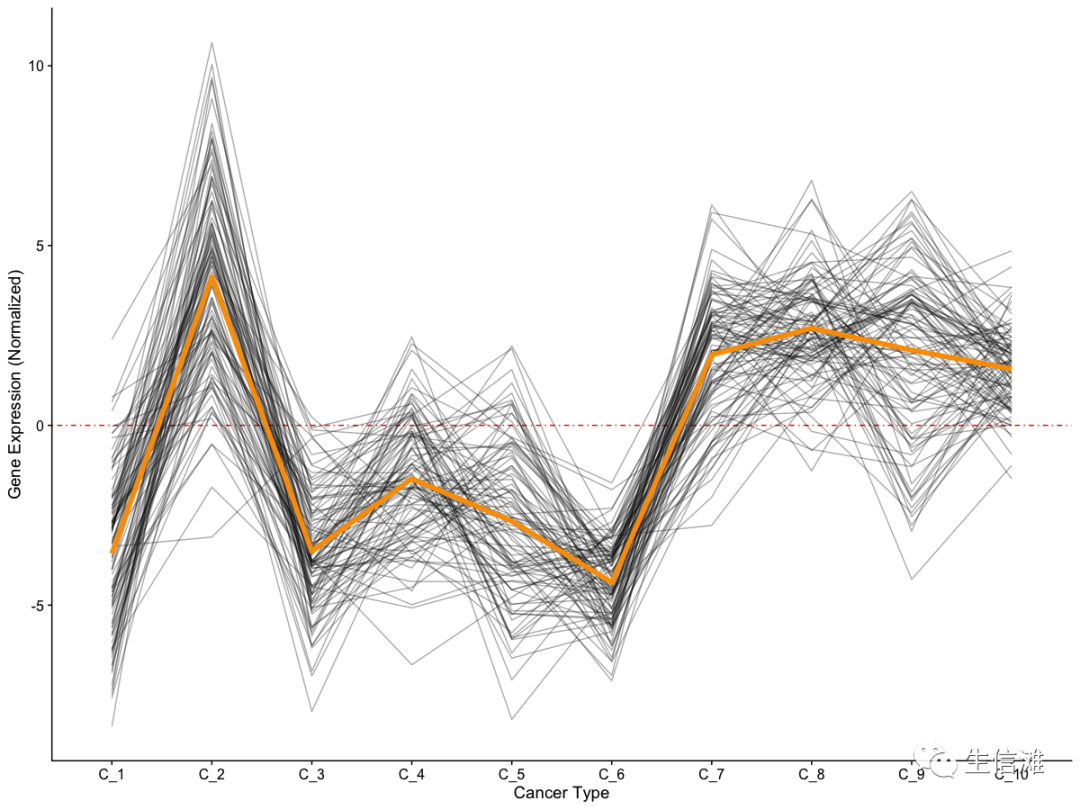
相
关
推
荐
1.https://stackoverflow.com/questions/63269006/gene-expression-profile-plot-in-r
2.https://www.kaggle.com/datasets/crawford/gene-expression?resource=download&select=actual.csv
3.https://www.researchgate.net/figure/Gene-expression-profile-clusters-The-maSigPro-R-package-was-used-to-identify-genes-with_fig3_337733551

点击“阅读原文”进入网址
