R包“gwasrapidd”:GWAS Catalog数据的获取
{ 点击蓝字,关注我们 }
孟德尔随机化分析常用的数据库,主要为OpenGWAS、GWAS Catalog和Phenoscanner三个。今天,小师妹给大家介绍一下获取GWAS Catalog数据库数据的“gwasrapidd”包。
gwasrapidd包于近期加入CRAN,意味着咱可以使用install。Packages安装gwasrapidd包了。
install.packages("gwasrapidd") #安装gwasrapidd包library(gwasrapidd) #引用gwasrapidd包
首先是get_studies()函数,get_studies()函数有以下参数可选,咱们这里就以自身免疫性疾病为例给大家演示一下。

通过efo_trait参数指定查询有关自身免疫疾病的文章my_studies <- get_studies(efo_trait = 'autoimmune disease')n(my_studies)#查看自身免疫疾病发文量my_studies@studies$study_id#获取文章的idmy_studies@publications$title#查询文章题目open_in_pubmed(my_studies@publications$pubmed_id)#在浏览器中快速打开pubmed界面

接下来是get_associations()函数,
my_associations<-get_associations(study_id=my_studies@studies$study_id)slotNames(my_associations)
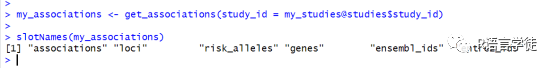
get_associations()函数会返回6个slot,分别为关联值大小,位点信息,风险等位基因信息,基因信息,基因的ENSEMBL编码和基因的ENTREZ编码信息,咱先把P值小于e-6的位点提取出来
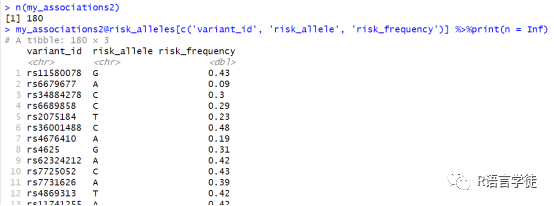
dplyr::filter(my_associations@associations, pvalue <1e-6) %>%tidyr::drop_na(pvalue) %>% dplyr::pull(association_id) ->association_idsmy_associations2 <- my_associations[association_ids]n(my_associations2)#查看显著SNP的个数my_associations2@risk_alleles[c('variant_id', 'risk_allele', 'risk_frequency')] %>%print(n = Inf)#显示显著信号位点的RS ID, risk allele,频率
接下来咱使用get_studies()获取含有特定位点的文献,咱就挑rs11580078来练练手
s2 <- get_studies(variant_id = 'rs11580078')s2@publications$title

好啦,gwasrapidd包教程就到这里吧,相信通过上面的教程大家可以发现,gwasrapidd包可以帮助我们快速查询GWAS Catalog数据库中想要的数据信息,虽然“gwasrapidd”包在Github上已存在好几年了,但直到最近才加入到CRAN里,目前来看,该包还是比较稳定和可靠的,有需要的小伙伴赶快学起来吧!
★

师妹微信
扫码添加