8+SCI生信文章常用机器学习算法LASSO,SVM-RFE推荐
机器学习,在现在生信文章中出现的频率越来越高,尤其是通过几种机器学习算法相结合的策略来识别相关疾病的特征基因,在现在是最火的生信分析热点,今天小果带着小伙伴也来蹭蹭热点,通过LASSO回归和SVM-RFE两种机器学习算法进行特征变量筛选,最后将两种算法获得的交叉基因作为特征基因;这就是小果为大家今天带来的分享内容,接下来和小果一起开启今天的学习之旅吧!
1.何为LASSO回归和SVM-RFE算法?
在进行实操之前,小果想为大家简单的介绍一下这两种算法的原理,SVM-RFE(support vector machine – recursive feature elimination)是基于支持向量机的机器学习方法, 通过删减svm产生的特征向量来寻找最佳变量;LASSO回归(logistic regression)也是机器学习的方法之一,通过寻找分类错误最小时的λ来确定变量,主要用于筛选特征变量,构建最佳分类模型。以上就是小果对两种算法的简单介绍。了解了原理之后,小伙伴们是不是有个疑问,什么时候该利用机器学习算法进行特征基因筛选?一般都是通过转录组数据差异分析结合WGCNA,获得疾病相关基因候选基因,对这些候选基因通过多种机器学习算法进行候选基因进一步筛选,这是最常见的分析套路。今天小果想利用这两种算法进行特征选择,最后取两种算法的交叉基因作为特征基因,话不多说,马上跟着小果开始今天的实操吧!
公众号后台回复“111″,领取代码,代码编号:231019
2.载入需要的R包
#安装需要的R包install.packages("tidyverse")install.packages("randomForest")install.packages('e1071')install.packages("glmnet")install.packages("VennDiagram")install.packages("ggplot2")#加载需要的R包library(tidyverse)library(glmnet)source('msvmRFE.R')library(VennDiagram)library(e1071)library(caret)
3.数据准备
svm.csv,行名为样本名,第一列为分组信息,其他列为基因所对应的表达矩阵。需要注意的是必须要有分组信息奥!
4.LASSO回归和SVM-REF筛选特征变量
train <- read.csv("svm.csv",row.names = 1,as.is = F) #后面svmRFE函数要求group的类型为factor# 转为lasso需要的格式x <- as.matrix(train[,-1])(y <- ifelse(train$group == "NR", 0,1)) #把分组信息换成01fit = glmnet(x, y, family = "binomial", alpha = 1, lambda = NULL)# 绘制LASSO回归曲线图pdf("1A_lasso.pdf", width = 30, height = 15)plot(fit, xvar = "dev", label = TRUE)dev.off()#绘制LASSO回归10折交叉验证图cvfit = cv.glmnet(x, y,nfold=10,family = "binomial", type.measure = "class")pdf("2cvfit.pdf")plot(cvfit)dev.off()#查看最佳lambdacvfit$lambda.min# 获取LASSO选出来的特征myCoefs <- coef(cvfit, s="lambda.min")lasso_fea <- myCoefs@Dimnames[[1]][which(myCoefs != 0 )](lasso_fea <- lasso_fea[-1])# 把lasso找到的特征保存到文件write.csv(lasso_fea,"3feature_lasso.csv")#SVM-REF算法输入数据input <- train#采用五折交叉验证 (k-fold crossValidation)svmRFE(input, k = 5, halve.above = 100) #分割数据,分配随机数nfold = 5nrows = nrow(input)folds = rep(1:nfold, len=nrows)[sample(nrows)]folds = lapply(1:nfold, function(x) which(folds == x))results = lapply(folds, svmRFE.wrap, input, k=5, halve.above=100) #特征选择top.features = WriteFeatures(results, input, save=F) #查看主要变量head(top.features)#把SVM-REF找到的特征保存到文件write.csv(top.features,"4feature_svm.csv")# 选前300个变量进行SVM模型构建,选取的变量越多,运行速度越慢!featsweep = lapply(1:300, FeatSweep.wrap, results, input) #300个变量# 画图no.info = min(prop.table(table(input[,1])))errors = sapply(featsweep, function(x) ifelse(is.null(x), NA, x$error))#绘制基于SVM-REF算法的错误率曲线图pdf("5B_svm-error.pdf",width = 5,height = 5)PlotErrors(errors, no.info=no.info) #查看错误率dev.off()#绘制基于SVM-REF算法的正确率曲线图#dev.new(width=4, height=4, bg='white')pdf("6B_svm-accuracy.pdf",width = 5,height = 5)Plotaccuracy(1-errors,no.info=no.info) #查看准确率dev.off()# 图中红色圆圈所在的位置,即错误率最低点which.min(errors)#比较lasso和SVM-REF方法一找出的特征变量,画Venn图(myoverlap <- intersect(lasso_fea, top.features[1:which.min(errors), "FeatureName"])) #交集#统计交叉基因有多少个summary(lasso_fea%in%top.features[1:which.min(errors), "FeatureName"])#绘制venn图pdf("7C_lasso_SVM_venn.pdf", width = 15, height = 8)grid.newpage()venn.plot<-venn.diagram(list(LASSO=lasso_fea, SVM_RFE=as.character(top.features[1:which.min(errors),"FeatureName"])), NULL,fill = c("#E31A1C","#E7B800"),alpha = c(0.5,0.5), cex = 4, cat.fontface=3,category.names = c("LASSO", "SVM_RFE"),main = "Overlap")grid.draw(venn.plot)dev.off()
5.结果文件
1.1A_lasso.pdf
该结果图片为Lasso回归图。

2.2cvfit.pdf
该结果图片为10折交叉验证图。
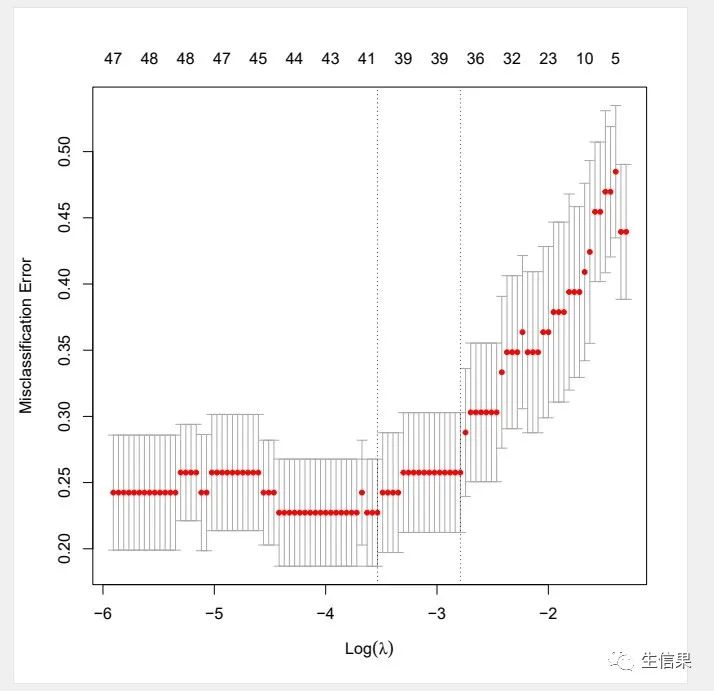

3.5B_svm-error.pdf
该结果图片为基于SVM-RFE算法5折交叉验证的错误率曲线图。


4.6B_svm-accuracy.pdf
该结果图片为基于SVM-RFE算法5折交叉验证的正确率曲线图
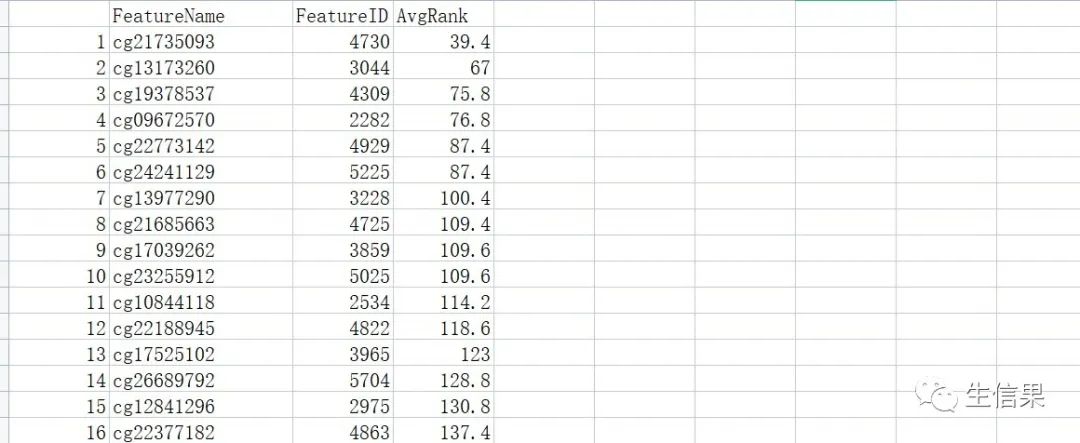
5.3feature_lasso.csv
该结果是基于Lasso算法经过特征选择,筛选出的特征基因。


6.4feature_svm.csv
该结果是基于SVM-REF算法经过特征选择,筛选出的特征基因,FeatureName表示基因名。

7.7C_lasso_SVM_venn.pdf
该结果图片为通过LASSO回归和SVM-REF算法进行特征选择,最后取两种方法得到的16个交叉基因作为特征基因的Venn图。


最终小果顺利完成了利用 lassso回归和SVM-REF两种机器学习算法进行了特征基因筛选,小伙伴们多多理解代码的意义哦~
往期推荐

