小果三分钟教会你如何使用R语言包scater分析单细胞数据
R语言在生物信息学和生物学研究中是一种常用的编程语言,它提供了丰富的工具和库来处理和分析生物数据。其中,一个重要的R包是scater,它是一个专为单细胞RNA测序(scRNA-seq)数据分析而设计的包,为研究人员在处理、可视化和理解单细胞数据方面提供了强大的支持。

scRNA-seq技术
scRNA-seq技术已经成为了研究单个细胞基因表达的主要手段。然而,这种高维度的数据需要复杂的处理和分析方法才能从中提取有用的生物学信息。在面对如此复杂的挑战时,scater包成为了研究人员不可或缺的伙伴和解决方案。
scater
scater包是一个强大的工具集,它为单细胞数据分析提供了一系列的功能,帮助用户从原始数据中提炼出有意义的结果。在这个包的引导下,用户能够在分析中执行关键的步骤,如质量控制、归一化、可视化和差异表达分析,从而更好地理解单细胞数据所蕴含的信息。
质量控制
质量控制是单细胞数据分析的首要任务之一,而scater包在这方面发挥着重要作用。通过该包,用户可以识别并移除低质量细胞,从而减少分析中的噪声和误差。这一步的关键在于保障后续分析的可靠性和准确性,使得用户能够更加自信地解释和应用分析结果。
归一化方法
除了质量控制,scater包还提供了多种归一化方法,允许用户在不同样本之间进行比较。这在研究中尤为重要,因为不同实验批次和技术差异可能会引入偏差。scater的归一化方法可以帮助用户消除这些偏差,使得数据更具可比性,从而更容易捕捉到生物学上真实的差异。
要使用scater包,可以在R中使用以下命令进行安装和加载:
公众号后台回复“111”领取代码,代码编号:231008
> install.packages("scater") #安装scater语言包> library(scater) #加载语言包
在单细胞RNA测序数据分析中,数据质量控制和预处理是确保准确可靠分析的基础步骤。scater作为一款强大的分析工具,为用户提供了一系列高效的功能来处理这些关键任务。
首先,数据质量控制方面,scater允许用户通过计算多种质量指标,如细胞的测序深度、基因表达丰度和细胞的dropout率,来评估数据的质量。这些指标可以帮助用户识别出低质量的细胞和基因,从而保留高质量的数据用于后续分析。此外,scater还可以生成详细的质量控制报告,帮助用户全面了解数据中的问题和趋势。通过识别和剔除技术噪声和批次效应,scater确保了后续分析的可靠性和准确性。
细胞聚类和降维分析是单细胞数据解析的关键步骤,有助于揭示数据中的细胞类型和结构。与此类似,scater提供了多种聚类算法和降维方法,如主成分分析(PCA)、t分布随机邻域嵌入(t-SNE)和UMAP(Uniform Manifold Approximation and Projection)。这些方法可以将高维数据映射到更低维度的空间,使用户能够在二维或三维图中观察数据的分布,更好地理解不同细胞之间的相似性和差异性。这种细胞聚类和降维分析为数据的可视化提供了有力支持,有助于研究人员更深入地探索细胞群的结构和特征。
差异表达分析是了解不同条件下基因表达差异的关键步骤,有助于识别在特定生物学过程中起关键作用的基因。scater提供了丰富的统计方法和可视化工具,使用户能够比较不同细胞类型、条件或时间点之间的基因表达水平差异。通过差异表达分析,研究人员可以深入了解在不同细胞群之间的生物学差异,为进一步的机制解析提供线索。
对于细胞发育轨迹分析,scater是一款强大的工具,它能够帮助用户揭示细胞分化和发育的动态变化过程。通过建立细胞状态的发展轨迹,研究人员可以更好地理解细胞状态之间的关系,探索细胞分化和演化的轨迹,从而深入了解发育过程中的重要性质。
scater的数据可视化功能为研究人员呈现分析结果提供了有力的支持。通过内置的多种数据可视化工具,如散点图、热图和轨迹图,用户能够以直观的方式展示他们的发现。这样的可视化有助于更清晰地传达分析结果,并为其他研究人员理解单细胞数据的洞察提供了方便。
示例:
当使用scater包时,通常需要导入单细胞RNA测序数据集,并进行一系列的预处理、可视化和分析步骤。首先,假设你已经安装了scater包和其他必要的依赖项。接下来,我们将加载示例数据集并进行基本的分析和可视化。
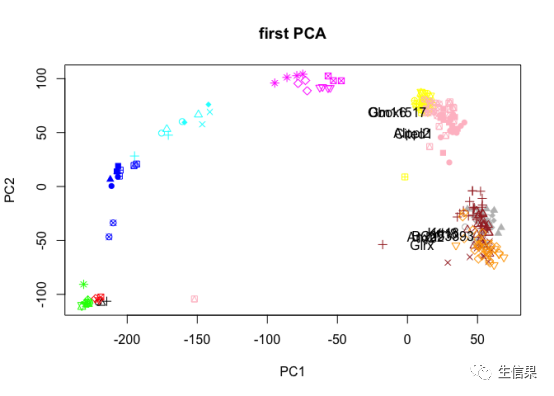
# 加载所需的库> library(scater)> library(SingleCellExperiment)> library(ggplot2)# 从scater包加载示例数据集> data("sc_example_counts")# 将示例数据集转换为SingleCellExperiment对象> sce <- SingleCellExperiment(+ assays = list(counts = as.matrix(sc_example_counts))+ )# 进行质量控制> sce <- calculateQCMetrics(sce)# 绘制细胞数目和基因数目的散点图> plotQC(sce, type = "cell", x = "total_features_by_counts", y = "n_genes_by_counts")# 进行归一化> sce <- normalize(sce)# 使用PCA进行降维> sce <- runPCA(sce)# 绘制PCA图> plotReducedDim(sce, dimred = "PCA")
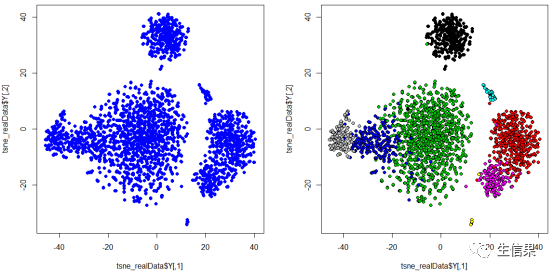
# 进行聚类> sce <- clusterCells(sce)# 绘制聚类结果的t-SNE图> sce <- runTSNE(sce)> plotReducedDim(sce, dimred = "TSNE", label = "clusters")
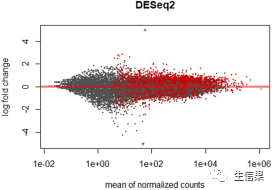
# 进行差异表达分析> sce <- DESeq(sce)# 绘制差异表达基因的MA图> plotMA(sce, contrast = "condition_B_vs_A")
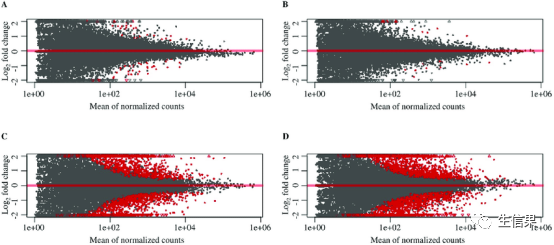
# 绘制差异表达基因的热图> plotHeatmap(sce, contrast = "condition_B_vs_A", cluster_rows = FALSE)
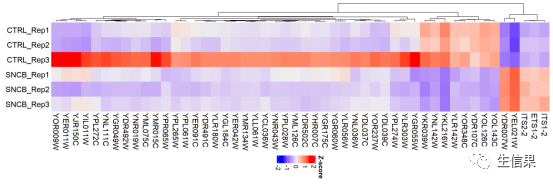
上述示例代码演示了如何使用scater包加载示例数据集、进行质量控制、归一化、降维、聚类和差异表达分析,并生成细胞数目与基因数目的散点图、PCA图、t-SNE图、MA图和热图。这些可视化图表有助于我们直观地理解数据的特征和模式,进而从中挖掘生物学相关的信息。
结论
差异表达分析在scRNA-seq数据分析中扮演着至关重要的角色,而scater包为这一环节提供了强大的支持。差异表达分析旨在识别在不同细胞类型或条件之间表达水平显著变化的基因,这有助于我们更深入地理解细胞的功能和调控机制。scater包通过实现多种差异表达分析方法,如edgeR和DESeq2,为研究人员提供了多样化的工具,帮助他们从海量的数据中挖掘出有意义的生物学信息。
在使用scater包进行差异表达分析时,研究人员可以根据实际情况选择适合的方法。例如,edgeR和DESeq2是两种常用的统计方法,它们能够考虑到数据的离散性和批次效应,从而更准确地捕捉基因表达之间的差异。通过这些方法,用户可以鉴定出在不同细胞状态或处理条件下表达显著变化的基因,这些基因往往与特定的生物学过程密切相关。
通过差异表达分析,研究人员可以找到一系列与细胞类型特异性或环境响应相关的基因。这些基因可能在特定的发育阶段、疾病状态或环境刺激下发挥着关键作用。通过深入研究这些差异表达的基因,我们可以更好地理解细胞的功能和调控机制,揭示其在不同生理和病理过程中的作用。
文章小结
以上就是对R语言包scater的简单介绍啦,scater是一个强大的R包,为生物学家和生物信息学研究人员在单细胞RNA测序数据分析中提供了丰富的功能和工具。它不仅可以帮助用户进行质量控制、归一化和可视化,还支持差异表达分析,从而在研究细胞多样性、功能和调控机制方面提供有力的支持。无论是在癌症研究、免疫学还是发育生物学领域,scater都在帮助科研人员深入挖掘单细胞数据背后的生物学信息。
小伙伴们,今天有没有学到新知识呢,想要继续了解R语言内容可以持续关注小果~或者可以扫描下面的二维码进行咨询哦~
往期推荐

