相关性分析热图怎么画才能足够直观?快进来学习!
点击蓝字 关注我们
小伙伴们好!今天的主题大家一定都很熟悉,相关性分析,几乎所有的数据分析过程都少不了它,同时热图更是相关性分析不可少的可视化方式,它可以帮助我们直观地了解数据中各个变量之间的相关性程度。通过色彩的渐变和聚类算法,相关性热图能够让数据中的模式和趋势一目了然,为我们深入分析提供了有力支持。
可以这么一个常用的分析方法,大海哥这几天却看到很多小伙伴都有所苦恼,自己画的热图不是很好看,不直观,只有一个R值,怎么可以同时显示P值和R值呢?看到大家的苦恼后,大海哥马不停蹄回去写代码,终于大功告成,这就来和大家分享!
大海哥今天使用的案例是要对基因表达与免疫细胞水平做一个相关性分析,首先来看看我们的数据吧!
首先是我们的基因表达数据
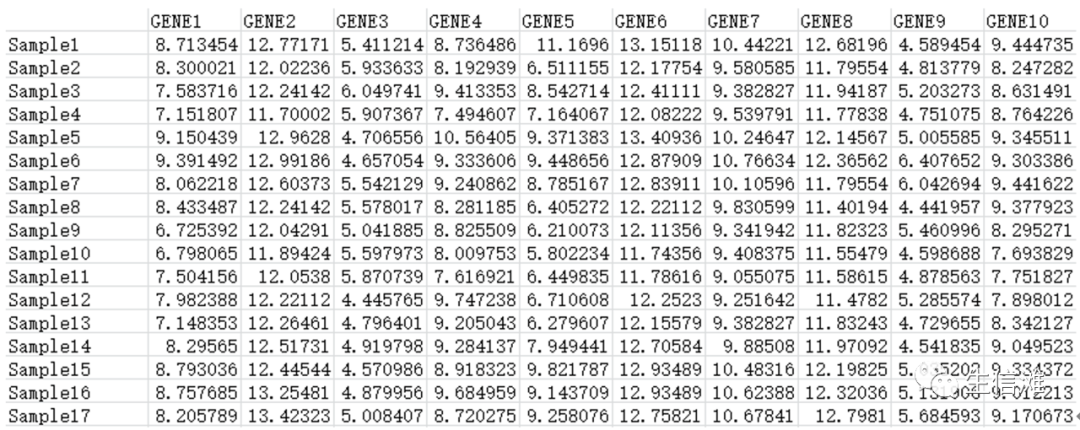
然后是我们的免疫细胞水平数据(免疫细胞水平数据的分析获得方式在大海哥的往期文章中有介绍哦!)

有了这两个数据,就可以开始相关性分析啦!
代码展示
#首先导入我们需要用到的R包library(psych)library(ggpubr)#列出我们需要分析的基因sig_gene <- c("GENE1"," GENE2"," GENE3"," GENE4"," GENE5")#读取基因表达矩阵expr <- read.csv("exp.csv",header=T,row.names=1,check.names=F)#转置,要求行为样本,列为基因,符合的数据就不需要这一步啦expr_data <- t(expr)#读取免疫细胞数据immu_data <- read.table("ciber_result.txt",sep="t",header=T,row.names=1)#去掉最后三列的"P.value","Correlation","RMSE"(这里用的是cibersort计算方式,所以会有这三列数据哦)immu_data=immu_data[,1:22]#读取关键基因基因表达(如果大家是全部基因分析的话,就不需要这一步了,直接hub <- expr_data就可以啦!)hub <- expr_data[,sig_gene]my <- immu_data#计算相关性(这里使用的是spearman方式,大家也可以选择pearson等其他方式哦!)d <- corr.test(hub,my,use="complete",method = 'spearman')#获取计算结果r值和p值r <- d$rp <- d$p#来画一个普通的热图看看ggcorrplot(t(d$r), show.legend = T,p.mat = t(d$p), digits = 2, sig.level = 1,insig = 'blank',lab = T)

#这就是普通的热图,第一眼看上去感觉很棒,但是细琢磨发现这只是根据R值来决定的,没有办法直观的看出P值啊,该怎样同时显示P值和R值呢?
#这里就要清楚我们的老朋友ggplot2啦!
#导入R包library(ggplot2)#导入相关性分析结果cor_result<-read.table("corr_result.txt",header=T,check.names=F,sep="t")#这一步很多小伙伴懵了,数据哪来的?别着急,容我慢慢介绍#首先导出上一步得到的P值和R值write.table(t(rbind(r,p)), "corr_result.txt",sep=”t”)#然后就得到了如下的数据
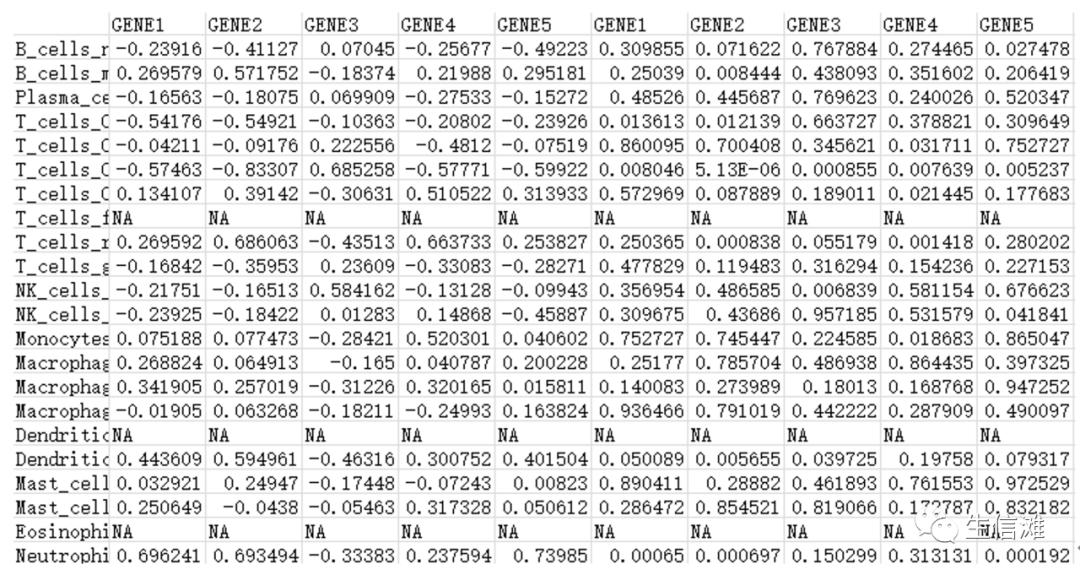
#接下来我们需要对这个数据进行手动处理一下,整理成下面这个样子

#一共四列,第一列为基因,第二列为免疫细胞名,第三列为相关性R值,第四列为相关性p值#整理完数据然后就可以开始绘图啦#先去除空值哦,数据里有一些NA都需要去掉再开始绘图#导入数据,指定基因和免疫细胞以及R值对应的列gg<-ggplot(cor_result,aes(y=im_cell,x=Gene,fill=Cor))+geom_tile()+theme_bw()+#设置标题字体大小及颜色theme(plot.title = element_text( face = "bold", color = "black", size = 15, hjust = 0.5))+theme(panel.grid.major = element_blank()) +theme(legend.key=element_blank())+#设置X轴刻度theme(axis.text.x=element_text(angle=45,vjust=0.2,hjust=0.3,size=13,colour="black"))+#设置标签字体大小颜色theme(axis.title=element_text(size=13,colour="black"))+#设置y轴刻度theme(axis.text.y=element_text(size=13,colour="black"))+#定义相关性模块颜色渐变scale_fill_gradient2(low="blue",mid="white",high="red",limits=c(-1,1))+labs(title="Correlation between immune cells and genes",y = "Immune Cell",x="Gene")+geom_text(aes(label=(paste(round(Cor,2),paste("(",signif(p.value,3),")",""),sep="n"))),size=5,color="black",hjust=0.5)+scale_y_discrete(limits=rev(levels(cor_result$im_cell)))#保存图片ggsave(gg,file="corr.pdf",width=15,height=12)#然后让我们看看结果图是什么样子

#看看这个图,直观好看,既有P值又有R值,每个格子中上半部分为R值,下半部分为P值,是不是比之前的那个图好多了?这个热图不仅适用于基因与免疫细胞像关性分析,基因之间相关性分析也是可以的哦!简直就是万能通用!
点击“阅读原文”进入网址