小果带你探索GATK-HaplotypeCaller 变异检测原理
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

-
Hello,这里是小果,我们将持续不断为大家带来生物信息软件和分析的系列讲解,欢迎大家关注我们,这样才能更及时看到我们的文章哦。
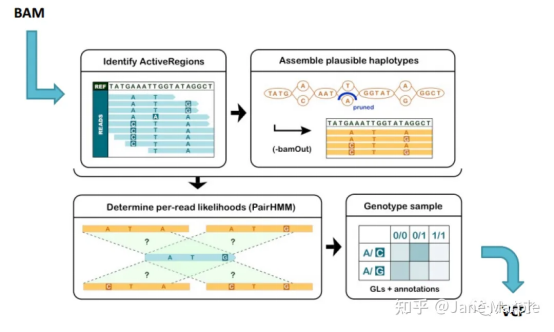
GATK-HaplotypeCaller简介
众所周知,基因组学最重要的内容之一就是变异检测,一般测序公司会完成snp calling的工作,但是作为一个合格的生信狗,怎能不了解snp calling的原理呢。
目前snp calling主流的方法有bcftools call和GATK HaplotypeCaller,二者原理相近,一般测序公司会同时进行,取snp的交集,今天小果就带大家了解一下GATK HaplotypeCaller的原理。

GATK-HaplotypeCaller的变异检测的基本原理
当HaplotypeCaller运行时,程序会从头开始组装短序列,并进行比对,当序列余参考基因组存在差异时,它可能时测序错误、软件精确度不够、抽样误差等原因,当组装当变异活跃的区域时,HaplotypeCaller会局部重新组装区域来确定基因型,假设一共由30条read比对到这个位点上,其中有20条read都和参考基因组不同,那么很大概率上他就是一个真实变异位点。

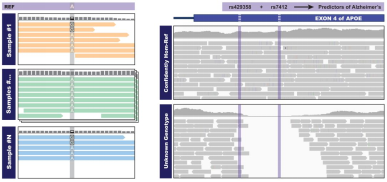
SNP calling策略的选择
当你有多个样本时,是应该将所有样本进行snp calling后再merge vcf文件。还是将所有样本作为input文件进行snp calling呢?GATK提供了多个snp calling的策略。
single sample calling:每一个样本单独进行snp calling,然后将每个样本snp calling结果再合成一个总的vcf文件。
batch calling:将样本分组进行snp calling,然后再merge在一起
joint calling:所有样本的bam一起call 出一个包含所有样本变异信息的vcf文件。
通常来说,如果条件允许(服务器:你闭嘴)的话,使用joint calling是较优的选择。因为在joint calling中,所有样本同时进行snp calling,对于低频位点具有更好的calling效果,因为不同样本的测序深度、覆盖率不同,单样本进行snp calling很容易由遗漏,当所有样本同时进行calling时,低频位点可能在别的样本中频率较高,从而更全面的call基因组的变异位点。
好啦,这期就讲到这里,下期咱们继续讨论变异位点的筛选条件。

微信号 | 18502195490
知乎 | 生信果
点击“阅读原文”立刻拥有
↓↓↓