手把手教你免疫细胞与基因表达可视化-棒棒糖图(Lollipop包)
点击蓝字,关注我们
之前小师妹已经做过了基因表达数据以及免疫细胞数据之间的关系,但有着较多的图,以帮助更好地理解两者之间的关系,并在图形中标示出不同程度的相关性,以及在相关性较高且 p 值较小的情况下使用红色标记。
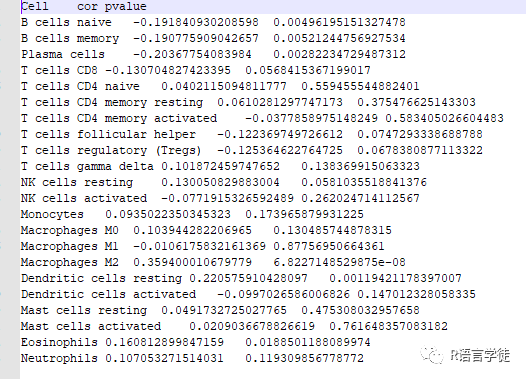
上期小师妹带你学习单基因与免疫浸润水平分布及相关性分析的cor.result分析结果,对多种免疫细胞相关性和p值得结果文件。
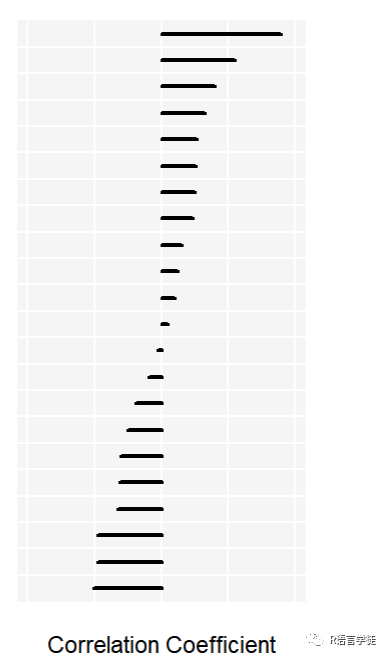
本期主要给大家带来绘制一个相关性系数的”Lollipop”图。该图用于展示不同变量(在此代码中表示为免疫细胞)与目标变量(在此代码中表示为基因表达相关性)之间的相关性,同时在图形中以不同的圆圈颜色和大小表示相关性系数的显著性和强度。用一张图就把所有免疫细胞与基因的关系展示清楚。
inputFile="cor.result.txt" #输入文件setwd("") #设置工作目录
#读取输入文件data = read.table(inputFile, header=T, sep="t", check.names=F)
首先,通过读取输入文件”cor.result.txt”,该文件包含了不同免疫细胞类型与基因之间的相关性和p-value等信息。
#定义圆圈颜色的函数p.col = c('gold','pink','orange','LimeGreen','darkgreen') #五种颜色的向量
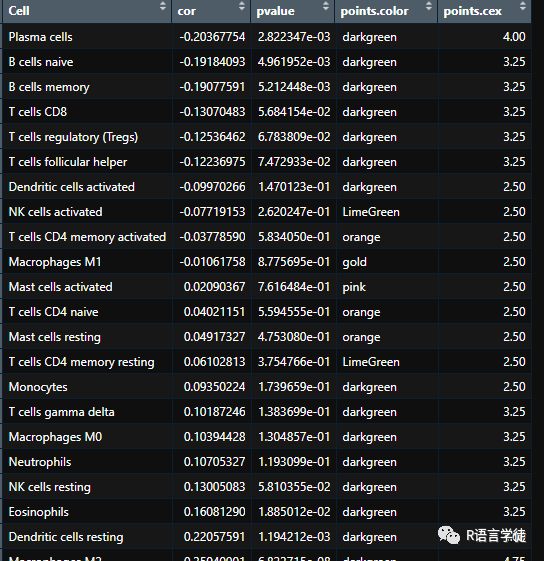
定义了两个函数,分别用于根据相关性系数确定圆圈的颜色和大小。
这里采用了预先定义的颜色和大小范围,根据相关性和p-value来确定圆圈的颜色和大小。
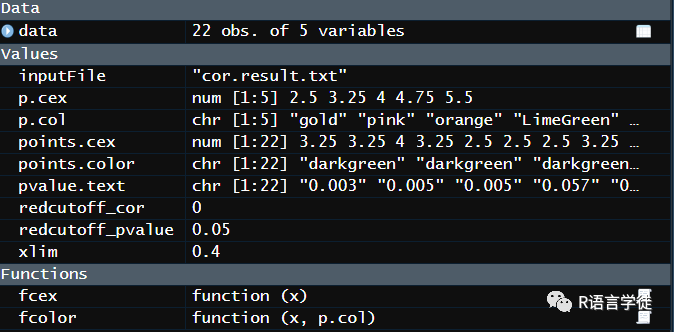
color = function(x,p.col){#fcolor函数接受两个参数:x是相关性系数的值,p.col是颜色选项的向量。color = ifelse(x>0.8,p.col[1],ifelse(x>0.6,p.col[2],ifelse(x>0.4,p.col[3],ifelse(x>0.2,p.col[4], p.col[5]))))# 如果相关性系数大于0.8,则使用p.col中的第一个颜色('gold')。#如果相关性系数介于0.8和0.6之间,则使用p.col中的第二个颜色('pink')。#如果相关性系数介于0.6和0.4之间,则使用p.col中的第三个颜色('orange')。#如果相关性系数介于0.4和0.2之间,则使用p.col中的第四个颜色('LimeGreen')。#如果相关性系数小于0.2,则使用p.col中的第五个颜色('darkgreen')。return(color)}#定义设置圆圈大小的函数p.cex = seq(2.5, 5.5, length=5)fcex = function(x){x=abs(x)cex = ifelse(x<0.1,p.cex[1],ifelse(x<0.2,p.cex[2],ifelse(x<0.3,p.cex[3],ifelse(x<0.4,p.cex[4],p.cex[5]))))# 如果绝对值小于0.1,则使用p.cex中的第一个大小值(2.5)。#如果绝对值介于0.1和0.2之间,则使用p.cex中的第二个大小值(3.125,取自2.5和5.5的中点)。#如果绝对值介于0.2和0.3之间,则使用p.cex中的第三个大小值(3.75,取自2.5和5.5的1/4处)。#如果绝对值介于0.3和0.4之间,则使用p.cex中的第四个大小值(4.375,取自2.5和5.5的1/2处)。#如果绝对值大于等于0.4,则使用p.cex中的第五个大小值(5.0)。return(cex)}#根据pvalue定义圆圈的颜色points.color = fcolor(x=data$pvalue,p.col=p.col)data$points.color = points.color#根据相关系数定义圆圈的大小points.cex = fcex(x=data$cor)data$points.cex = points.cexdata=data[order(data$cor),]
########绘制图形#########设置图形的布局,包括左边的绘图区域和右边的图例。然后在绘图区域内进行如下操作:#绘制x轴范围,设置图形的背景颜色、边界颜色,绘制网格线。# 使用segments函数绘制图形的线段,表示相关性系数。#使用points函数绘制圆圈,表示相关性的大小,颜色和大小由之前定义的信息决定。#使用text函数在图上标注免疫细胞的名称。#使用text函数在图上标注p-value信息,其中显著性较高的相关性显示为红色。#绘制x轴。xlim = ceiling(max(abs(data$cor))*10)/10pdf(file="Lollipop.pdf", width=9, height=7) #输出图形#下面就是图形各种参数的调整
layout(mat=matrix(c(1,1,1,1,1,0,2,0,3,0),nc=2),width=c(8,2.2),heights=c(1,2,1,2,1))par(bg="white",las=1,mar=c(5,18,2,4),cex.axis=1.5,cex.lab=2)plot(1,type="n",xlim=c(-xlim,xlim),ylim=c(0.5,nrow(data)+0.5),xlab="Correlation Coefficient",ylab="",yaxt="n",yaxs="i",axes=F)rect(par('usr')[1],par('usr')[3],par('usr')[2],par('usr')[4],col="#F5F5F5",border="#F5F5F5")grid(ny=nrow(data),col="white",lty=1,lwd=2)

#绘制水平的网格线,将图形分成等间隔的行。ny=nrow(data)表示网格的行数与数据的行数相同。网格线的颜色设置为白色,线型为1,线宽为2。#绘制图形的线段segments(x0=data$cor,y0=1:nrow(data),x1=0,y1=1:nrow(data),lwd=4)
#绘制图形的圆圈points(x=data$cor,y = 1:nrow(data),col = data$points.color,pch=16,cex=data$points.cex)#展示免疫细胞的名称text(par('usr')[1],1:nrow(data),data$Cell,adj=1,xpd=T,cex=1.5)
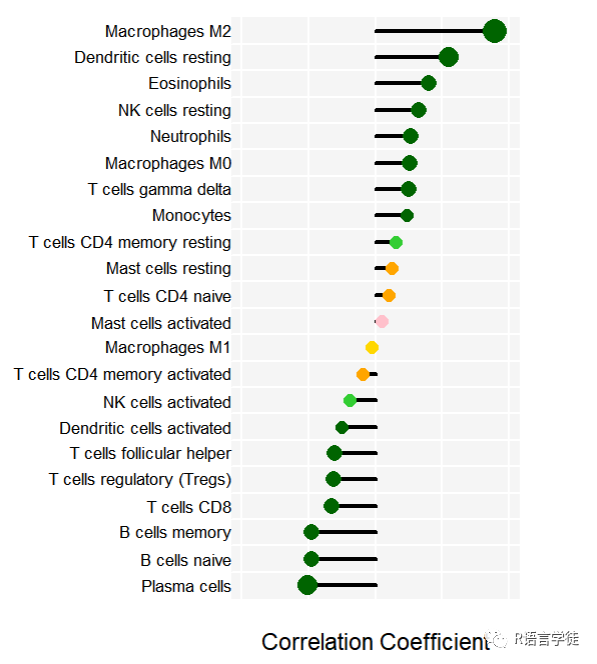
#展示pvaluepvalue.text=ifelse(data$pvalue<0.001,'<0.001',sprintf("%.03f",data$pvalue))redcutoff_cor=0redcutoff_pvalue=0.05text(par('usr')[2],1:nrow(data),pvalue.text,adj=0,xpd=T,col=ifelse(abs(data$cor)>redcutoff_cor & data$pvalueaxis(1,tick=F)
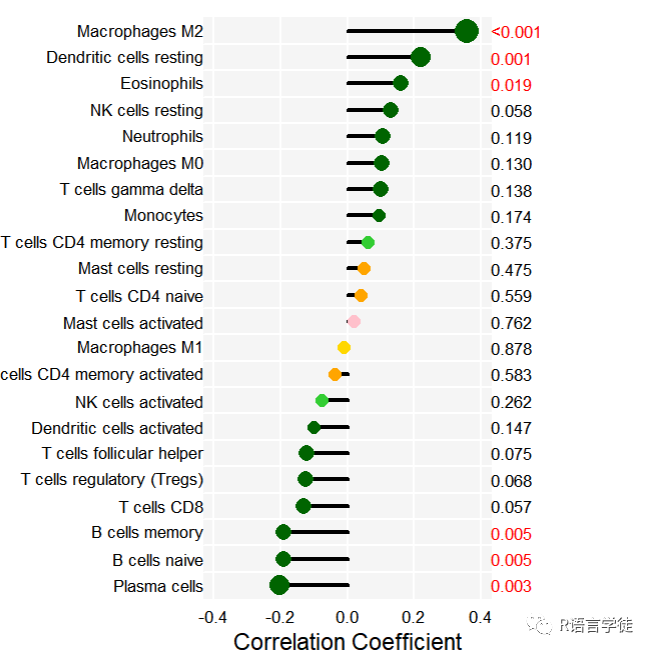
#绘制圆圈大小的图例par(mar=c(0,4,3,4))plot(1,type="n",axes=F,xlab="",ylab="")legend("left",legend=c(0.1,0.2,0.3,0.4,0.5),col="black",pt.cex=p.cex,pch=16,bty="n",cex=2,title="abs(cor)")#绘制圆圈颜色的图例par(mar=c(0,6,4,6),cex.axis=1.5,cex.main=2)barplot(rep(1,5),horiz=T,space=0,border=NA,col=p.col,xaxt="n",yaxt="n",xlab="",ylab="",main="pvalue")axis(4,at=0:5,c(1,0.8,0.6,0.4,0.2,0),tick=F)dev.off()
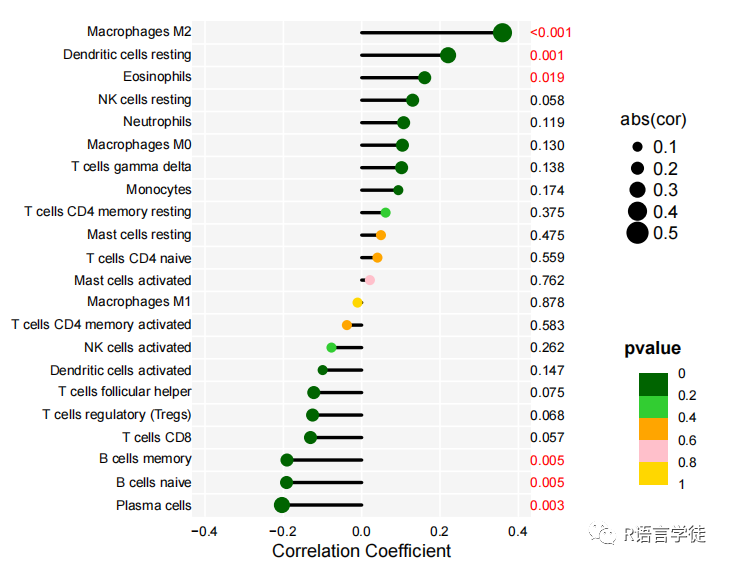
生成一个具有不同圆圈颜色和大小的”Lollipop”图,用于展示免疫细胞与基因表达之间的相关性,同时强调显著性和强度。这个图简单清楚的将大部分信息包括其中了,以上就是本期内容,需要代码和示例文件私小师妹。下期将为你带来更多R语言的骚操作技巧,以下推荐的是一个多功能的生信平台。
云生信平台链接:http://www.biocloudservice.com/home.html(点击文末阅读原文查看哦)


