XGBoost也可以用于模型特征提取?快来get!
点击蓝字 关注我们
嗨!亲爱的小伙伴们,大家好!我是大海哥。今天大海哥要和大家聊聊机器学习领域里的一颗闪耀之星✨ ——XGBoost算法!
XGBoost,全称Extreme Gradient Boosting,是一种强大的机器学习算法,我们都听说过这句话,Machine learning is changing the world! 而XGBoost更在各种数据挖掘和预测任务中表现出色,它以其卓越的性能和出色的鲁棒性,成为众多数据科学家和机器学习从业者的最爱哦!这个算法的闪亮之处在于它的梯度提升框架,可以在处理分类和回归问题时都表现得相当出色。它会通过多轮迭代,逐步提升预测模型的准确性,不断优化模型表现。就像是一位聪明的学生,不断吸收知识,进步突飞猛进!
其还有一个主要特点,就是它对于复杂的数据集和高维特征的适应能力非常强大。它不容易受到过拟合的困扰,而且能够处理缺失值和异常数据,使得数据预处理变得相当便捷。就像是一位数据的魔术师,总能从混沌的数据中找到规律!而且,XGBoost的模型解释性也很强,这意味着你可以更好地理解模型是如何做出预测的,从而为业务决策提供支持。
那么介绍了这么多,就让大海哥带领小伙伴们来领悟一下这个算法的魅力吧!
#首先加载R包require(xgboost)require(Matrix)require(data.table)library(survival)data(cancer, package="survival")df <- data.table(colon, keep.rownames = FALSE)df<-df[1:100,]#这里我们选择前一百个数据进行处理,在使用自己数据的时候一定要处理缺失值head(df)

#把结局变量生存状态转换为因子型df$status<-factor(df$status,levels = c(0,1),labels = c("alive","dead"))#查看数据结构str(df)
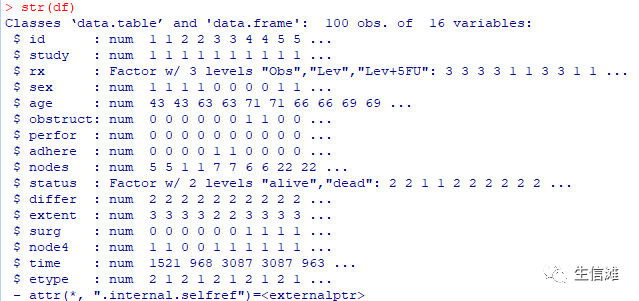
#将年龄新增一个变量为二分类变量head(df[,AgeCat:= as.factor(ifelse(age >=65, "Old", "Young"))])
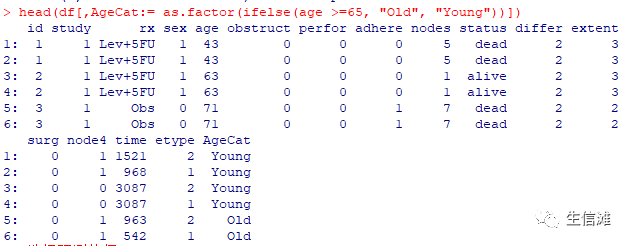
#选择预测特征df<-subset(df,select = c(-id,-study,-age,-time))#实现XGBoost#将数据转化为稀疏矩阵sparse_matrix <- sparse.model.matrix(status~ ., data = df)[,-1]#查看前几行head(sparse_matrix)

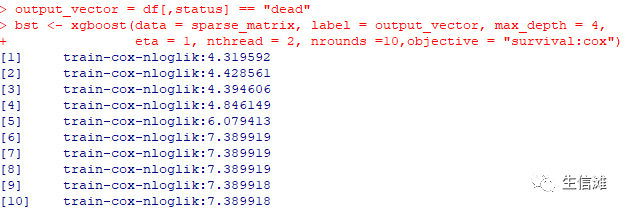
output_vector = df[,status] == "dead"#建立模型!bst <- xgboost(data = sparse_matrix, label = output_vector, max_depth = 4,eta = 1, nthread = 2, nrounds =10,objective = "survival:cox")
#在该模型中nrounds是迭代次数,数值越大运行时间越长。objective是模型类型,我们常用的一般是二分类logistics回归和Cox回归,这里我选择后者。#每行的train-cox-nloglik代表模型效果,一般越小越好。#但是要注意,在这里我们观察到在第2次之后该值逐渐增大,这表示模型出现了过度拟合,因此,我们选择迭代次数2来构建模型bst <- xgboost(data = sparse_matrix, label = output_vector, max_depth = 4, eta = 1, nthread = 2, nrounds =2,objective = "survival:cox")#然后关键的一步到了#提取变量重要性importance <- xgb.importance(feature_names = colnames(sparse_matrix), model = bst)head(importance)
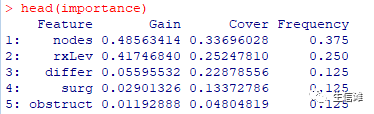
我们其实再深入一点分析这个模型。我们已经发现哪些特征可以预测疾病结局。但我们还不知道这些特征的作用。例如,我们想知道不同治疗方案对患者预后是否有影响?因此,我们在上述分析的基础上添加两个参数data和label来解决这一问题。importanceRaw <- xgb.importance(feature_names = colnames(sparse_matrix), model = bst, data = sparse_matrix, label = output_vector)importanceClean <- importanceRaw[,`:=`(Cover=NULL, Frequency=NULL)]head(importanceClean)
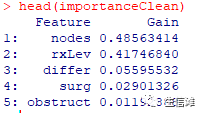
#可以看到最重要的特征就是这几个了!#然后我们对其进行可视化一下xgb.plot.importance(importanceClean, rel_to_first = TRUE, xlab = "Relative importance")
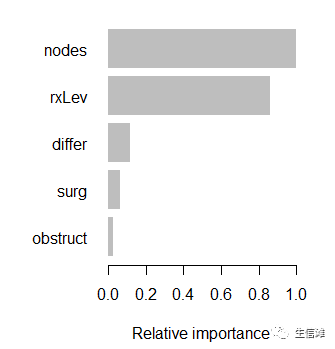
根据该结果,我们可以看出,对于预测患者预后最重要的前三个变量分别为nodes、surg和obstruct。
难道到这里就完了吗?
小伙伴们肯定也会说,这个图也太难看了!强烈要求画好看一点!放心,接下来我们用ggplot2重新画一个重要性评估图。#因为importanceClean是一个data.table,因此先转换为数据框data<-as.data.frame(importanceClean)#接下来开始绘图,这里需要注意:#1.如果我们以Feature为x,Gain为y值绘制条形图,结果肯定和原图不一样,#所以需要对坐标轴进行转换;#2.上次我们也说到变量排序的事,这里还是用reorder函数对y值由大到小重新排序p<-ggplot(data=data,mapping=aes(x=reorder(Feature,Gain),y=Gain/0.551046708,fill=Feature))+geom_bar(stat = 'identity',width = 0.8,position=position_dodge(0.7))+scale_fill_brewer(palette ="Set1")+coord_flip()+#坐标轴变换ylab("Variable relative importance")+xlab("")+theme_bw()+theme(legend.position = "")ggsave("vip.png",plot = p,width = 4.5,height = 5,dpi = 600,units = "in",device = png)
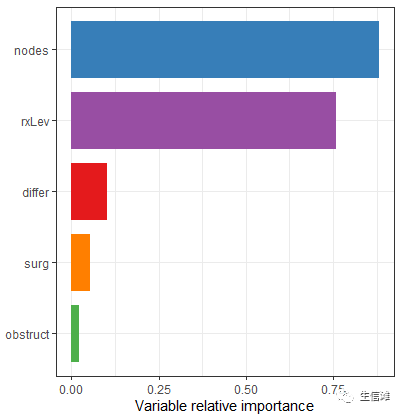
这就完美了!五个特征用五种颜色表示,好看又实用!

点击“阅读原文”进入网址
