【一文一分钟】带你读懂WGCNA结果图
点击蓝字
关注小图
Hello~又到小图【一文一分钟】课堂时间
前面的文章小图介绍了WGCNA,大家去搜索WGCNA肯定也会找到很多教程。其实我们最重要的一步是理解算法的原理,跑完这个代码,能够对结果进行一个正确的解读那么如何解读WGCNA的结果?这一文小图会进行详细介绍。
今天不讲代码,只讲讲这些图怎么看,能表达什么内容。想看代码可以移步 你的高分文章还差一套WGCNA分 后再返回看此文
WGCNA分析流程图主要包括:构建共表达网络→模块识别→模块与性状关联→模块相关性→关键模块中识别核心基因

一、样品聚类,通过聚类查看是否存在异常样品

输出文件解读
结果显示:可以看到,最左边明显有几个样品和别人不在一条枝叶上,因此,我们需要将那几个明显偏离的分支给剪去。
二、在构建共表达网络之前,还有一步比较关键的步骤——寻找最优软阈值(soft thresholding或power),使构建的网络更符合无标度拓扑结构。这时,我们会看到如下图:

输出文件解读
Scale independence
Power一般选在16以下·R方要高于0.8。如果调试后还不能满足可能是表达矩阵没有处理恰当,或者不满足分析条件比如样本里太少,基因数目过多等原因(发表文章一般不放正文)
根据结果,绘制power值对应的散点图;结果显示,1到2,3到4,整个变化趋势都还很明显,而5往后,随着power值的增大,其变化趋势已经不明显了;因此,选取7或者8为最佳的power值。
mean connectivity(为不同软阈值情况下的网络连通度)
平均连接度和power取值间关系选取power般在平均连接度变化呈平稳处(发表文章一般不放正文)
三、基于最优软阈值构建共表达网络,将基因划分到不同模块后,可以绘制基因聚类树:

输出文件解读
Cluster Dendrogram
聚类树状图,这个图可以分为两部分看:上半部分是基因的层次聚类树状图,下半部分是基因模块,也就是网络模块。上下对应,可以看到距离较近的基因(聚类到同一条分支)被划分到了同一模块。Merged是合并相似模块之后的颜色。每个颜色代表一个模块,灰色代表里面基因不属于任何模块。
四、接着,计算模块与感兴趣的临床特征之间的相关性与显著性,绘制相关性热图:

输出文件解读
Module-trait relationships
上图中,最左侧的颜色块代表模块,最右侧的颜色条代表相关性范围。中间部分的热图中,颜色越深相关性越高,红色表示正相关,蓝色表示负相关;每个单元格中的数字表示相关性和显著性。如上图,plink模块与性状表现为正相关且相关性最高。此时,我们可以选择相关性最高的plink模块作为关键模块。一般,我们会按相关性的绝对值筛选最相关模块,即负相关模块也应该考虑在内。需要注意的是,grey模块中包含了所有未参与聚类的基因,因此是无效模块,不应用于后续分析。
五、也可以看看模块之间的聚类树
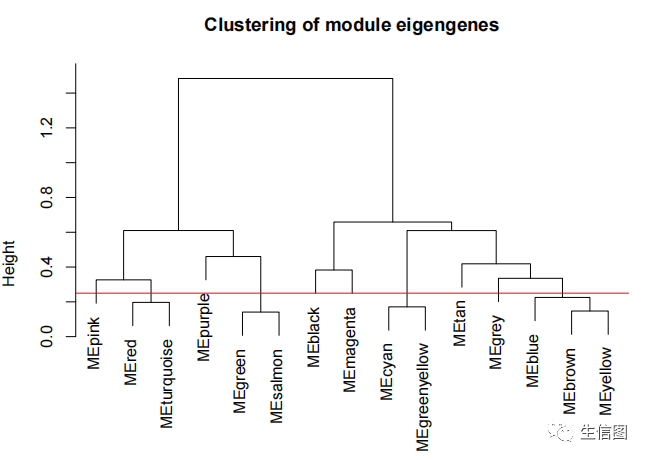
之后针对关键模块plink和感兴趣的性状进一步挖掘,看看基因与模块的相关性(Module Membership, MM)和基因与性状的相关性(Gene Significance, GS)之间是否有某种关联。

输出文件解读
通过以上散点图,可以发现MM和GS呈正相关,说明这些与性状高度相关的基因,在关键模块中也扮演着举足轻重的角色。
六、我们也可以基于关键模块中基因之间的相似性,构建关键模块网络,以Cytoscape软件可视化展示网络中基因之间的互作关系:

最后,也是最重要的一步——基于关键模块筛选核心基因。这一步的可用方法比较多,没有固定限制。比如,可以根据MM>0.8且GS>0.3筛选核心基因;可以根据关键模块的网络degree筛选top20作为核心基因;也可以计算kME值,依据|kME|≥0.7筛选;或者将网络导入Cytoscape后,利用插件cytoHubba筛选。
下课啦~今天就到这里了。有兴趣的朋友可以继续关注小图的微信公众号(生信图)定期分享生信小技巧知识。另外,云生信生物信息学平台(http://www.biocloudservice.com/home.html),可以深入挖掘差异表达基因,完成多种高级分析内容,例如WGCNA、GSEA、PPI等内容,为您提供最全面、最高效的生物信息分析以及整合利用公共数据库的解决方案。
欢迎使用:云生信平台 ( http://www.biocloudservice.com/home.html)

|
往期推荐 |
|
|
|
|
|
|