不容错过的基因家族进化分析的知识点!

这次小果给大家带来的干货是关于一个物种里面的基因家族进化分析。
什么是基因家族?
基因家族(gene family),是来源于同一个祖先,由一个基因通过基因重复而产生两个或更多的拷贝而构成的一组基因,它们在结构和功能上具有明显的相似性,编码相似的蛋白质产物。
为什么要进行基因家族分析?
1.基因家族的基因在物种之间都是比较保守的,通过基因家族分析可以得到某物种特有的家族基因,而这些基因则有可能与该物种的特异性有关。
2. 通过对多物种构建系统发育树,从而得到物种起源进化或亲缘关系方面的信息,并为后续遗传操作提供参考。
3. 基于单拷贝基因家族,可估算出物种间的分歧时间。
4. 可以挖掘某物种中哪些基因发生了明显的扩增/收缩,这些变化可能与该物种某些强/弱化的生物学分子功能有关。
5. 通过分析家族基因在进化过程受到的正向选择,确定与该物种环境适应性相关的基因。
怎么进行基因家族分析?
通过以上的解释,大家应该能大概明白怎么去预测一个物种中的基因家族了吧。不过要分析,最好要有多点的内容,所以接下来小果会讲一下基因家族分析的大致流程。
1,挑选一个基因id,并使用十种动物
2,从ensembl数据库中,下载pep文件
https://asia.ensembl.org/index.html
2.1选择自己目标物种
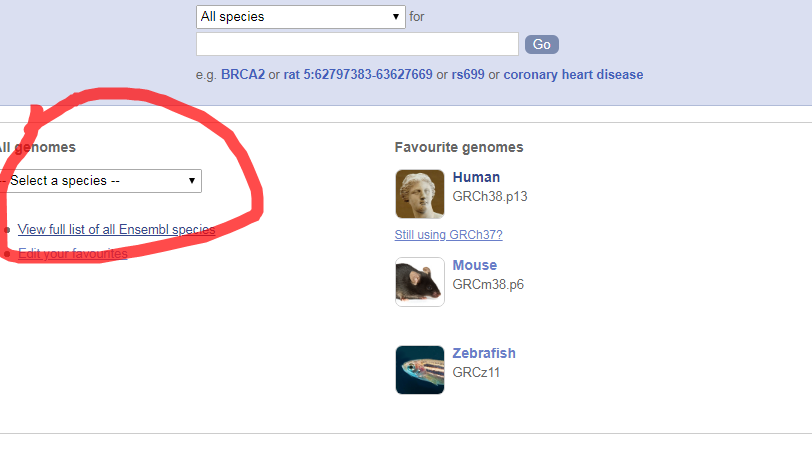
2.2点击下载DNA序列
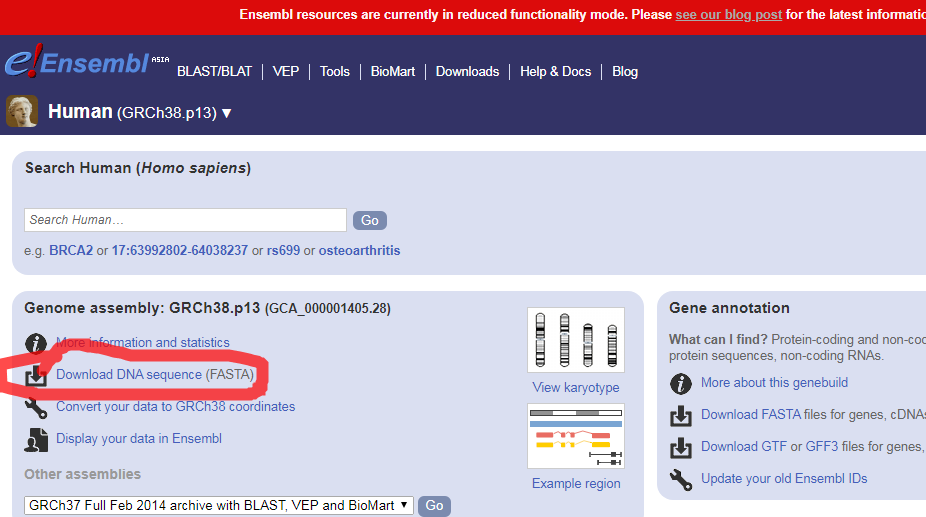
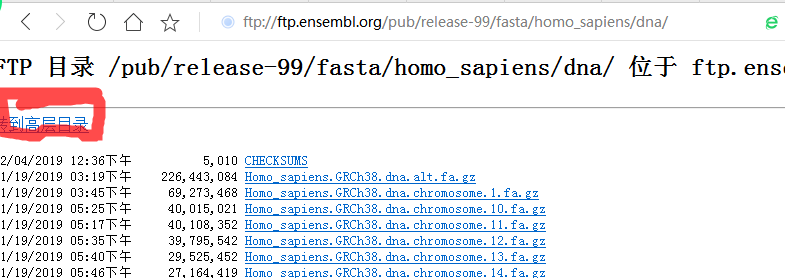
2.3我们的目的下载pep文件,不是dna文件,改变路径,先回到上一级
2.4点击pep
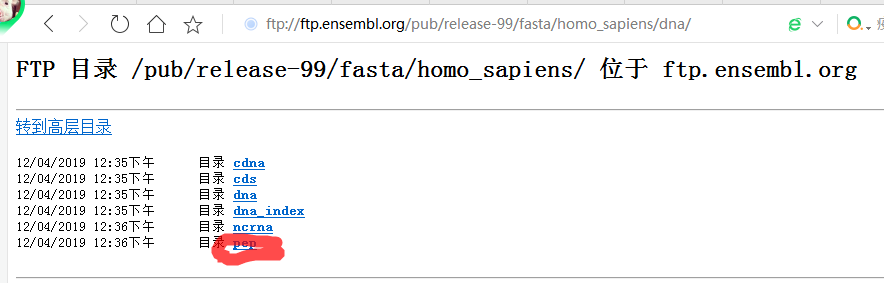
2.5 下载xx.pep.all.fa.gz文件,下载后需要解压

也在浏览器中直接输如
ftp://ftp.ensembl.org/pub/release-99/fasta/ 物种名字(拉丁文)/dna/
3,从ncbi中获取pfam模型id
https://www.ncbi.nlm.nih.gov/
3.1
3.2
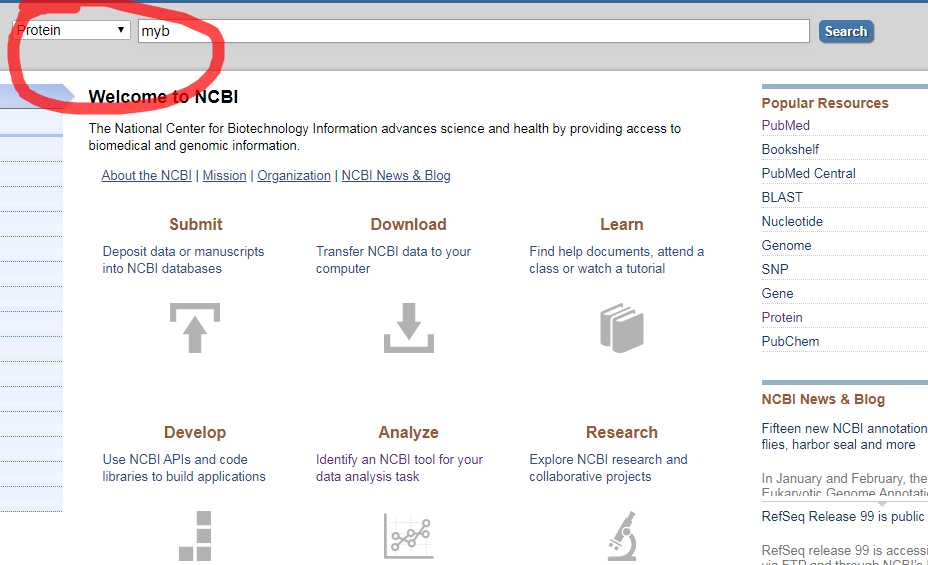
3.3

3.4点击自己目标物种蛋白
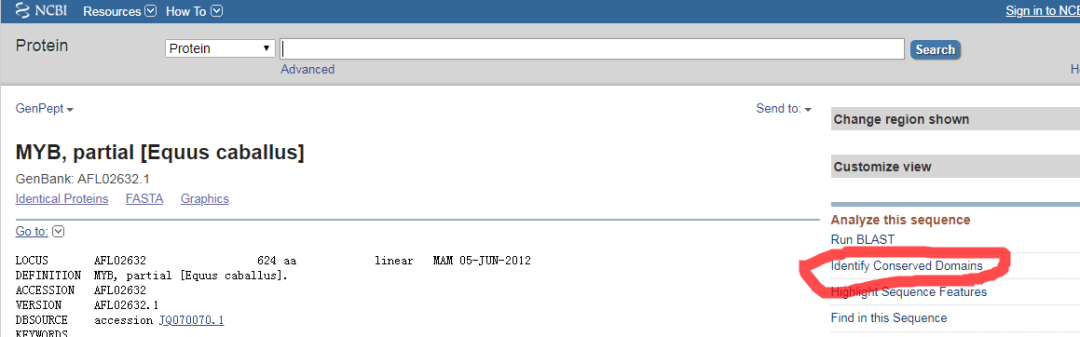
3.5选择自己的基因,并点击链接入口

3.6 pfam模型id
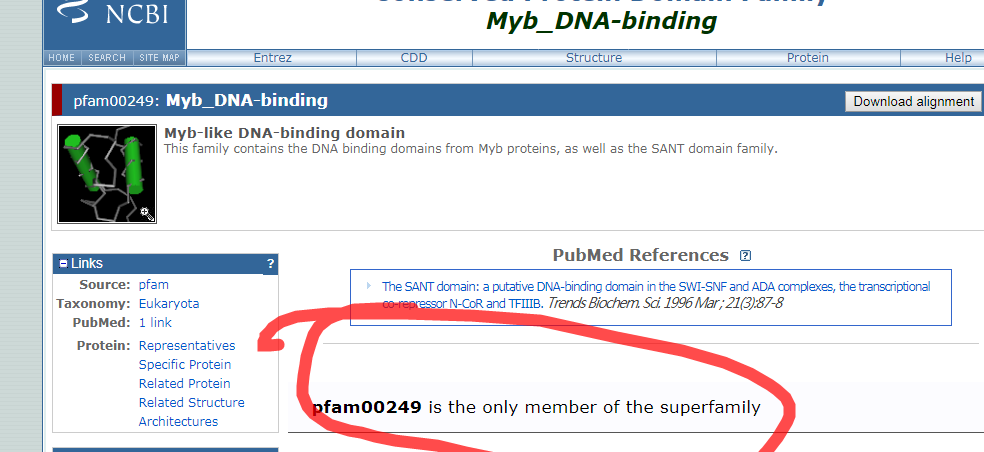
4,从pfam网站,获取pfam模型
https://pfam.xfam.org/
4.1将pfam00249中“pfam”改为PF
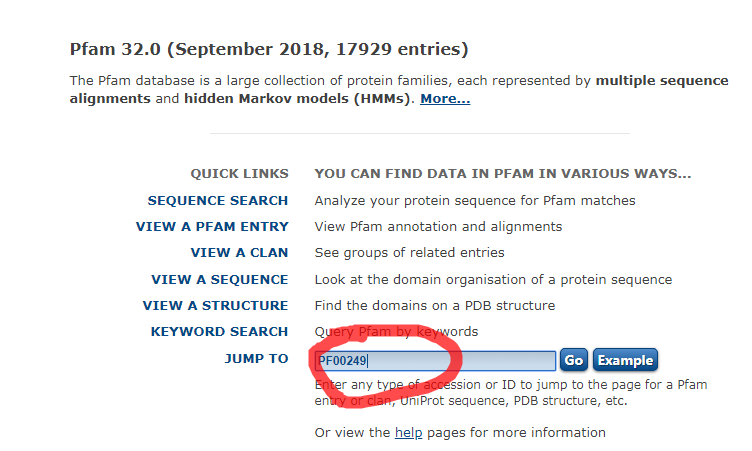
4.2进入这个,点击这
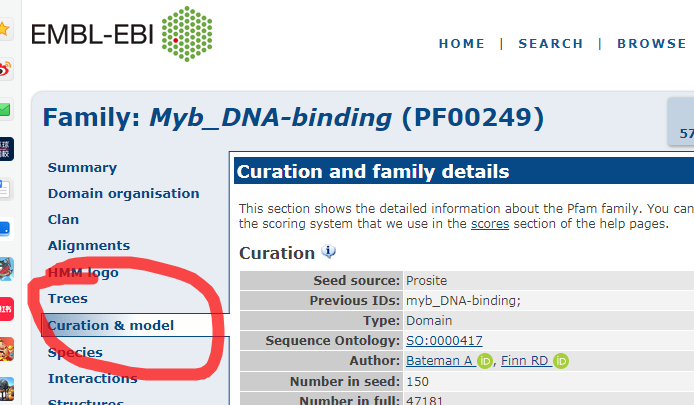
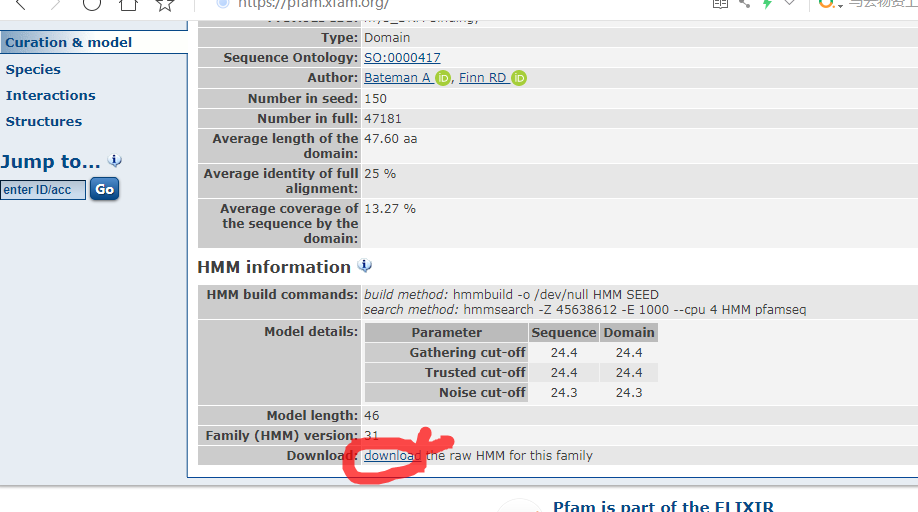
4.3 这个页面最下面有download,就下载好了
5,利用hmmer对蛋白进行鉴定
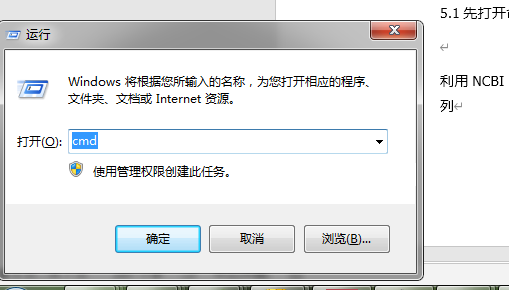
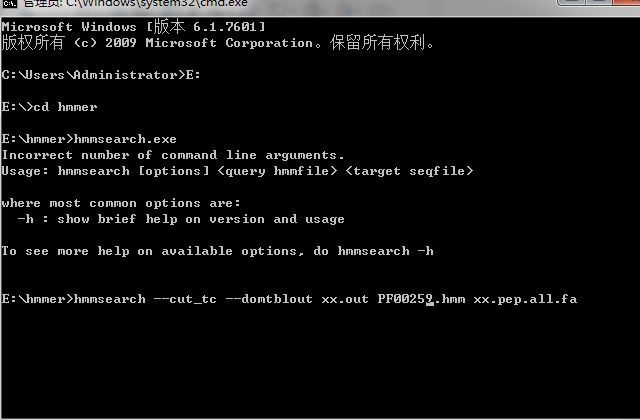
5.1先打开命令窗口,win键(就是键盘上的田字键)+R
5.2 输入命令e:
再输入命令 cd hmmer
再输入hmmsearch.exe
再输入
hmmsearch –cut_tc –domtblout xx.out PF00249.hmm xx.pep.all.fa
5.3对得到的out数据质控,使用Editplus打开xx.out文件
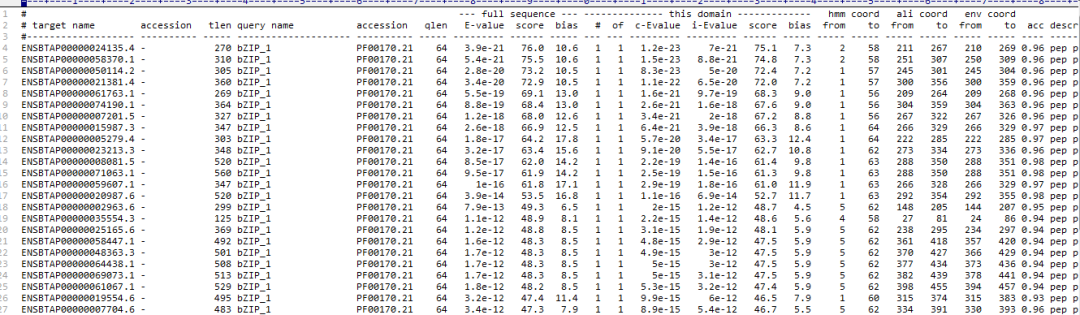
5.4根据e值1e-20,对数据进行质控,然后的可信序列的id,然后复制下来,可用excel做,也可以直接肉眼观察
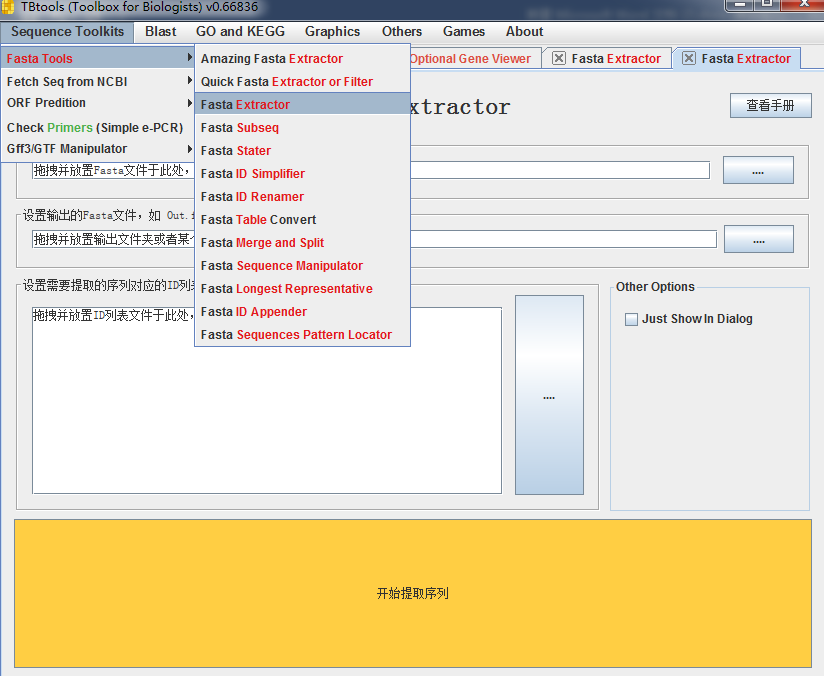
5.5 使用tbtools,提取蛋白质序列,
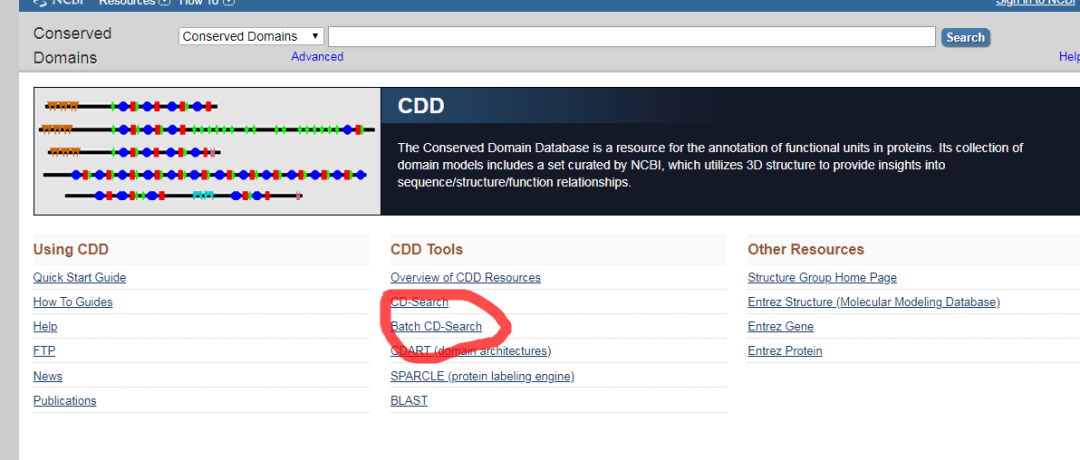
6,利用NCBI CDD和smart数据库对保守结构域进行鉴定,最终确定保留那些序列
6.1打开ncbi,选择conserved Domain
6.2 两个都行,
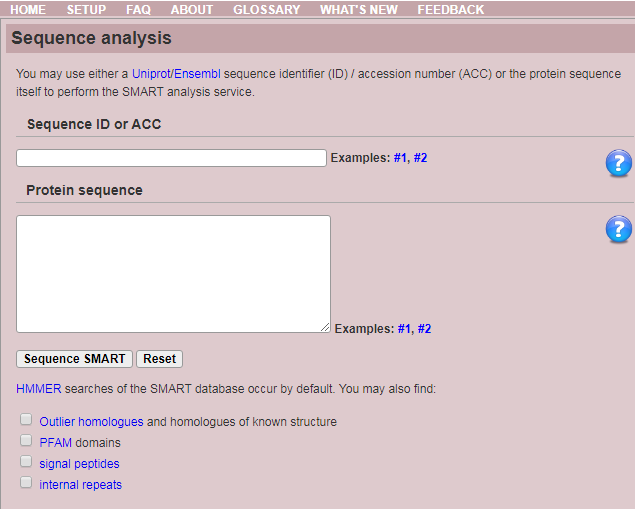
6.3 smart数据库,与CDD类似
http://smart.embl-heidelberg.de/smart/set_mode.cgi?GENOMIC=1#
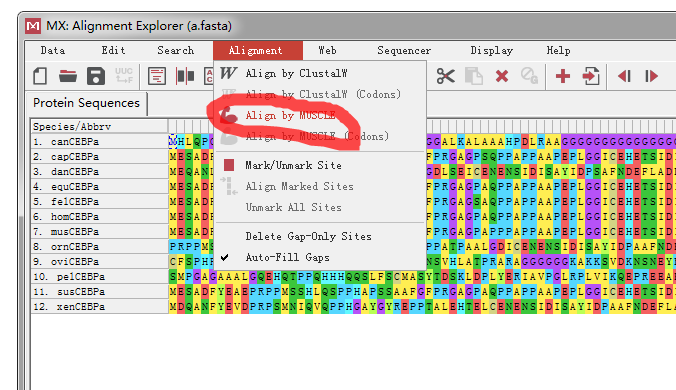
7,利用软件MEGA X画NJ进化树,
7.1打开MEGA X,打上传要分析的蛋白文件

7.2会进入这个页面,点击这儿,之后再点击OK
7.3 结果分析ok后,输出文件,

7.4将这个文件包存后,把表叉掉,
再次输入上面的已经保存的结果文件。
7.5构建进化树
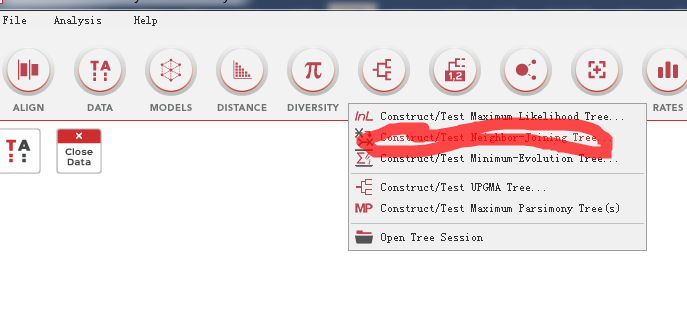
7.6参数设置
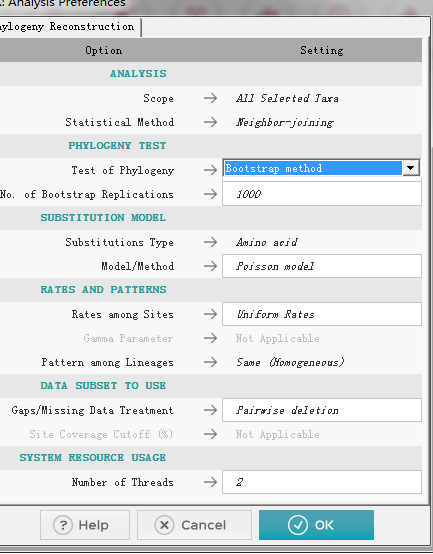
7.7 等待结果,电脑不能关机呦
以上就是目前基因家族进化分析的大致介绍,其实每个环节背后都有很多新知识,也不可能一两篇科普就能讲清楚的。有兴趣的朋友可以继续关注小果的微信公众号(生信果)和云生信生物信息学平台:
http://www.biocloudservice.com/home.html
小果也是边学习边理解边交流边工作。有疑惑可以询问小果,欢迎大家彼此多多交流学习。
小果还提供思路设计、定制生信分析、文献思路复现;有需要的小伙伴欢迎直接扫码咨询小果,竭诚为您的科研助力!

定制生信分析
服务器租赁
扫码咨询小果


往期回顾
|
01 |
|
02 |
|
03 |
|
04 |