小果带你复现多样数据可视化!轻松拿下IF30.8文献组合图:柱状图+饼图+线型图!!

在当今信息时代,数据可视化成为解读复杂数据的重要工具,这次小果将带小伙伴一同探索多样化的数据可视化,通过简单的R代码轻松复现一篇IF30.8高分文献中的令人赏心悦目的组合图:柱状图+饼图+线型图。我们先来看我们这次复现的内容,是来自IF30.8《Pangenome analysis provides insight into the evolution of the orange subfamily and a key gene for citric acid accumulation in citrus fruits》中的图3a和b
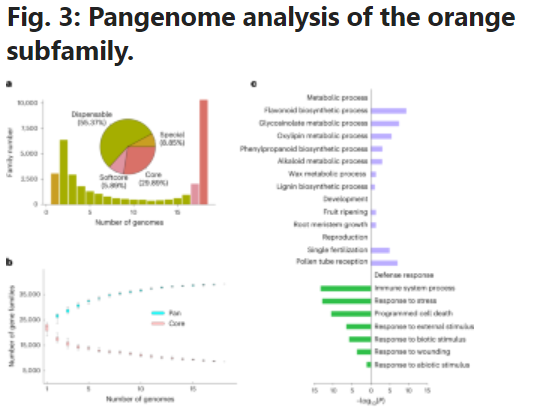
其实,数据可视化是解读和交流数据的关键工具,而R语言以其强大的绘图能力成为科学家们的首选。在这个图中,我们将聚焦文献中的组合图,这个图包括柱状图、饼图和线型图。这篇文献以其卓越的质量和独特的数据呈现方式获得高度评价。所以,跟随“小果”的指引,将带小伙伴亲身体验如何用简单的代码在R中实现这些引人入胜的可视化效果。
柱状图是展示各类别之间关系的经典工具,而饼图则以清晰的部分整体比例为我们呈现数据的构成。线型图则适用于描绘趋势和变化。这三者的巧妙结合使得我们能够全面了解文献中所探讨的数据背后的故事。
这里呢小果还要说明一下,我们这次做的数据分析数据量是非常大的,我们操作占用内存比较大,所以建议各位小伙伴可以使用服务器去运行,如果没有自己的服务器也没有关系,可以欢迎联系我们使用服务器租赁~
公众号后台回复“111”
领取本篇代码、基因集或示例数据等文件
文件编号:240130

library(tidyverse)#这里我们还有加载一个包library(readxl)#将 Excel 文件(.xls 和 .xlsx)读取到 R 中而设计的 dat1<-read_excel("data1.xlsx", sheet = "Sheet1")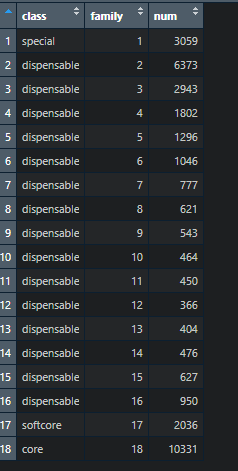
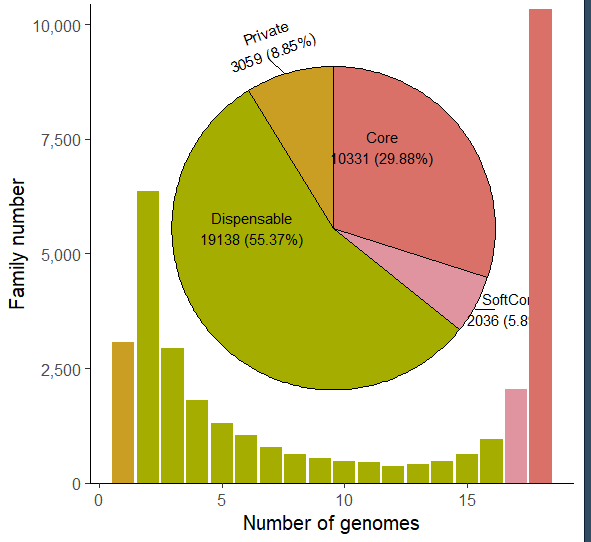
p1<-ggplot(data=dat1,aes(x=family,y=num))+geom_col(aes(fill=class),show.legend = FALSE)+# # 基于“class”进行颜色填充条形图theme_bw(base_size = 15)+##将主题设置为黑白,基本字体大小为 15theme(panel.grid = element_blank(),# 删除网格线panel.border = element_blank(),# 删除面板边框axis.line = element_line())+# 删除轴线scale_y_continuous(expand = expansion(mult=c(0,0.01)),# 稍微扩大 y 轴范围breaks = seq(0,10000,2500),# 设置 y 轴断点labels = scales::comma)+# 用逗号格式化 y 轴标签scale_x_continuous(breaks = c(0,5,10,15))+# 设置 x 轴中断labs(x="Number of genomes",y="Family number")+# 设置轴标签scale_fill_manual(values = c("special"="#c99e22",# 设置'class'的手动填充颜色"dispensable"="#a5ae00","softcore"="#df94a0","core"="#d97168"))p1
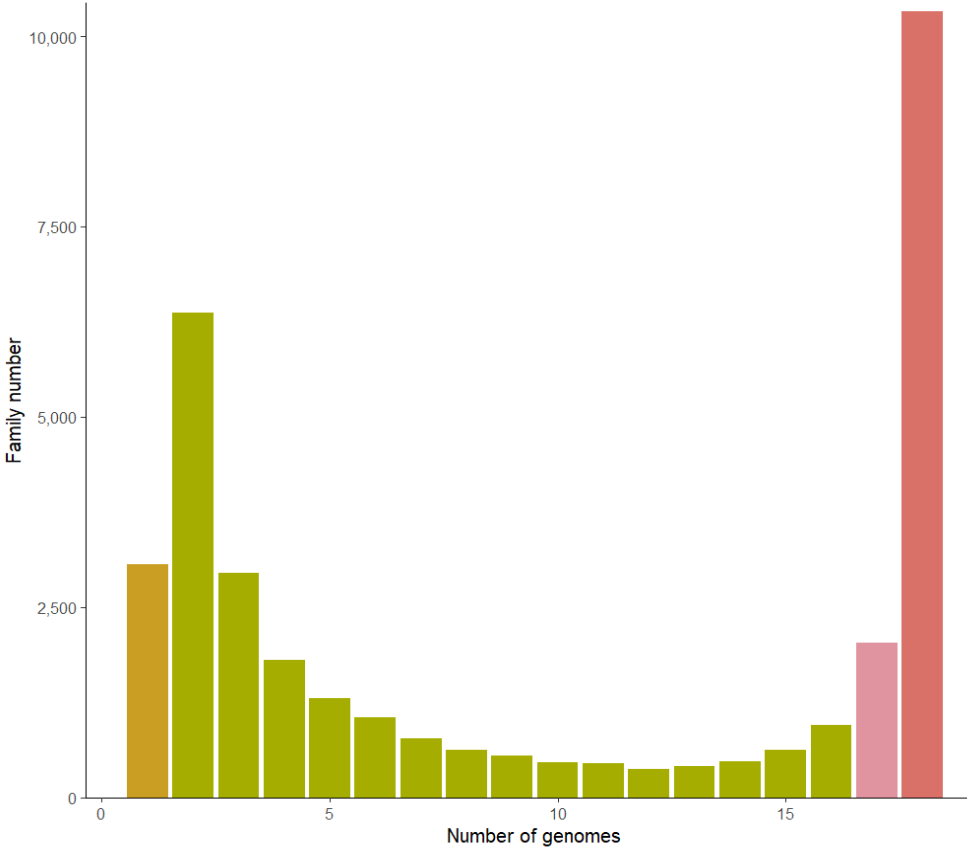
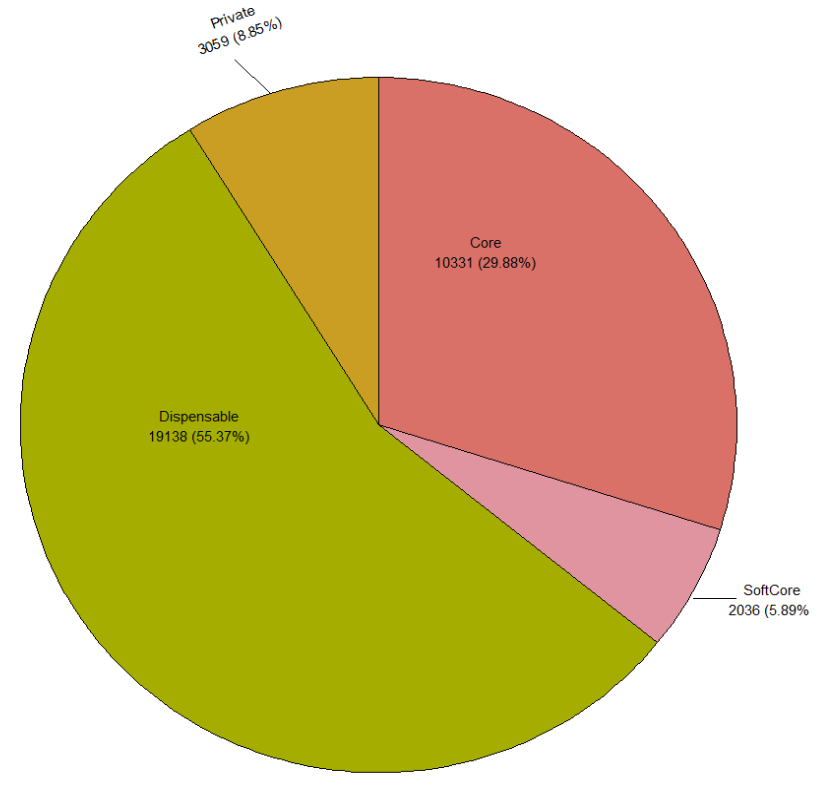
library(scatterpie)#创建带有饼图标记的散点图的包然后小果创建了一个名#为dat2“x”、“y”、“region”、“Core”、“Softcore”、“Dispensable”和“Special”列的数据框=1,y=1,region=1,Core=10331,Softcore=2036,Dispensable=19138,Special=3059)
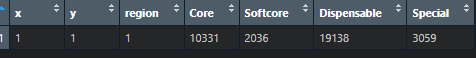
p2<-ggplot()+#初始化一个新的 ggplot 对象#将带有饼图标记的散点图添加到 ggplot 中geom_scatterpie(data=dat2,#指定数据框dat2aes(x,y,group=region,r=1),#定义美观的x 和 y 坐标、按“区域”分组以及饼图标记的固定半径 1cols=c("Core","Softcore","Dispensable","Special"),#指定用作饼图类别的列color="black",#将饼图边框的颜色设置为黑色show.legend=FALSE)+#隐藏图例coord_equal()+#确保 x 轴和 y 轴的缩放比例相等theme_void(base_size = 15)+#将主题设置为空(无背景、轴线或标签),基本字体大小为 15#向绘图添加文本注释,参数设置x和y坐标、标签文本和文本旋转角度annotate(geom = "text",x=1.3,y=1.5,label="Coren10331 (29.88%)",angle=0)+annotate(geom = "text",x=2.1,y=0.5,label="SoftCoren2036 (5.89%)",angle=0)+annotate(geom = "text",x=0.5,y=1,label="Dispensablen19138 (55.37%)",angle=0)+annotate(geom = "text",x=0.6,y=2.15,label="Privaten3059 (8.85%)",angle=20)+#向绘图添加线段,参数包括每条线段的起始和结束坐标annotate(geom = "segment",x=0.7,xend=0.6,y=1.95,yend=2.05)+annotate(geom = "segment",x=1.88,xend=2.0,y=0.5,yend=0.5)+#动设置不同饼图类别的填充颜色scale_fill_manual(values = c("Special"="#c99e22","Dispensable"="#a5ae00","Softcore"="#df94a0","Core"="#d97168"))p2
#组合library(ggpmisc)然后我们就进行绘制p1+##指定新子图的数据,其中“x”和“y”是坐标,“plot”是包含第二个图的列表 ( p2)geom_plot(data=tibble(x=2,y=10000,plot=list(p2)),aes(x=x,y=y,label=plot),#定义子图的美观,x 和 y 坐标以及代表第二个图的标签vp.width=0.8,vp.height=0.8)#设置子图视口的宽度和高度
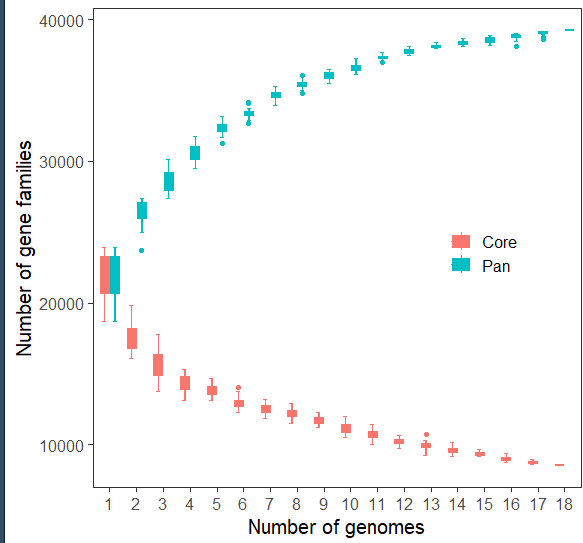
#线型图dat3<-read_excel("data2.xlsx",sheet = "Sheet1") %>%pivot_longer(!`Number of genomes`) %>%#用mutate函数将“基因组数量”列转换为具有指定级别的因子mutate(`Number of genomes`=factor(`Number of genomes`,levels = 1:18))
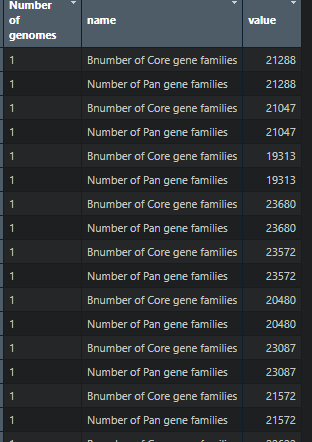
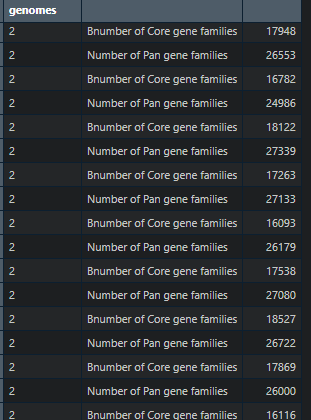
ggplot(data=dat3,aes(x=`Number of genomes`,y=value,fill=name,color=name))+stat_boxplot(geom="errorbar",width=0.3,position = position_dodge(0.8))+geom_boxplot()+theme_bw(base_size = 15)+theme(legend.position = c(0.8,0.5),legend.background = element_rect(fill="transparent"),panel.grid = element_blank())+labs(x="Number of genomes",y="Number of gene families")+scale_fill_discrete(name=NULL,labels=c("Core","Pan"))+scale_color_discrete(name=NULL,labels=c("Core","Pan"))
小果还提供思路设计、定制生信分析、文献思路复现;有需要的小伙伴欢迎直接扫码咨询小果,竭诚为您的科研助力!

定制生信分析
服务器租赁
扫码咨询小果


往期回顾
|
01 |
|
02 |
|
03 |
|
04 |