「付费」【代码-8】分析细胞代谢活性,有scMetabolism就够了!
收录于话题
今天小果教大家如何使用scMetabolism包来计算单细胞数据的细胞代谢活性以及可视化操作,感兴趣的话就和小果一起看下去吧!
什么是scMetabolism?
scMetabolism是一个用于单细胞代谢组学数据分析的R语言包。该包基于单细胞转录组学数据和代谢组学数据,并使用了机器学习算法来预测代谢网络中的代谢物相互作用。
scMetabolism包可以用于以下几个方面:
1.代谢通路分析:通过分析单细胞代谢组学数据,可以预测代谢通路的活性和代谢产物的积累情况,从而揭示细胞代谢网络的结构和功能。
2.代谢物互作分析:通过预测代谢物的相互作用,可以揭示代谢物之间的调节关系,为进一步研究细胞代谢调节提供基础。
3.细胞类型鉴定:通过比较不同细胞类型之间的代谢物组成,可以鉴定不同细胞类型的代谢特征,为研究细胞分化和发育提供基础。
总之,scMetabolism包为单细胞代谢组学数据分析提供了一个强大的工具,可以揭示细胞代谢网络的结构和功能,为研究代谢调节和细胞生物学提供了新的思路和方法。
具体的安装和使用方法,就和小果一起看下去吧!
下载分析所需要的所有R包
install.packages(c("devtools", "data.table", "wesanderson", "Seurat", "devtools", "AUCell", "GSEABase", "GSVA", "ggplot2","rsvd"))devtools::install_github("YosefLab/VISION")devtools::install_github("wu-yc/scMetabolism")
下载过程中可能遇到的问题:
1.网络问题:
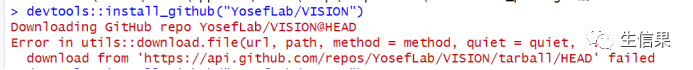
这是由于网络不佳、没有代理服务器等多种可能的因素造成的,如果你在下载时也遇到相同的问题,可以尝试改为手动下载再导入到R环境中:
手动下载VISION包的链接https://codeload.github.com/YosefLab/VISION/legacy.tar.gz/HEAD
下载好后,导入到R中:
install.packages("E:/R-packages/YosefLab-VISION-v3.0.1-0-g8dc5c4e.tar.gz", repos = NULL, type="source")此时又会弹出报错信息:
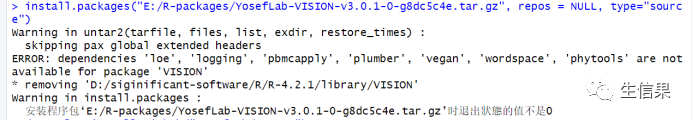
这是在提醒我们,安装VISION包之前,还需要导入loe、logging等R包,所以我们先下载这些需要导入的R包:
install.packages(c("loe", "logging", "pbmcapply", "plumber", "vegan", "wordspace", "phytools"))安装好后,就可以顺利安装VISION包啦!
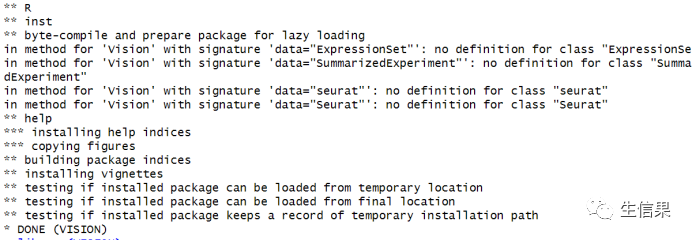
现在你已经完成了所有R包的下载与安装,接下来让我们进入今天的正题吧!
结果文件
如果需要完整的代码可以点击付费获取哦!今天小果的分享就到这里,如果小伙伴有其他数据分析需求,可以尝试本公司新开发的生信分析小工具云平台,季代码完成分析,非常方便奥!
往期推荐

(扫码领取整理好的输入文件,代码文件及示例结果)