噬菌体只是微生物中的小打小闹?神秘工具PhaGCN_newICTV,带你探索噬菌体分类的“黄金法则”!

公众号后台回复“111”
领取本篇代码、基因集或示例数据等文件
文件编号:240402
需要租赁服务器的小伙伴可以扫码添加小果,此外小果还提供生信分析,思路设计,文献复现等,有需要的小伙伴欢迎来撩~


gh repo clone KennthShang/PhaGCN_newICTV# 使用github官方下载工具gh下载。
# 直接下载.zip安装包到服务器。随后使用unzip工具进行解压,没有这个工具的同学可以使用conda进行安装。unzip main # 解压下载的文件到当前目录。ls # 显示当前目录文件

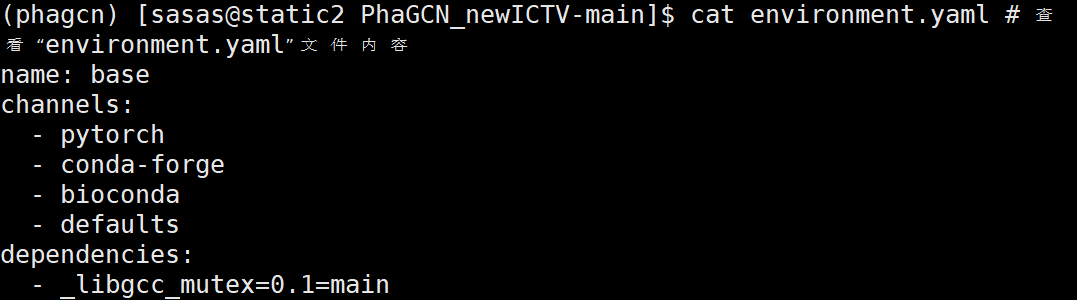
cd PhaGCN_newICTV-main # 进入文件夹ls # 显示当前目录文件至此我们已经成功下载PhaGCN_newICTV源文件,并且进入了源文件所在目录。配置PhaGCN_newICTV环境:cat environment.yaml # 查看“environment.yaml”文件内容
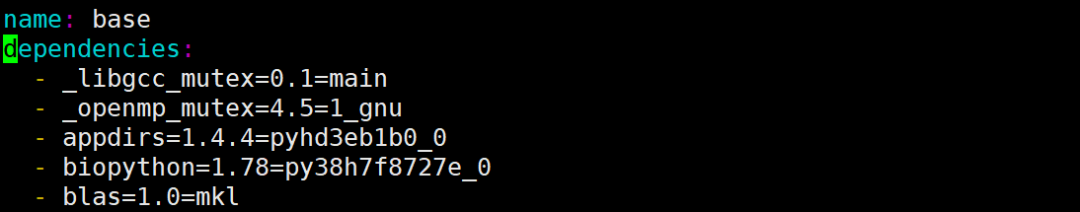
vim environment.yaml # 使用vim编辑器打开environment.yaml文件
conda env create -f environment.yaml -n phagcn # 按照配置文件创建conda环境
conda activate phagcn # 激活环境



小果还提供思路设计、定制生信分析、文献思路复现;有需要的小伙伴欢迎直接扫码咨询小果,竭诚为您的科研助力!

定制生信分析
服务器租赁
扫码咨询小果

往期回顾
|
01 |
|
02 |
|
03 |
|
04 |