「付费」【代码分享-20】更适合临床分析的机器学习算法-KNN算法和stepCox!小果手把手教你学会!
 小伙伴最近学习了不少生信分析,有的小伙伴有许多的困惑,小伙伴通常做完筛选基因后,发现基因有点多,而继续往后做却由于太多基因无从下手,一般的我们会在挑选对阈值进行缩小,或者在筛选一轮,小果这里有个好办法使用stepCOX,一种机器学习的方法,不过需要样本的临床的信息,不过这种方法更适合筛选完基因将要做预后分析的,这次小果带给小伙伴的方法可以有效的解决多个基因后续的分析,但是也适合与,面临预后分析适合,多余一些变量的筛选,比如不同分期情况下的基因变量如何,还以一种就是KNN算法,小果觉得这种算法适合于多个基因用于不同分型的变量情况下,来看看如何学习的吧!
小伙伴最近学习了不少生信分析,有的小伙伴有许多的困惑,小伙伴通常做完筛选基因后,发现基因有点多,而继续往后做却由于太多基因无从下手,一般的我们会在挑选对阈值进行缩小,或者在筛选一轮,小果这里有个好办法使用stepCOX,一种机器学习的方法,不过需要样本的临床的信息,不过这种方法更适合筛选完基因将要做预后分析的,这次小果带给小伙伴的方法可以有效的解决多个基因后续的分析,但是也适合与,面临预后分析适合,多余一些变量的筛选,比如不同分期情况下的基因变量如何,还以一种就是KNN算法,小果觉得这种算法适合于多个基因用于不同分型的变量情况下,来看看如何学习的吧!
我们从第一种方法开始,KNN算法,先简单了解一下这种算法
其实KNN(K-Nearest Neighbor)算法是机器学习算法中最基础、最简单的算法之一。它既能用于分类,也能用于回归。
这种KNN算法通过测量不同特征值之间的距离来进行分类。
简单来说KNN算法的思想非常简单:对于任意n维输入向量,分别对应于特征空间中的一个点,输出为该特征向量所对应的类别标签或预测值。
所以说这种算法适合于临床分析,因为有的数据会有临床分型的情况
看不懂的小伙伴也没关系,小果带你用代码实例去学习,这样更容易理解!
我们先载入两个包
library(mlr)library(tidyverse)
然后载入这个数据集,这里使用小果平时做过的实例,小伙伴可以按照数据形式自行去设置
a<-read.csv("a.csv",row.names = 1)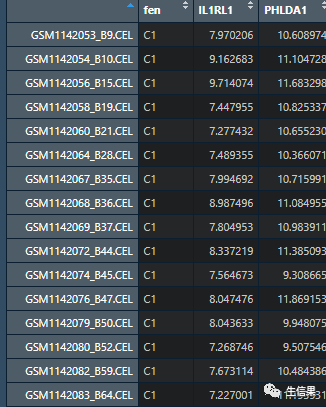
这个就需要小伙伴拥有数据样本的分组情况
a2<-read.csv("a2.csv",row.names = 1)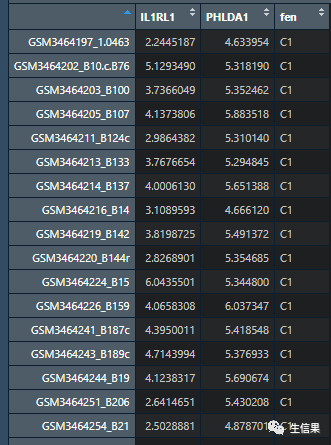
data<-read.csv("k.csv",row.names = 1)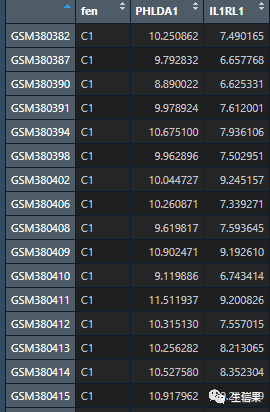
查看下运行的结果
kFoldCV$aggr
kFoldCV$measures.test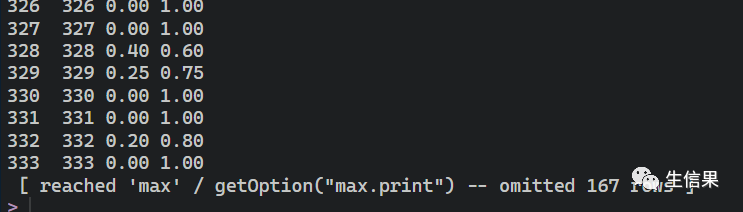
calculateConfusionMatrix(kFoldCV$pred, relative = TRUE)
我们仍需要调整一下K值
# K的超参数调整----knnParamSpace <- makeParamSet(makeDiscreteParam("k", values = 1:10))gridSearch <- makeTuneControlGrid()cvForTuning <- makeResampleDesc("RepCV", folds = 10, reps = 20)tunedK <- tuneParams("classif.knn", task = diabetesTask,resampling = cvForTuning,par.set = knnParamSpace,control = gridSearch)
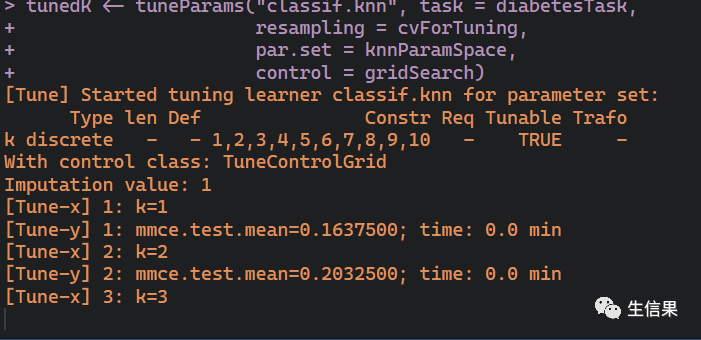
tunedK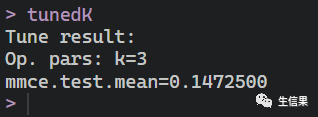
tunedK$x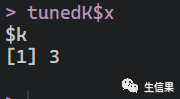
最优 的K值为3
knnTuningData <- generateHyperParsEffectData(tunedK)plotHyperParsEffect(knnTuningData, x = "k", y = "mmce.test.mean",plot.type = "line") +theme_bw()
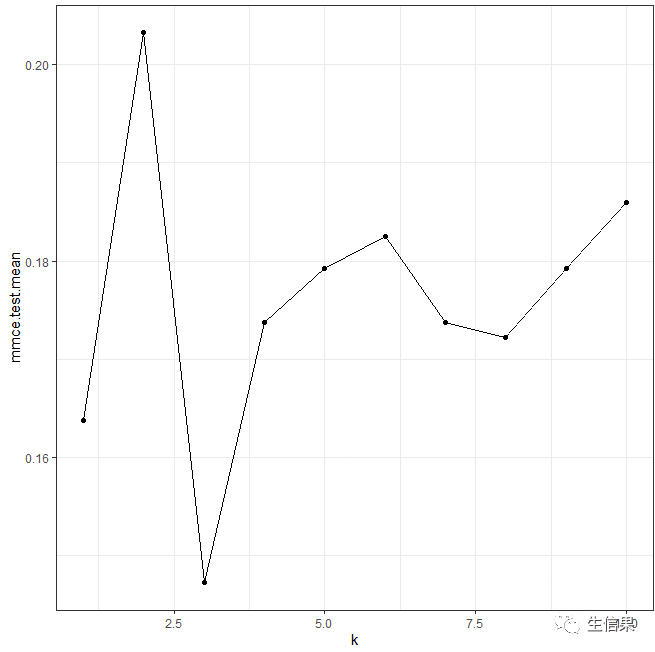
查看一下结果图,在K=3的适合最均衡,
然后使用试验去验证模型
# 用TUNED K训练最终模型----tunedKnn <- setHyperPars(makeLearner("classif.knn"), par.vals = tunedK$x)tunedKnnModel <- train(tunedKnn, diabetesTask)
继续调整参数
# 包括嵌套交叉验证中的超参数调整----inner <- makeResampleDesc("CV")outer <- makeResampleDesc("RepCV", folds = 10, reps = 5)knnWrapper <- makeTuneWrapper("classif.knn", resampling = inner,par.set = knnParamSpace,control = gridSearch)cvWithTuning <- resample(knnWrapper, diabetesTask, resampling = outer)
这里小伙伴多等待一下
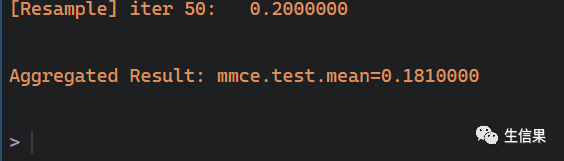
接下来
# 使用模型进行预测----newDiabetesPatients <- anewDiabetesPatients<-newDiabetesPatients[,-1]newPatientsPred <- predict(tunedKnnModel, newdata = newDiabetesPatients)library(caret)pred<-getPredictionResponse(newPatientsPred)confusionMatrix(table(pred,a$fen))
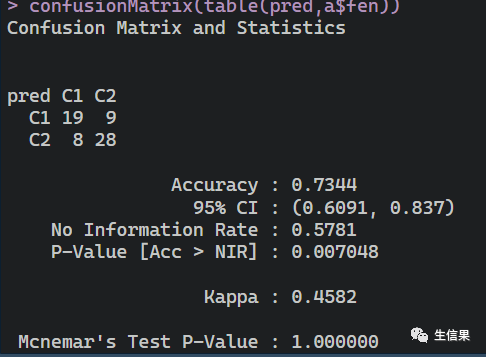
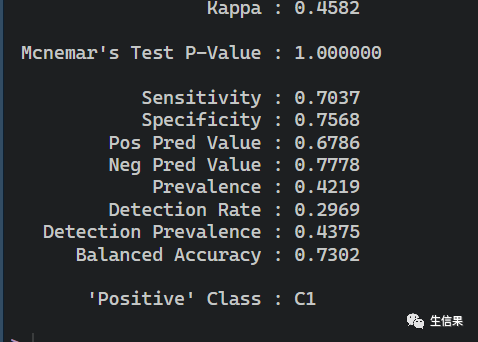
不同的模型去预测临床的信息
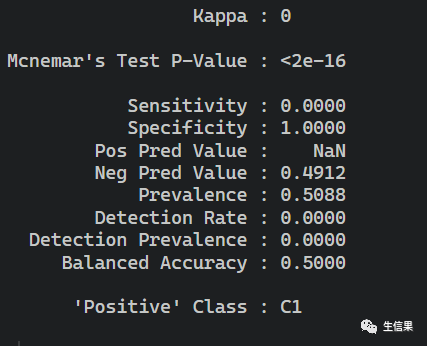
这样一来通过不同的临床分型,构建的模型就计算完成了,
下面我们学习一下setpcox计算
这一步骤其实就是 两个函数的连用,分别是step函数以及cox函数,简单理解就是通过step函数基于AIC选择一个cox模型,而这个cox模型需要满足AIC最小,那么自然在构建不同的cox模型的时候会选择不同的变量进而获得不同的AIC,所以就获得了变量筛选的能力,那么下面我们来看一下其代码是如何实现的
这里小果用实例数据集去分析,小伙伴可以更具数据集,自行去设置
a<-read.csv("data.csv",row.names = 1)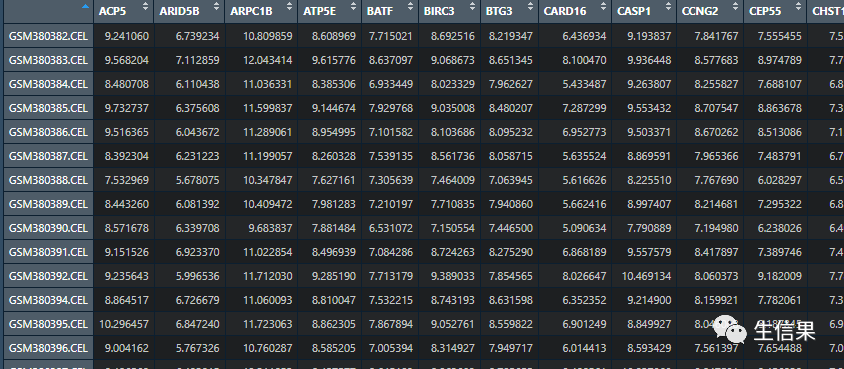
yu<-read.csv("预后信息.csv",row.names = 1)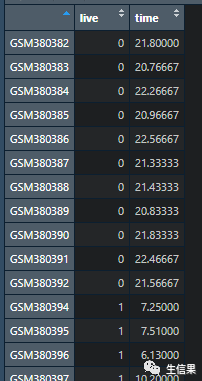
这里还是需要样本的临床信息的
其实这个方法计算很简单,只需要几步,
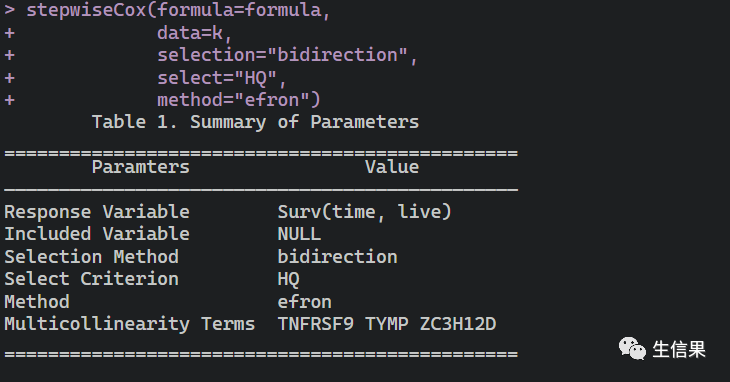
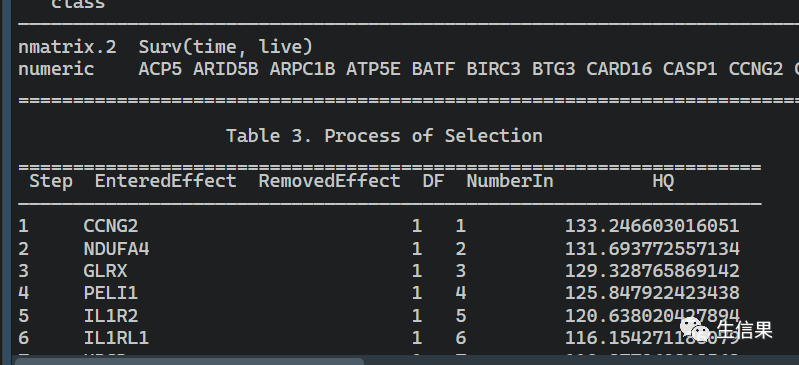
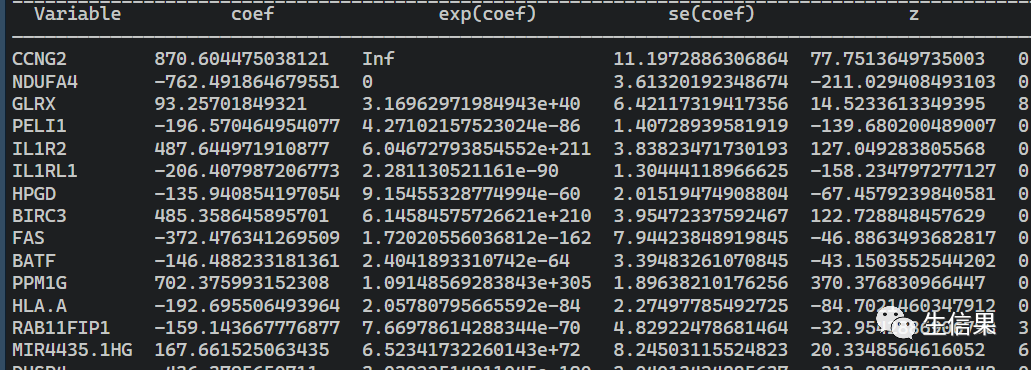
这些结果是很多的,小伙伴可以自行去保存,而且一些基因的预后风险值也计算出来,
好了这次的学习就结束了,小伙伴要亲手去试验哦,才能理解每一步的含义!

往期推荐
如果需要完整的代码可以点击付费获取哦!今天小果的分享就到这里,如果小伙伴有其他数据分析需求,可以尝试本公司新开发的生信分析小工具云平台,季代码完成分析,非常方便奥!
(扫码领取正好的输入文件,代码文件及示例结果)