一分钟,教你用pymol计算配体的RMSD!
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

今天我们来学习一下配体小分子的RMSD如何计算查看。
在动力学模拟分析中,很基本的一项分析就是RMSD值的求算。RMSD值即均方根偏差(Root Mean Square Deviation)。在统计学上,这个量就相当于标准差,反映的是数据偏离平均值的程度。在蛋白质结构解析,模建,结构联配(structure alignment)以及分子动力学模拟中,RMSD值是非常常用的一项参数,用于衡量原子偏离比对位置的程度。
今天我们来学习一下配体小分子的RMSD如何计算查看
1.首先我们使用pymol,选择一次对接结果中受体和配体的pdbqt文件,分别打开。File->open

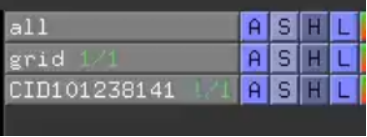
2.然后我们在命令栏输入align grid,CID101238141按回车
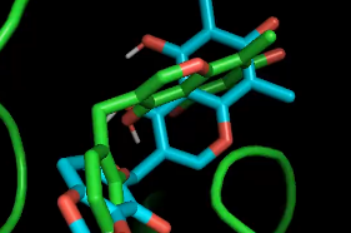
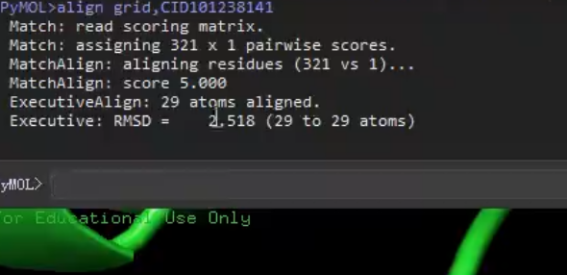
显示有29个原子atoms被匹配了,计算的RMSD值为2.518A。
小果还可以带你使用python中命令行计算RMSD
import pymolfrom pymol import cmd ##导入相应的包pymol.finish_launching() ##打开Pymol软件cmd.load("protein.pdb") ##加载蛋白质结构文件cmd.load("ligand.pdb", "ligand") ##配体结构加载到Pymol中cmd.select("ligand_selection", "ligand") ##将Pymol的选择焦点设置为配体。cmd.load("reference.pdb", "reference") ##选择并加载参考结构(可以是另一个配体结构或蛋白质结构)rmsd = cmd.align("ligand_selection", "reference_selection") ##执行RMSD计算print("RMSD:", rmsd)
这将计算配体与参考结构之间的RMSD,并将结果打印出来。
加载参考结构是为了计算配体与参考结构之间的RMSD。RMSD是一种用于衡量两个结构之间的结构相似性的指标,常用于比较蛋白质结构或配体结构的相似性。通过加载参考结构,您可以将其作为一个固定的结构,将配体结构与其进行比较。计算得到的RMSD值表示配体结构与参考结构之间的均方根偏差,即它们之间的结构差异程度。较低的RMSD值表示结构之间更相似,而较高的RMSD值表示结构差异较大。
这里推荐一下小果新开发的零代码云生信分析工具平台包含超多零代码小工具,上传数据一键出图,感兴趣的小伙伴欢迎了解~
网址:http://www.biocloudservice.com/home.html
今天小果的分享就到这里,欢迎大家和小果一起讨论学习,下期再见哦!
小果友情推荐
好用又免费的工具安利


