Volcano plot——别再问我这为什么是火山图
点击蓝字
关注小图
今天小图生信餐馆给小伙伴推荐道生信餐,能量轻餐:“火山包”
能量源于真材实料
火山图(Volcano plot)是一种比较“远古”的一种散点图,广泛应用于基因组、转录组、蛋白组等组间差异分析结果的展示。图表的横轴一般展示差异倍数的变化,而纵轴表示差异分析结果的可靠性。通过找到显著差异的基因,研究者可以进一步做后续的实验。
在生物信息分析中,火山图是非常常见的一种数据展示形式。由于火山图可以非常清晰的展示出哪些基因在不同样本中是具有差异表达显著性的基因,因此在生物医学中,常应用于病例和对照组的转录组研究,也能应用于基因组,蛋白质组,代谢组等统计数据。
轻食菜单
常见火山图类型:
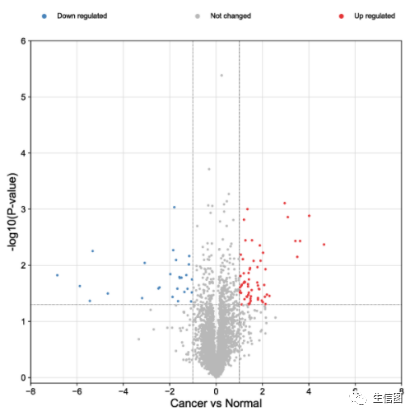
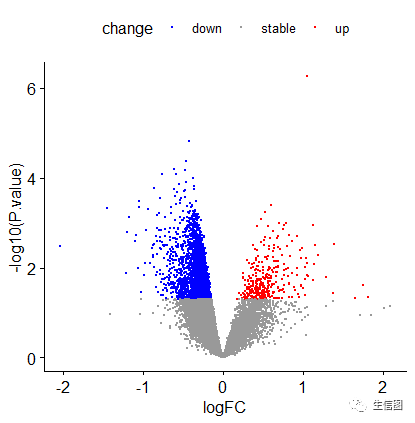
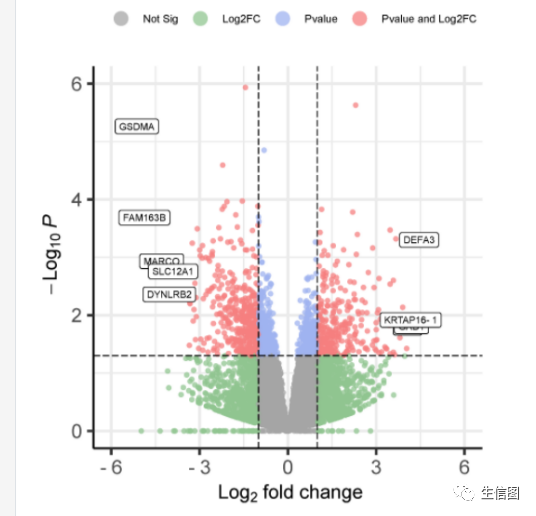
-
每个点代表一个检测到的基因。
-
横轴和纵轴用于固定点在空间的位置。
-
一般横轴是Log2(fold change),点越偏离中心,表示差异倍数越大。
-
纵轴是-Log 10 (pvalue),点越靠图的顶部表示差异越显著。
-
点的大小和颜色也可以表示更多的属性,如后面图中点的颜色标记其对应的基因是上调, 下调还是无差异。
-
大小也可用于展示基因表达的平均丰度,一般我们关注表达水平较高且差异较大的基因用于后续的分析和验证。
火山图是许多生信平台首批上线的常见图,虽然我们经过了大量优化,增加了诸多可调参数:颜色,点大小,甚至legend的位置和顺序都进行了优化,但是,还是不能满足小伙伴的需求:
能不能标注我感兴趣的基因?
能不能给点加上透明度?
能不能额外加些线?
能不能放大某些点?
能不能把p值为0的点阈值宽松点?
能不能标注下上调多少,下调多少?
小图可以负责任地告诉小伙伴,都能满足,But,任何在线工具都不能满足您的定制需求。要想满足,需要你自己编程或者编辑生成的图。
今天,给小伙伴带来的制作材料是RColorBrewer、ggrepel、ggplot R包。这里小图给不懂编程的小伙伴带来不一样的火山图体验。走起————
制作过程
rm(list = ls())#清除所有的变量library(RColorBrewer)library(ggrepel) #标签用
接下来准备作图的数据
今天用到的geo数据是经limma差异分析后的,数据的格式是文本text,可直接导入到R中:
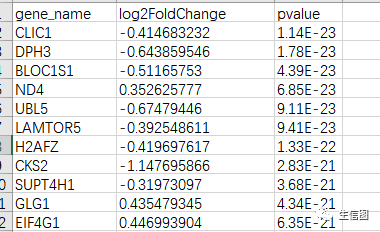
setwd('D:\limma')#加载路径df=read.table("火山图输入数据.txt",sep='t',header = T)#确定是上调还是下调,用于给图中点上色df$threshold = factor(ifelse(df$pvalue < 0.05 & abs(df$log2FoldChange) >= 0.25, ifelse(df$log2FoldChange >= 0.25 ,'Up','Down'),'NoSignifi'),levels=c('Up','Down','NoSignifi'))df$gene <- row.names(df) #添加一列基因名,以便备注p <- ggplot(df,aes(x=log2FoldChange,y= -log10(pvalue),color=threshold))+geom_point(data = df[df$pvalue<0.05&abs(df$log2FoldChange)>0.25,],size = 0.5)+geom_point(data = df[df$pvalue>0.05|abs(df$log2FoldChange)<0.25,],size = 0.5)+scale_color_manual(values=c("#4393C3","#00000033","#FC4E2A"))+#确定点的颜色geom_text_repel(data = df[df$pvalue<0.05&abs(df$fd)>1,],aes(label = gene),size = 4.5,color = "black",segment.color = "black", show.legend = FALSE )+#添加关注的点的基因名ylab('-log10 (P_value)')+#修改y轴名称xlab('log2 (FoldChange)')+#修改x轴名称geom_vline(xintercept=c(-0.25,0.25),lty=3,col="black",lwd=0.5) +#添加横线|logFoldChange|>0.25geom_hline(yintercept = -log10(0.05),lty=3,col="black",lwd=0.5) +#添加竖线padj<0.05theme_classic( # 主题设置,这个是无线条主题base_line_size = 1 # 坐标轴的粗细)+theme(axis.title.x = element_text(size = 15,color = "black",face = "bold"),axis.title.y = element_text(size = 15,color = "black",face = "bold",vjust = 1.9,hjust = 0.5,angle = 90),legend.title = element_blank(),legend.text = element_text(color="black", # 设置图例标签文字size = 10,face = "bold"),axis.text.x = element_text(size = 13, # 修改X轴上字体大小,color = "black", # 颜色face = "bold",vjust = 0.5, # 位置hjust = 0.5,angle = 0), #角度axis.text.y = element_text(size = 13,color = "black",face = "bold",vjust = 0.5,hjust = 0.5,angle = 0))
上菜
ppdf(file="volcano_plot.pdf", width=11, height=9)#导出图片print(p)dev.off()
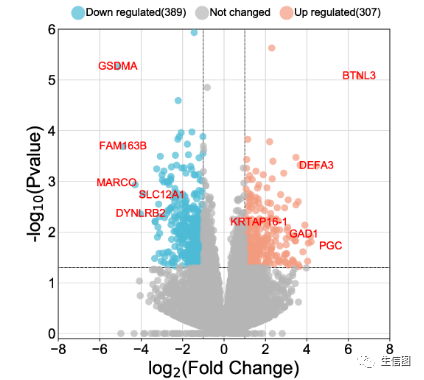
请享用
火山图的本质就是一张散点图,我们只要理清了绘图逻辑,无论用公共平台(云生信生物信息学平台:http://www.biocloudservice.com/home.html)还是和小图学(公众号:生信图),都可以绘制出漂亮的火山图!该程序还远远不止这些,例如文字重叠、连线交叉等等。小伙伴赶紧动手做起来,相信肯定绘制的图片比小图精美。
期待小图生信餐馆下次新品!
到此,GEO数据库芯片数据的下载,probe id的转换已经基本完成了,整个分析中的数据处理内容也已经基本解决。接下来,就是针对这些差异基因的常规分析,以及GO分析,KEGG分析,GSEA分析,蛋白-蛋白互作网络(Protein-protein interaction,PPI)。请期待小图的下期分享!如有分析问题或需求欢迎小伙伴们随时来和小图探讨。
欢迎使用:云生信平台 ( http://www.biocloudservice.com/home.html)

|
往期推荐 |
|
|
|
|
|
|