那些容易被人忽视的R包——基于caret的机器学习(一)
点击蓝字 关注我们
小伙伴们大家好,大海哥又来安利好东西啦!今天要推荐大家学习的是一个R包,这个R包基本可以实现我们生信分析中常用的大部分机器学习方法,并且还初始化了大部分参数,不再需要我们自己定义一大堆参数,省去了相当大的麻烦,听起来是不是觉得十分强大!是的,掌握了它,大海哥觉得屏幕前的你拿下几篇SCI不在话下!时间紧急,让我们先赶紧了解一下这个R包吧
caret(Classification And REgression Training的缩写)是一个在R语言中广泛使用的包,用于简化机器学习模型的训练、评估和预测过程。它为用户提供了一个统一的界面,可以在多种机器学习算法中进行选择、参数调整、交叉验证等操作,从而使整个模型开发流程更加高效和方便。大海哥大致统计了一下,其包含238种模型和众多函数,简直强到窒息~。不过目前,caret包已经停止更新,其主要作者已加入Rstudio开发了tidymodels,从tidymodels中我们还能看到caret的影子,但对于机器学习的初学者来说,caret是容易理解和学习的,所以大家一定不可以忽视这个R包哦!
caret包的功能包含下面几种
数据拆分
数据预处理
特征选择
模型构建及优化
变量重要性评估
其他函数部分
由于内容过多,所以本文主要介绍数据拆分、数据预处理和模型构建及优化,其余部分在后续文章中介绍。
1、数据预处理
1.1创建虚拟变量
library(caret)library(earth) #利用包内的数据data(etitanic)#使用baseR中的model.matrixhead(model.matrix(survived ~ ., data = etitanic)[,-1])
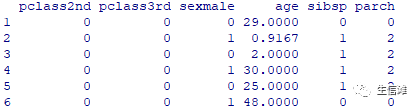
#数据集etitantic中pclass变量有1st,2nd, 3rd三个水平,需要转换为虚拟变量#model.matrix()函数将pclass转换为三个变量并把第一个水平作为参考,同时生成一个常数列#使用caret包的dummyVars()函数dummies <- dummyVars(survived ~ ., data = etitanic)head(predict(dummies, newdata = etitanic))
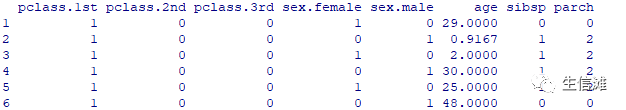
可以看到函数将pclass转换为三个单独的变量
1.2数据预处理
#preprocess函数#大海哥先介绍一下它的用法preProcess(x, 需要处理的数据集,数据集为数值型的矩阵或数据框method = c("center","scale"), 数据处理方#thresh = 0.95, PCA的阈值pcaComp = NULL, PCA主成分个数na.remove = TRUE, 是否去除缺失值#k = 5, knn算法的k值knnSummary = mean, knn插值的方法outcome = NULL, 结局变量fudge = 0.2, 公差值numUnique = 3, Box-Cox变换需要多少个唯一值verbose = FALSE, 是否显示处理过程freqCut = 95/5, 最常见值与第二常见值的比值uniqueCut = 10, 不同值占总样本数的百分比cutoff = 0.9, 相关性的阈值rangeBounds = c(0, 1), 指定范围内变换数据预处理方法method还有:"BoxCox","YeoJohnson", "expoTrans", "center","scale", "range", "knnImpute","bagImpute", "medianImpute", "pca","ica", "spatialSign", "corr", "zv","nzv", and "conditionalX"#然后我们找个示例演示一下吧library(AppliedPredictiveModeling)data(schedulingData)#引入示例数据str(schedulingData)

pp <- preProcess(schedulingData[, -8],method = c("center", "scale", "YeoJohnson", "nzv")

#处理完成后,看看能不能拿来进行预测predict(pp, newdata =schedulingData[1:6, -8])
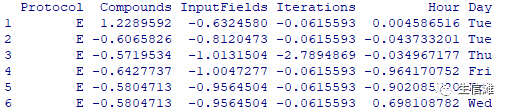
顺利进行哦!
2、数据拆分
还可以进行数据拆分data(iris)iris <- na.omit(iris)set.seed(123) # 设置随机种子,以确保结果可重复train_indices <- createDataPartition(iris $Species, p = 0.7, list = FALSE)train_data <-iris[train_indices, ]test_data <- iris[-train_indices, ]
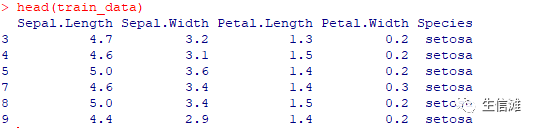
这样就可以实现3:7随机抽取测试集和训练集哦!很方便吧!
3、模型训练
#基本参数设定fitControl <- trainControl(## 10-fold CVmethod = "repeatedcv",number = 5,## repeated ten timesrepeats = 3)# method:重采样方法,包括"boot", "boot632", "optimism_boot","boot_all", "cv", "repeatedcv","LOOCV", "LGOCV" (for repeated training/test splits),"none" (only fits one model to the entire training set),"oob" (only for random forest, bagged trees, bagged earth, bagged flexible discriminant analysis, or conditional tree forest models), timeslice,"adaptive_cv", "adaptive_boot" or"adaptive_LGOCV"#number用于设定交叉验证和重复交叉验证的折数、bootstrap抽样次数#repeats 用于设定交叉验证的次数#模型拟合set.seed(1)gbmFit1 <- train(factor(Species) ~ .,data = train_data,method = "gbm",trControl = fitControl,## This last option is actually one## for gbm() that passes throughverbose = FALSE)
可以看到模型拟合并且构建成功!
4、模型调参
迭代次数,即树(在函数中调用)n.trees树的复杂性,称为interaction.depth学习速率:算法适应的速度,称为shrinkage节点中开始拆分的训练集样本的最小数量(n.minobsinnode)#定义调参过程gbmGrid <- expand.grid(interaction.depth = c(1, 5, 9),n.trees = (1:20)*100,shrinkage =0.1,n.minobsinnode = 20)nrow(gbmGrid)
#开始调参set.seed(2)gbmFit2 <- train(factor(Species) ~ .,data = train_data,method = "gbm",trControl = fitControl,verbose = FALSE,tuneGrid = gbmGrid)gbmFit2$bestTune #调优结果#这一步需要消耗一点点时间哦!

#可以看到,给出了调优结果gbmFit2$finalModel #最终模型

#我们还可以根据结果,绘制重采样曲线ggplot(gbmFit2) #以Accuracy为基准
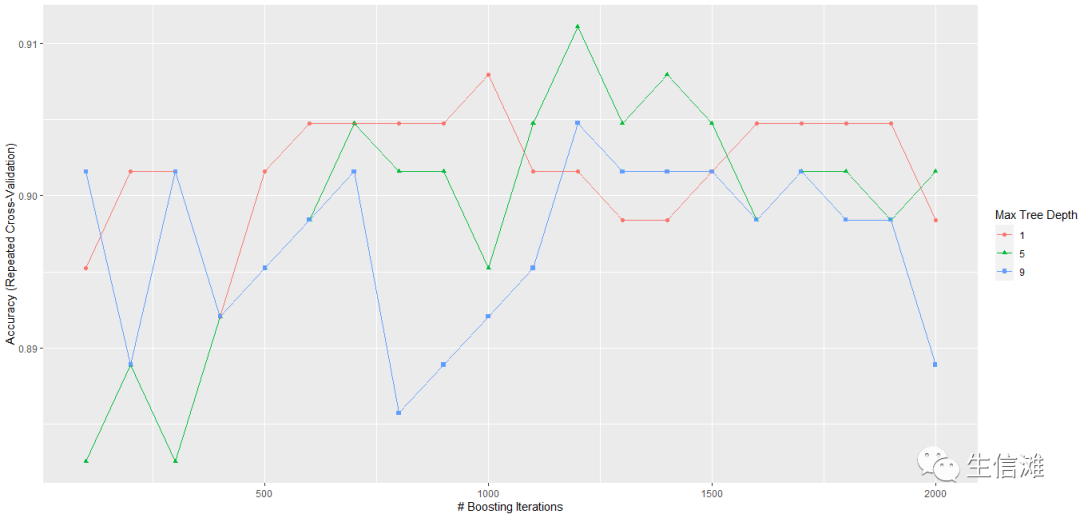
#也可以选择ggplot(gbmFit2,metric = "Kappa") #以Kappa为基准5、模型预测
#这一步也是我们最重要的一步了predict(gbmFit2, newdata = head(test_data))#默认预测分类

#预测成功啦!#还可以预测概率哦!predict(gbmFit2,newdata = head(test_data), type = "prob")#预测概率
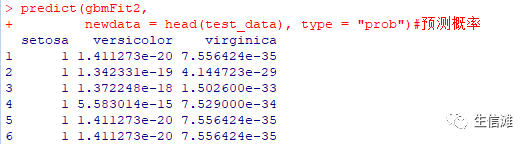
有了预测结果,大家就可以自由发挥啦!
着是不是也还不错
还可以基于ggfittext方法修改节点标签哦

点击“阅读原文”进入网址
