全村人的希望:多组学孟德尔随机化整合分析实战:从数据处理到生物学解释,揭示氧化应激与肠道菌群的奥秘!

孟德尔随机化将遗传变异作为自然随机化实验,结合多层面组学数据,这种创新性的研究方法为生信领域带来了全新的机遇。不仅能够更深入地探究疾病的潜在因果关系,还可以系统性地阐明关键调控网络和通路,为疾病发病机制的解析和新靶点的开发提供重要线索。
今天馆长跟小伙伴们分享一篇高分孟德尔随机化类文章。本篇文章的作者采用多组学整合的孟德尔随机化(SMR)分析方法,系统探讨OS基因在CD发病中的潜在因果关系及其调控机制。你也对这个方向感兴趣吗?那就快来和馆长一起学习探索吧,相信一定会有意想不到的收获!馆长这就带小伙伴们一起揭秘一下吧~
1、首先本研究采用多组学整合方法,研究CD致病基因OS受到DNA甲基化和宿主-微生物群相互作用的调控,这为未来针对性的功能研究提供了证据,用于开发合适的治疗干预措施和疾病预防。
2、本文章运用了大规模样本数据增加了研究结果的可靠性。
3、通过DNAm、基因表达以及与肠道菌群的相互作用,对候选OS基因进行了研究。我们新发现的致病OS基因与微生物类群和途径之间的宿主-微生物群相互作用值得在功能水平上进行研究,以更深入地了解潜在的生物学机制。这项研究推进了OS在CD中作用的基础研究,并为临床实践指出了潜在的新治疗靶点。
总的来说,这项研究充分利用多组学数据,首次系统阐明了OS基因在CD发病中的潜在因果关系及其可能的表观遗传和菌群互作机制,为后续靶向干预提供了重要线索。这种融合生信分析和实验验证的创新研究模式,必将推动IBD发病机制研究迈向新的里程碑。不知道怎么写出创新的生信方向的小伙伴可以来找馆长!这里有新鲜出炉的生信热点方向,还有一大波的可复现的创新思路,感兴趣的直接来公众号后台!


题目:氧化应激基因表达、DNA甲基化和肠道菌群相互作用触发克罗恩病:一项多组学孟德尔随机研究
l杂志:BMC Medicine
l影响因子:IF=9.3
l发表时间:2023年5月
研究背景
数据来源
|
数据库 |
数据类型 |
详细信息 |
|
GEO |
转录组数据集 |
克罗恩病患者(CD)和健康对照者(HCs)的肠活组织样本。 |
|
GeneCards |
OS相关基因 |
使用关键词“oxidative stress”提取OS相关基因,相关性≥评分为7分 |
|
GWAS- |
2个独立的IBD GWAS的元分析 |
12,194名CD患者和28,072名HCs, |
|
eQTLGen |
血液eQTL总结统计,37个数据集 |
包括31,684个个体的遗传数据, |
|
BSGS和LBC元分析 |
血液mQTL总结数据 |
两个欧洲队列的元分析,BSGS (n = 614), LBC (n = 1366) |
|
GTEx项目和1000IBD队列 |
肠道eQTL数据 |
GTEx (n = 860), 1000IBD (n = 299) |
|
荷兰微生物组项目(DMP) |
粪便mbQTL数据 |
包括7738个个体的数据,评估宿主遗传对肠道微生物的影响 |
|
第一附属医院中山大学(FAH-SYS) IBD多组学队列 |
RNA测序和宏基因组测序 |
招募了46名治疗前CD患者和44名HC,收集了肠活组织和粪便样本进行RNA测序和宏基因组测序,用于外部验证。 |
通过转录组数据集荟萃分析和CSEA,识别差异表达的CD相关OS基因。运用三步SMR法整合GWAS、血液中的顺式-eQTLs/顺式-mQTLs数据,确定与CD风险相关的推定血液OS基因及其调控元件。进行敏感性分析以检验异质性。整合肠道顺式eQTL和粪便mbQTL数据,通过SMR、灵敏度和共定位分析揭示OS基因与肠道菌群间的潜在相互作用。利用FAH-SYS IBD多组学队列验证差异表达基因,并研究共定位基因-微生物群之间的定向关联。通过这些方法,揭示了OS基因在克罗恩病发病机制中的作用及其与肠道菌群的相互作用。

图1 文章研究工作流程

研究结果
v克罗恩病和HC患者差异表达OS基因的Meta分析
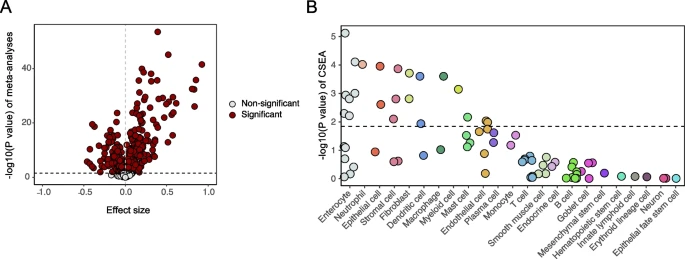
使用了6个基因表达数据集(3个微阵列数据集和3个RNA测序数据集),比较了克罗恩病(CD)患者和健康对照的肠道组织中的基因表达。通过荟萃分析,发现438个与氧化应激(OS)相关的基因在CD和对照组间存在差异性表达,其中排名前5的差异基因为DUOX2、MMP3、S100A8、MMP1和IL1B。最后,细胞类型特异性表达分析(CSEA)表明这些差异表达的OS基因在肠道上皮细胞中显著富集。

v血液甲基化调控基因表达介导的推定CD致病基因
运用三步式SMR方法,对克罗恩病(CD)患者与健康对照(HC)中的438个差异表达的氧化应激(OS)相关基因进行分析,并将这些基因的eQTL和mQTL与最大规模的CD GWAS数据整合。结果发现了16个可能存在因果关系的OS基因和8个可能调控5个邻近基因(BAD、SHC1、STAT3、MUC1和GPX3)的差异甲基化位点。进一步利用三步式SMR方法,从整合的eQTL、mQTL和GWAS数据中,鉴定出两个关键的氧化应激相关基因STAT3和GPX3可能在克罗恩病发病机理中起因果作用。其中 STAT3 增强子区域的较低 DNAm 水平上调了 STAT3 的表达,随后增加了 CD 风险。

v整合来自肠道组织的GWAS和OS-eQTL/mbQTL数据
这项研究进一步扩展了分析范围,利用来自GTEx和1000IBD项目的肠道组织eQTL数据,通过元分析识别出392个与氧化应激相关的差异表达基因。随后采用SMR方法,从中筛选出5个可能在克罗恩病发病中起因果作用的肠道表达基因:MUC1、CD40、PARK7、PRKAB1和NDUFS1。为了探讨这些肠道氧化应激基因与肠道菌群互作的可能机制,研究团队进一步整合了肠道甲基化QTL(mbQTL)数据,采用共定位分析的方法发现6对基因表达-微生物通路之间存在显著关联,其中包括前述3个基因MUC1、CD40和PRKAB1。这些结果表明,通过整合肠道组织特异的基因调控机制数据,可以发现一些新的候选基因靶标,它们可能参与了宿主基因与肠道菌群之间的互作,从而影响克罗恩病的发病过程。这为进一步阐明克罗恩病发病的复杂分子机制提供了新的线索。
v参与肠道基因-微生物群相互作用的推定CD致病基因
这项研究通过SMR和共定位分析,深入探讨了MUC1、CD40和PRKAB1三个重点基因在克罗恩病发病机制中的作用。发现MUC1基因表达上调可能是CD发病的因果因素,而CD40和PRKAB1基因表达的变化可能分别通过调控肠道炎症相关的微生物代谢产物和嘌呤核苷代谢来降低CD风险。
 |
vFAH-SYS 队列中 OS DEG 的外部复制和合理的基因-微生物群相互作用
利用独立的肠道转录组测序数据集(FAH-SYS队列)验证了前期meta分析得到的438个差异表达基因,发现大部分基因在克罗恩病组和对照组中的表达方向一致。接着,通过肠道转录组和肠道菌群测序数据,探讨了基因表达与微生物通路之间的相关关系,发现MUC1和PRKAB1基因的表达与特定细菌丰度呈负相关。进一步支持了这些基因在克罗恩病发病机制中的作用,提供了更可靠的证据支持。

文章小结
这篇文章无疑为从事肠道疾病研究的小伙伴们提供了一个很好的参考范例。首先,这篇文章选择了一个临床高需求的热点领域 – 克罗恩病相关的氧化应激基因,这无疑能引起审稿人和读者的高度关注。这篇文章还巧妙地将生信分析与实验验证相结合,既可以加快研究进度,又可以提高研究结果的可靠性。这种融合创新的研究思路确实非常值得学习。馆长觉得大家可以在这个思路的基础上,将研究对象换成其他肠道疾病,或者针对某些国自然项目进行相应的拓展,相信一定能取得不错的成果。感兴趣的话,快快行动起来吧!想复现思路的朋友快快联系馆长吧!
小果还提供思路设计、定制生信分析、文献思路复现;有需要的小伙伴欢迎直接扫码咨询小果,竭诚为您的科研助力!

定制生信分析
服务器租赁
扫码咨询小果

往期回顾
|
01 |
|
02 |
|
03 |
|
04 |