临床分类变量如何确认关联?教你一招卡方检验
点击蓝字 关注我们
小伙伴们大家好,我还是那个大海哥!今天大海哥在各大论坛社区学习的时候突然看到一个问题:我的数据有很多分类变量,我该怎么看它们是否有关联性比较好?下面有的小伙伴表示,直接相关性分析不就好了。大海哥觉得,相关性分析更适合连续性变量,而分类变量的话,可以考虑使用卡方验证来验证变量之间的相关性。那么今天大海哥就特意准备了一个案例教学,来让大家遇到这类问题时,不慌不忙的完美解决!
首先,我们来了解一下,卡方验证到底是什么?
卡方检验(Chi-Square Test)是一种常见的统计方法,用于比较观察频数与期望频数之间的差异,从而判断两个或多个分类变量之间是否存在关联性。它常用于分析分类数据,例如不同组别的频数或百分比。其原理呢?就是基于观察频数与期望频数之间的差异是否显著。观察频数值得是实际观察到的样本数据中各个类别的频数,而期望频数是在无关联情况下,根据总体比例和样本大小计算得出的理论值。
一般来说,卡方验证的流程有如下6步,
1、建立假设:分为两种假设
a)零假设(H0):两个或多个分类变量之间没有关联,差异是由随机因素引起的。
b)对立假设(H1):两个或多个分类变量之间存在关联,差异不是由随机因素引起的。
2、计算期望频数:
根据总体比例和样本大小计算出每个类别的期望频数。
3、计算卡方值:
计算观察频数与期望频数之间的差异,使用以下公式:
卡方值 = Σ[(观察频数 – 期望频数)2 / 期望频数]
4、确定自由度
自由度是一个决定卡方分布的参数,自由度的计算方法为:自由度 = (行数-1) * (列数-1),其中行数和列数分别是分类变量的类别数。
5、计算P值
将计算得到的卡方值与自由度对应的卡方分布表进行比较,或者使用统计软件计算出P值(显著性水平)。
6、做出统计决策
如果P值小于设定的显著性水平(通常为0.05),则拒绝零假设,认为分类变量之间存在关联。如果P值大于显著性水平,则无法拒绝零假设,认为分类变量之间没有关联。
好了,理论学习完毕,开始同样重要的实践部分吧!
#卡方检验R语言实现#首先我们先生成一个临床数据样例set.seed(7897)data <- data.frame(agegroup=sample(c(">=60","<60"),45,replace = T),smoke=sample(c("tobacco","NOtobacco"),45,replace=T),drink=sample(c("alcohol","NDalcohol"),45,replace=T),tea=sample(c("leaf","NOleaf"),45,replace =T))#先来看看数据head(data)

#现在假如我们想用卡方检验看看age和smoke两者之间是否存在一定的联系,就可以单个检验#先看看大致实际情况table(data$agegroup,data$smoke)

#卡方检验s=chisq.test(data$agegroup,data$smoke,correct = TRUE)#看看期望频数是多少

#再看看卡方值和P值是多少

#结果P值为0.67,大于0.05,并没有说明分类变量之间没有关联#就这么简单?当然不是,如果上面期望频数小于1,我们就需要用fisher检验去对这两列进行重新计算#我们手动调整一下数据data$drink[3:45]="alcohol"data$tea[1:44]="NOleaf"#再试一次s=chisq.test(data$agegroup,data$drink,correct = TRUE)s#expected
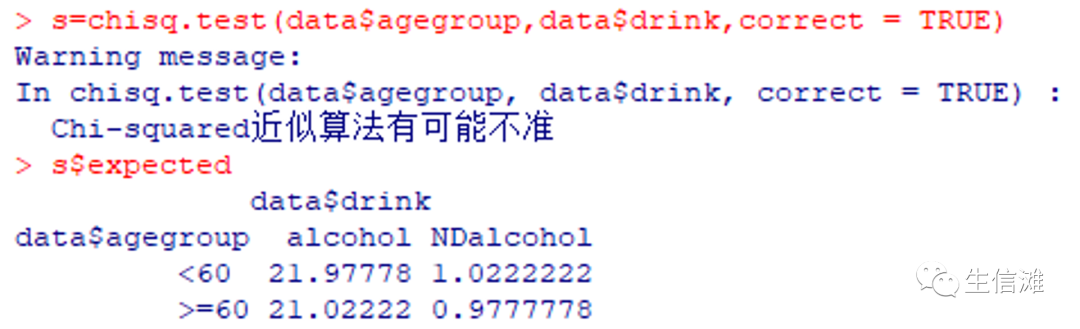
#会出现提示,同时期望频数出现小于1的情况#就需要fisher检验fisher.test(data$agegroup,data$drink)
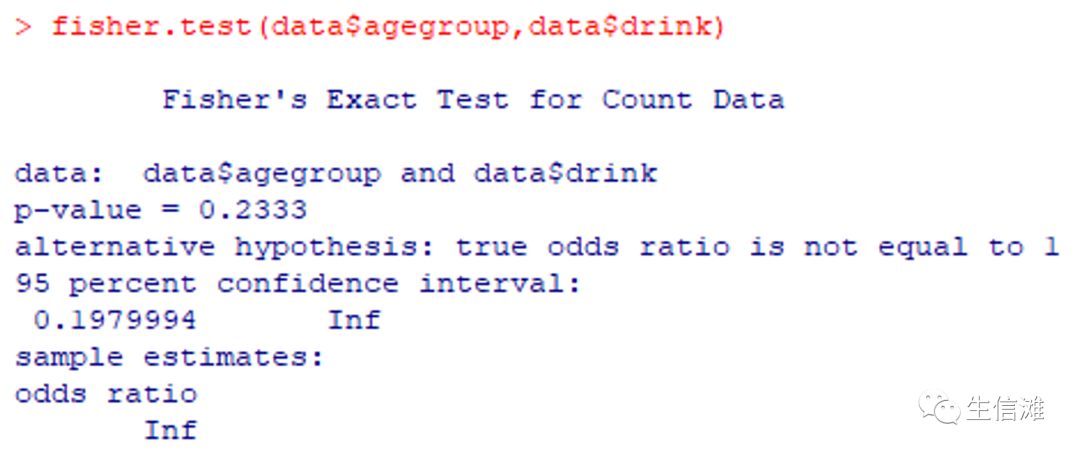
#那如果变量很多该怎么办?不能一个一个的运行吧?#别着急,大海哥这就教你如何批量处理#首先导入R包purrrlibrary(purrr)#主要分类变量选择年龄,大家可以选择自己需要分析的变量哦!FUN<- function(x){chisq.test(data[,"agegroup"],x ,correct = TRUE)}#需要计算的变量为2到4列chi_result<-purrr::map(data[,2:4],FUN)FUN2<-function(x){b1=x$expected}#把期望频数都放入结果中expected_result<-map(chi_result,FUN2)expected_result

#转换为二维矩阵expected_result2<-map_dfr(chi_result,FUN2)expected_result2=as.data.frame(as.matrix(expected_result2))names(expected_result2)
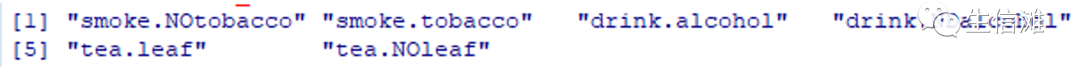
#看看哪些关联计算的期望频数出现小于1的情况a1=unique(which(expected_result2<1,arr.ind=T)[,2])a1
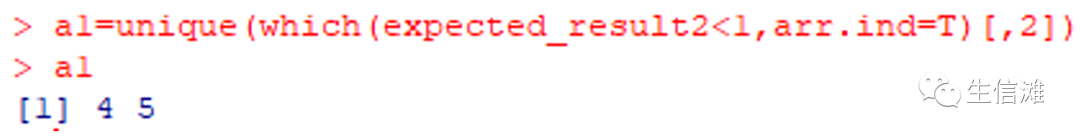
#说明drink和tea在计算期望频数时出现小于1的情况#可以针对这几个做fisher处理啦!#直接导出所有的结果,然后对drink和tea做fisher验证tab1<-CreateTableOne(data=data,#指定分组变量strata="agegroup",#指定要分析的变量vars = colnames(data)[2:4],#设置属于分类变量的factorVars=colnames(data)[2:4],#添加Overall列的分析结果addOverall=TRUE)#最终结果Table1<-print(tab1,showAllLevels=TRUE,#显示所有水平,不折叠cramVars=colnames(data)[1:4],#设置属于分类变量的#nonnormal=“”,设置属于两虚非正态的变量exact= c("drink","tea"),#指定需要进行fisher检验的变量catDigits=4,#分类变量保留小数位数contDigits=4, #连续变量保留小数位数quote=FALSE,#不显示引号noSpace=TRUE,#是否删除为对齐而添加的空间printToggle=FALSE#输出matrix)#打印一下Table1

#结果一目了然,因为大海哥是随机生成的数据,所以结果不具备参考性,但是代码及分析流程都是可以直接使用的哦!大家放心的拿自己手头上的数据试一试吧!如果结果不错,说明大海哥的这次分享非常有价值!快去动手试试吧!(最后推荐一下大海哥新开发的零代码云生信分析工具平台,包含超多零代码小工具,上传数据一件出图,感兴趣的小伙伴欢迎来参观哟,网址:http://www.biocloudservice.com/home.html)
点击“阅读原文”进入网址