药物分子设计part6:保姆级教程直通大神!全网最详细的GOLD对接教程
收录于话题

小伙伴们大家好,在前面的内容中小果给大家分享了如何定义活性位点、Libdock分子对接的详细参数介绍以及操作流程和Discovery studio提供的各项分子对接功能以及如何查看分子对接结果,相信小伙伴们都是收获满满。
但是如果想要成长为大神,分子对接只掌握Libdock一种是远远不够的。那么这一次小果就给大家分享一下GOLD分子对接详细教程。废话不多说,我们直接开始吧~
GOLD全部参数:

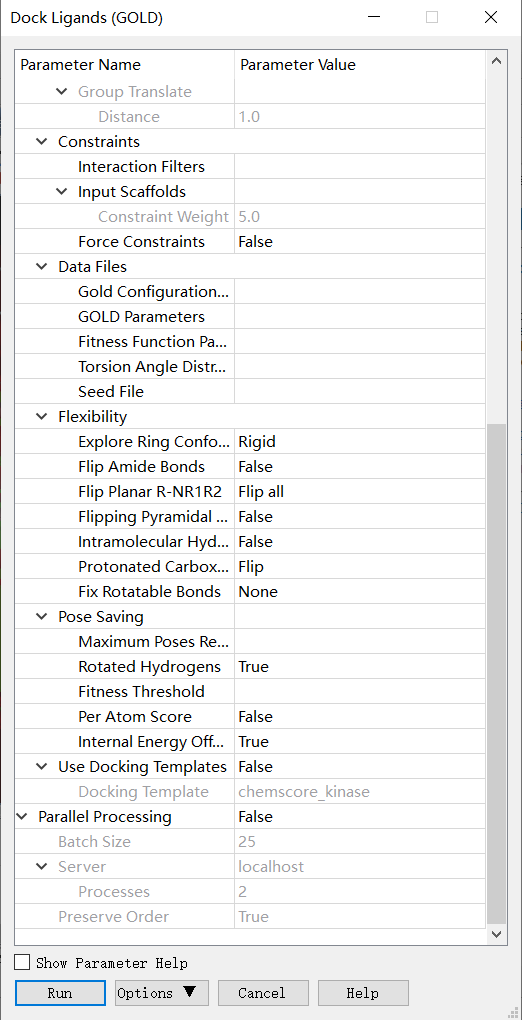
GOLD官方文档:



GOLD 5.2用户手册:Protein–ligand Docking in | CCDC (cam.ac.uk)
GOLD参数官方文档:







参数详解:
下面小果就给小伙伴们详细介绍一下GOLD的各项参数信息以及应该如何设定:
基础设置:
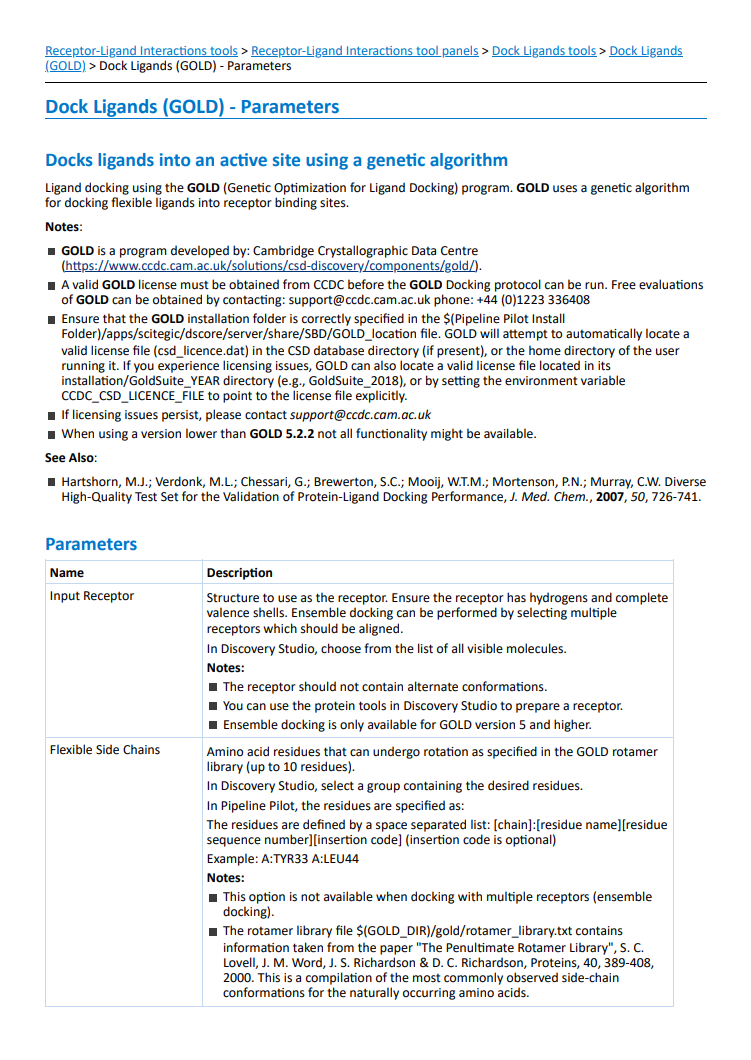
Input Receptor:选择受体,一般设置为蛋白名:蛋白名;(注意:受体不应包含交替构象)
Flexible Side Chains:当进行某些分子模拟或蛋白质结构的计算时,可以允许一些氨基酸残基不保持固定的构象而发生旋转或变动,从而更好地模拟分子或蛋白质在不同环境下的柔性行为。
在Discovery Studio软件中,我们可以选择一个包含你所需残基的群组。而在Pipeline Pilot中,我们需要通过指定链的名称、残基的名称、残基的序号和插入代码(可选)来定义残基。(例如,A:TYR33表示链A上的TYR残基的第33个残基,A:LEU44表示链A上的LEU残基的第44个残基。)
(注:在进行特定的对接计算时,可能不支持同时对多个受体进行对接。)
GOLD软件内部包含了一个rotamer库文件,其中记录了常见氨基酸侧链的旋转构象信息。
Input Receptor Fix All Bonds:在对接过程中,受体中的丝氨酸、苏氨酸、赖氨酸NH3+和酪氨酸羟基团将被进行优化(旋转)。如果不希望进行这种旋转,可以将该值设置为True以禁用。
Input Ligands:选择对接的小分子,一般设置为小分子名:All;
Input Site Sphere:参数为定义好的活性位点的坐标;小伙伴们可以在前面定义活性位点时查看每个位点的坐标,从而在这里进行选择。这一项也可以填写空白,在下一项Input Site Atoms中通过受体原子索引选择。
Input Site Atoms:选择包含所需原子的群组即可定义结合位点。
适应度函数:
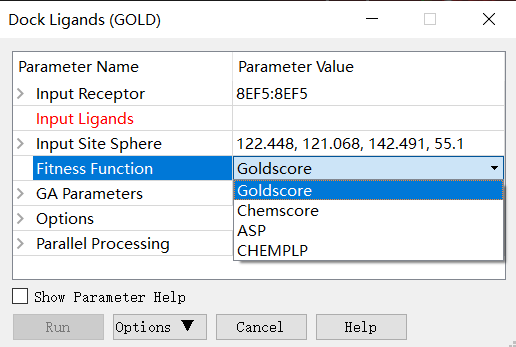
Fitness Function:用于评估或计算在蛋白质-配体对接过程中不同构象(姿态)的质量或适应性。GOLD提供四种评分函数:Goldscore, Chemscore, ASP, CHEMPLP。
Goldscore:是GOLD软件提供的原始评分函数。它经过优化,用于预测配体结合位置,并考虑了氢键能、范德华能和配体扭曲应变等因素。
Chemscore:通过回归分析结合亲和力数据进行训练,估计配体结合时发生的总自由能变化。
ASP:Astex Statistical Potential (ASP) 是从蛋白质-配体复合物数据库中得出的原子-原子势能,可与其他评分势能(如PMF和Drugscore)进行比较。ASP具有与Chemscore和Goldscore相当的准确性。
CHEMPLP:Piecewise Linear Potential 是一种针对位姿预测进行优化的经验适应度函数。Piecewise Linear Potential (PLP) 用于建模蛋白质和配体之间的立体互补性,而CHEMPLP还考虑了Chemscore中基于距离和角度的氢键和金属键结合项。

遗传算法:

GA Parameters:预定义的遗传算法(Genetic Algorithm),用于控制结果的速度和质量。
有以下几个选项供大家选择:
GOLD Default:具有较高的预测准确性,但相对较慢。
2 times speed up, 3 times speed up, 7-8 times speed up:两倍、三倍和七到八倍的对接速度。
Automatic:自动参数设置。
Automatic Library Screening:最快的设置,因此可靠性较低。Automatic Virtual Screening:适用于常规工作,通常与较慢的设置具有可比较的预测准确性,除非配体具有大量可旋转的扭曲。
Automatic Default:GOLD将尝试为每个配体应用最佳设置。
Automatic Very Flexible:一般用于大型高度灵活的配体。该设置预测准确性较高,但相对较慢。
GA Automatic Search Efficiency:控制对接速度和预测准确性的参数。如果留空,则会使用基于GA参数的自动预设设置。该参数可设置在10%和200%之间。
当选择自动设置时,GA参数将根据每个配体进行实时确定,并进行优化。由于遗传算法依赖于变化来寻找解决方案,限制操作次数会使搜索空间更受限制。该参数允许我们减少搜索空间的覆盖范围,从而增加对接速度。
如果需要更快的对接时间,可以降低搜索效率百分比。使用默认设置(100%),GOLD将尝试为每个配体应用最佳设置。对于具有5个可旋转键的中等大小的配体,大约需要进行30,000次GA操作。当设置为50%时,对于该配体,在对接之前将缩小该值为15,000,从而使对接速度加快2倍。
GA Automatic Minimum Operations:将每个配体都设定一个最小操作次数。如果留空,将使用基于GA参数的自动预设设置。
GA Automatic Maximum Operations:设置每个配体的最大操作次数。同样的,如果留空,将使用基于GA参数的自动预设设置。
控制配体对接:

Options:控制配体对接的选项。
对接次数
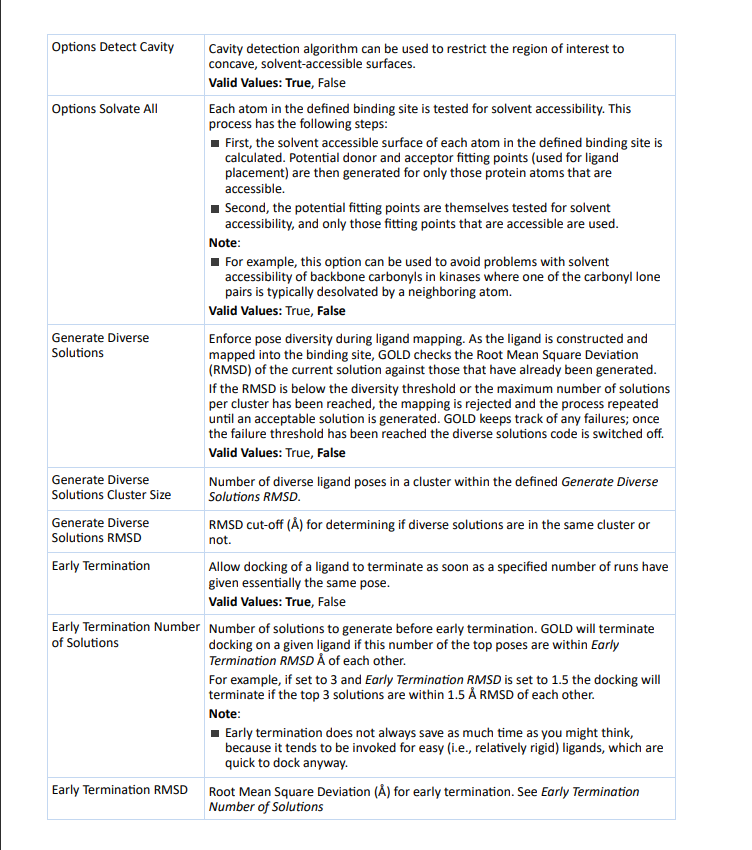
Options Number of Dockings:GOLD将对每个配体进行多次对接,每次从不同的配体取向随机种群开始,最终将不同的对接运行结果按适应度评分进行排名。减少对接次数可以加快GOLD的速度,但增加次数会提高得到最合适对接结果的机会。如果在多个不同的对接运行中找到相同的答案,通常意味着该答案是正确的。
小果提示:可以使用提前终止选项来防止GOLD在简单的配体上执行多次对接而浪费时间。
空腔检测
Options Detect Cavity:空腔检测算法可以用来限制感兴趣区域为凹凸不平、溶剂可及的表面。
溶剂可及性测试
Options Solvate All:对定义的结合位点中的每个原子进行溶剂可及性测试(计算分子中原子或分子的表面是否能够被溶剂(通常是水)接触到的方法)
该过程包括以下几个步骤:首先计算定义的结合位点中每个原子的溶剂可及表面。然后仅对可访问的蛋白质原子生成潜在的供体和受体配位点(用于配体放置)。其次对这些潜在的配位点进行溶剂可及性测试,只使用那些可访问的配位点。
小果在这里给大家举个例子,例如这个选项可以用来避免激酶中主链羰基的溶剂可及性问题,因为其中一个羰基孤对电子通常会被相邻原子脱溶剂化。
姿态多样性
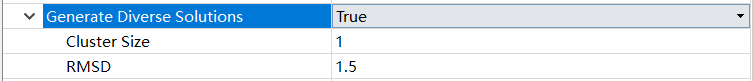
Generate Diverse Solutions:在配体映射过程中,为了增加姿态(构象)的多样性,GOLD软件会评估生成的配体构象与已有解决方案之间的均方根偏差(RMSD)。RMSD是一种用于比较两个结构之间相似性的度量。
如果生成的配体构象与已有解决方案之间的RMSD低于设定的多样性阈值,或者已经达到了每个聚类簇允许的最大解决方案数,GOLD将拒绝该映射,并重新进行映射过程。这样做是为了确保生成的解决方案具有足够的多样性。
GOLD还会追踪映射过程中的任何失败情况。如果达到了设定的失败阈值,多样性解决方案的生成代码将被关闭,即不再尝试生成更多的多样性解决方案。
Generate Diverse Solutions Cluster Size:在定义的生成多样性解决方案的RMSD范围内,聚类中不同配体姿势的数量。
Generate Diverse Solutions RMSD:用于确定不同解决方案是否属于同一聚类的RMSD截断(Å)值。
提前终止
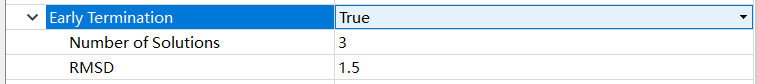
Early Termination:允许配体的对接在特定次数的运行中给出基本相同的姿势后终止。
Early Termination Number of Solutions:在提前终止之前生成的解决方案数量。如果在给定的配体中,顶部姿势的数量在提前终止RMSD Å范围内相互接近,则GOLD将终止对该配体的对接。
例如,如果将其设置为3,并将提前终止RMSD设置为1.5 Å,则当前3个解决方案的RMSD相互之间在1.5 Å以内时,对接将终止。
小果提醒大家,提前终止并不总能节省时间,因为它往往用于易于对接(即相对刚性)的配体,这些配体本身的对接速度就很快。
Early Termination RMSD:提前终止的均方根偏差(Å)值。
加入水分子
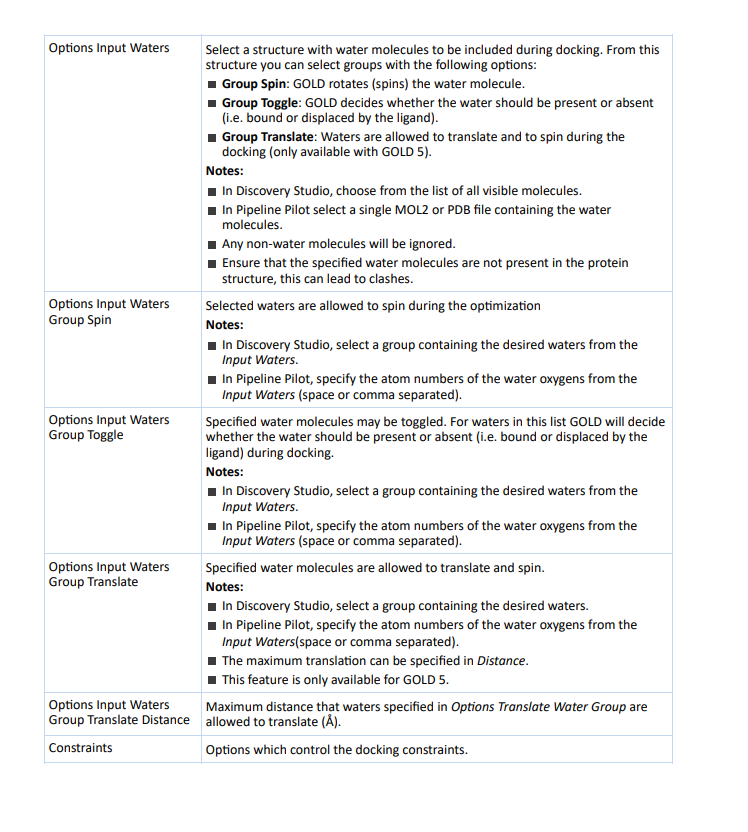
Options Input Waters:在对接过程中加入一个含有水分子的结构。
可以使用以下选项选择其中的组:
组旋转(Group Spin):GOLD旋转(旋转)水分子。
组切换(Group Toggle):GOLD决定是否存在水分子(即被配体所结合或被置换)。
组平移(Group Translate):允许水分子在对接过程中进行平移和旋转(仅适用于GOLD 5)。
Options Input Waters Group Spin:允许优化过程中所选的水分子旋转。
在Discovery Studio中,从输入水分子(Input Waters)中选择包含所需水分子的组。在Pipeline Pilot中,从输入水分子(Input Waters)中指定水氧原子的原子编号(空格或逗号分隔)。
Options Input Waters Group Toggle:指定的水分子可以进行切换。对于列表中的水分子,在对接过程中,GOLD将决定水分子是否存在(即被配体所结合或被置换)。
Options Input Waters Group Translate:指定的水分子可以进行平移和旋转,最大平移距离可以在距离中指定。
小果提醒大家,此功能仅适用于GOLD 5版本。
Options Input Waters Group Translate Distance:指定水分子允许进行平移的最大距离(单位为Å)。
约束选项

Constraints:控制对接约束的选项
Interaction Filters:应该与配体形成氢键的蛋白质原子。
小伙伴们可以使用Discovery Studio中的”Dock Ligands”工具,使用”Define Interaction Site”命令来定义这些相互作用。在Pipeline Pilot中,需指定约束权重(constraint weight)、几何权重(geometry weight)和原子编号。约束权重定义了GOLD在形成指定氢键时施加的偏好强度。权重可以在Discovery Studio中的数据表或首选项中进行设置。
几何权重定义了一个氢键相互作用必须有多好才被GOLD认为是一个氢键(取值范围为0到1,默认为0.005)。
例如:10 0.005 1983 1984表示对于原子编号为1983和1984的原子,指定了约束权重为10,几何权重为0.005。
小果提醒大家,在使用多个受体进行对接(集合对接)时不可用此功能。
Constraints Input Scaffolds:指定一个或多个包含配体或片段的文件(通常是MDL MOL/SD文件)用于限制匹配过程。我们可以通过使用脚手架匹配约束,将片段固定在结合位点的特定位置上,而在对接过程中不会改变片段的形状。
小伙伴们可能会有疑问,什么是“脚手架”呢?在药物发现和化学合成中,脚手架(scaffold)是指分子结构中的核心框架或基本骨架。它是具有特定功能和活性的化合物系列中共有的部分。脚手架通常由一系列原子和键连接而成,代表了化合物的主要结构特征。
对于每个配体,只能同时匹配一个脚手架。系统将按照我们指定的顺序逐个评估脚手架,首先与配体匹配成功的脚手架将被使用。这意味着你可以指定两个或多个不同的脚手架,系统将选择首先匹配成功的脚手架。这在对接多个不同系列的化合物时非常有用。
在Discovery Studio中,我们还可以选择指定活动分子窗口中的所有、选定或可见的配体。然而,为了获得最佳性能,小果建议大家使用文件来指定配体或片段。
需要注意的是,如果指定的文件在服务器上可用,性能会更好,因为系统不需要创建文件的副本。
Constraints Input Scaffolds Constraint Weight:该参数决定配体原子与脚手架的匹配程度,将权重设置得更高将会更严格地强制配体被放置在脚手架的位置上,如果将权重设置得更低,配体在脚手架上的放置可能会更加灵活,允许与脚手架指定的精确位置有些偏离。
Constraints Force Constraints:指定约束条件,当它在物理上无法满足时(例如,配体中没有适合形成所需氢键约束的适当基团),配体将不会被对接。
导入配置文件

小果提醒大家:配置文件的格式是严格的,不正确的语法可能导致GOLD表现出意外的行为,甚至崩溃。由于参数众多,无法保证程序在除默认参数化以外的情况下能够可靠运行。
Data Files Gold Configuration File:使用指定的配置文件来覆盖对接参数,但保留输入配体不变。请大家在预先准备好的配置文件上使用此选项。
小果提醒大家:当从Pipeline Pilot客户端或Web端口运行时,请大家确保GOLD配置中的数据文件路径已正确指定并对服务器可见。
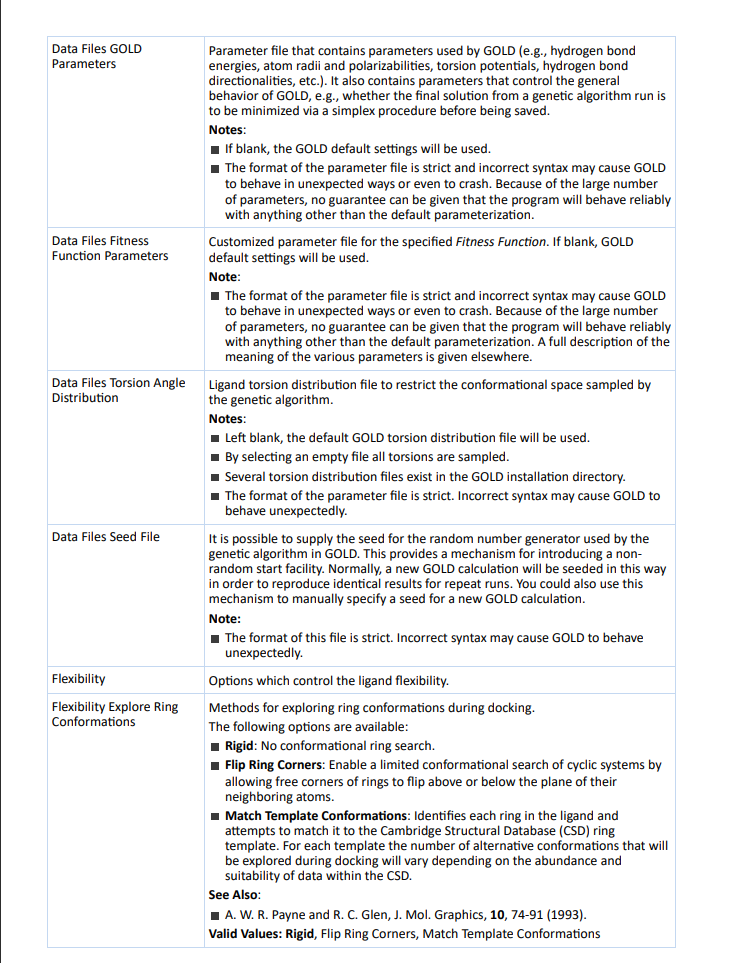
Data Files GOLD Parameters:参数文件包含GOLD使用的参数,例如氢键能量、原子半径和极化率、扭转势能、氢键方向性等。它还包含控制GOLD的整体行为的参数,例如是否在通过遗传算法运行的最终解进行保存之前,要通过单纯形过程进行最小化。如果留空,则将使用GOLD的默认设置。
Data Files Fitness Function Parameters:为指定的适应度函数定制的参数文件。如果留空,将使用GOLD的默认设置。
Data Files Torsion Angle Distribution:配体扭曲分布文件用于限制遗传算法采样的构象空间。如果留空,将使用默认的GOLD扭曲分布文件。
Data Files Seed File:为GOLD中遗传算法使用的随机数生成器提供种子。这提供了引入非随机起始设施的机制。通常,为了重现重复运行的相同结果,新的GOLD计算将以此种子为基础。我们可以使用此机制来手动指定新的GOLD计算的种子。
小伙伴们可能不太理解,什么是种子呢?
在计算机科学中,种子(seed)是用于生成随机数序列的初始值或起始点。随机数生成器根据种子来确定生成的随机数序列,相同的种子将产生相同的随机数序列。种子可以被视为随机数生成器的状态的表征。通过指定种子,我们可以控制随机数生成的可重复性,使得在相同种子下生成的随机数序列是可预测且可重现的。
在GOLD中,种子用于控制遗传算法的随机数生成过程,以确保在重复运行时可以获得相同的结果。
小分子柔性
Flexibility Explore Ring Conformations:在对接过程中探索环构象的方法。
有以下选项可供选择:
1.刚性(Rigid):不进行环构象搜索。
2.翻转环角(Flip Ring Corners):通过允许环的自由角在邻近原子的平面上方或下方翻转,对环系统进行有限的构象搜索。
3.匹配模板构象(Match Template Conformations):识别配体中的每个环,并尝试将其与剑桥结构数据库(Cambridge Structural Database,CSD)中的环模板进行匹配。对于每个模板,将根据CSD中数据的丰富性和适用性来决定在对接过程中将探索的可替代构象数量。
Flexibility Flip Amide Bonds:在配体初始化过程中,配体的酰胺基团(例如硫酰胺、脲和硫脲)将被设置为反式构象。如果(O)C-N(H)的扭转角大于平面外20°,也会进行平化处理,使其达到反式构象。
为了在顺式和反式构象之间进行翻转,首先将CO-NRR’的扭转角设置为平面构象(以初始化的反式构象为基准)。
小果提醒大家:如果(O)C-N(H)的扭转角大于5°但小于20°,并不会进行平化处理。
Flexibility Flip Planar R-NR1R2:允许配体中与sp2碳原子相连的平面三价氮在对接过程中在顺式和反式构象之间翻转(否则,它们将被固定在输入几何构型上)。
以下选项可供选择:
1.Fixed:将键固定在其输入构象上。
2.Flip non-ring only:仅翻转非环部分。
3.Flip non-ring and ring-NHR1:允许非环部分和环-NHR1翻转(即进行180°旋转)。
4.Flip non-ring and ring-NR1R2:允许非环部分和环-NR1R2翻转(即进行180°旋转)。
5.Flip all:允许全部翻转。
Flexibility Flipping Pyramidal Nitrogens:允许在对接过程中非平面的sp3氮原子进行倒转(否则,它们将被固定在输入几何构型上)。对于非平面的基团RR’R”N或四面体环绕的RR’R”NH,”Flip pyramidal N”开关使氮周围的局部立体化学发生翻转(围绕氮进行伞状几何变化的能垒较低)。
小果提醒大家:翻转仅改变RR’R”N和RR’R”NH氮原子周围的立体化学,不会影响其他手性中心。
Flexibility Intramolecular Hydrogen Bonds:允许在对接过程中形成配体内部的氢键。
小果提醒大家:慎重使用此选项,因为它可能会导致类似甲氨蝶呤的配体卷曲起来。
Flexibility Protonated Carboxylic Acids:控制在对接过程中质子化羧酸的行为。
有以下选项可供选择:
1.Fixed:保持刚性,不发生变化。
2.Flip:允许翻转(即旋转180°)。
3.Rotate Freely:允许自由旋转。
Fix Rotatable Bonds:固定特定键或配体的部分几何构型,例如为了研究预定配体几何结构的可能结合。以下选项可供选择:
1.None:允许完全灵活性。
2.All:固定所有可转动键,即保持输入构型。
3.All But Terminal:固定所有非端部可转动键(即不包括-CH3,-OH等),在其输入构型上。
小果提醒大家:
当将所有可转动键固定在其输入构型上(即进行刚性配体对接)时,GOLD将通过映射给体受体键(以及疏水-疏水)匹配点来找到配体在结合位点中的最佳方向。但是,GOLD不会对最终解进行局部优化(单纯形法)。这可能会导致对接结果接近最优构型而受到惩罚。在对接之前进行几次分子力学最小化循环,可以帮助将配体靠近其局部势能最小值。
保存构象

Pose Saving Maximum Poses Retained:保存的最大构象数,留空或小于1表示保存所有解决方案,否则将保留每个对接配体的指定数量的构象。
Pose Saving Rotated Hydrogens:保存在对接过程中生成的极性蛋白质氢原子的优化位置(这些位置通常对于每个对接配体姿势都是不同的)。此选项控制是否将旋转的蛋白质氢原子的位置保存到对接解决方案文件中:
Gold.Protein.RotatedTorsions:旋转原子扭转。
Gold.Protein.ActiveResidue:活性残基。
Gold.Protein.RotatedAtoms:旋转原子。
Pose Saving Fitness Threshold:根据指定的数值筛选所有适应度得分低于该值的解决方案。筛选通过只保留最佳解决方案来减少姿势的数量。如果留空,则不启用筛选。
Pose Saving Per Atom Score:将各个配体和蛋白质原子的评分贡献属性添加到对接解决方案中。对于每个原子,将添加其对总适应度得分的贡献,以及组成的评分术语。
Pose Saving Internal Energy Offset:根据运行过程中遇到的这些项的最佳能量,修正内部能量项(内部扭转、内部范德华和内部氢键)。通过应用此修正,内部能量将相对于接近最优非结合构象的能量进行计算,从而考虑到任何不可消除的内部能量。
对接模板
Use Docking Templates:使用在”对接模板”中指定的GOLD模板。
小果提醒大家:模板文件中设置的参数将覆盖等效的协议参数。
Docking Template:对接模板为多种不同的对接协议分配了优化的设置。
以下是GOLD安装中提供的默认模板:
chemscore_kinase:针对激酶对接进行优化。
chemscore_p450_csd:针对细胞色素P450对接进行优化。
gold_folate_enzymes_VS:用于识别叶酸酶活性分子的虚拟筛选模板。
gold_kinase_VS:用于识别激酶活性分子的虚拟筛选模板。
gold_metallo_protease_gen_VS:用于识别金属蛋白酶活性分子的虚拟筛选模板。
gold_nuclear_hormone_rec_VS:用于识别核激素活性分子的虚拟筛选模板。
gold_phosphodiesterase_VS:用于识别磷酸二酯酶活性分子的虚拟筛选模板。
gold_serine_protease_VS:用于识别丝氨酸蛋白酶活性分子的虚拟筛选模板。其中,对接评分函数使用Goldscore,重评分函数使用ASP。
goldscore_p450_csd:针对细胞色素P450对接进行优化的模板。
小果提醒大家:参数列表列出了在GOLD/templates目录中可用的所有模板文件。大家可以轻松地添加或修改GOLD/templates目录中的文件。任何在模板文件中指定的参数都会覆盖协议接口中设置的参数。如果找不到GOLD/templates目录,将抛出错误。
并行执行:
Parallel Processing Batch Size:在Pipeline Pilot服务器上并行执行此协议。我们可以设置每个处理批次中发送的数据记录数量、服务器名称、每个服务器上执行的协议进程数量,以及是否保留已处理的数据记录的顺序。
小果提醒大家:协议的并行部分不是全部运行,这可能会影响扩展性。一般来说,当并行化的好处超过指定批次大小的数据传输开销时,性能会提高。默认情况下,Pipeline Pilot服务器配置允许每个协议最多四个进程。大家可以在Pipeline Pilot服务器管理门户上进行调整。
Parallel Processing Server:提供逗号分隔的服务器列表(服务器名称:端口号),用于并行处理。
在多CPU/多核系统上(其中子协议作业在同一台机器上启动)和Pipeline Pilot Linux集群上(其中”localhost”表示在同一集群上启动子协议作业),如果要包含本地服务器,大家可以添加”localhost”。
Parallel Processing Server Processes:对每个服务器同时执行的最大批次数进行逗号分隔的列表设置。
小果提醒大家:每个值必须与服务器列表中的一项对应,此参数中的条目数量必须等于服务器的数量。
Parallel Processing Preserve Order:保留数据记录的初始顺序。
结语
到这里为止,GOLD分子对接的全部参数详细信息以及如何设置和适用范围小果都给大家分享完毕啦,小伙伴们可能会觉得内容太多记不住,没有关系,小果特意给大家按功能进行了小标题分类,小伙伴们在看完之后大概记得住有哪些功能,在使用时再翻出来这篇教程查询就可以啦~
小果最后还要提醒大家,参数根据自己的需求设置,没有统一标准,但每一项参数的设置都应该十分谨慎,因为会对后续研究造成很大的影响,对于没有把握的参数一定要谨慎查阅教程,不可以随便修改哦~
那么本次的分享就到这里结束啦,如果小伙伴们平时在生信分析的操作过程中遇到困难,欢迎大家使用小果开发的生信工具平台http://www.biocloudservice.com/home.html哦。我们下次再见啦,拜拜~


往期推荐