宝藏知识点!单基因生存分析-最佳截断点选取(cutoff)

许多小伙伴们在做单基因生存分析时候,通常使用中位数或者四分位数等特殊位置为截断点。但是结果往往没有差异甚至毫无意义。
其实呢,并不一定使用中位数为cutoff,必要的时候使用最佳的cutoff。这个时候小果发现,如果找到一个最佳的截断的便可以使生存分析有差异。目前有很多方法去选择:如X-tile软件、R的CatPredi包、cutoff包等,但在这里小果主要给小伙伴们介绍一种方法使用R包survminer中surv_cutpoint()函数进行计算,选择生存分析的最佳截断点,然后绘制生存曲线图,这样得出的结果是有差异的。话不多说,小果带你走一遍。
首先载入数据:
setwd("E:\生信果")surv <- read.table("data.txt",sep = "t",row.names = 1,check.names = F,stringsAsFactors = F,header = T)
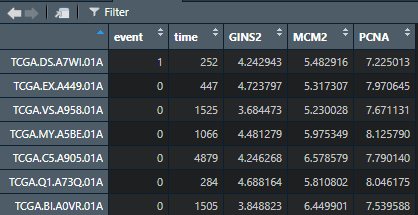
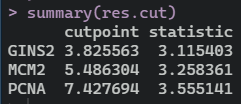
接下来计算最佳阶段点:library(survival)library(survminer)res.cut <- surv_cutpoint(surv,time = "time",event = "event",variables = c("GINS2","MCM2","PCNA"))summary(res.cut)
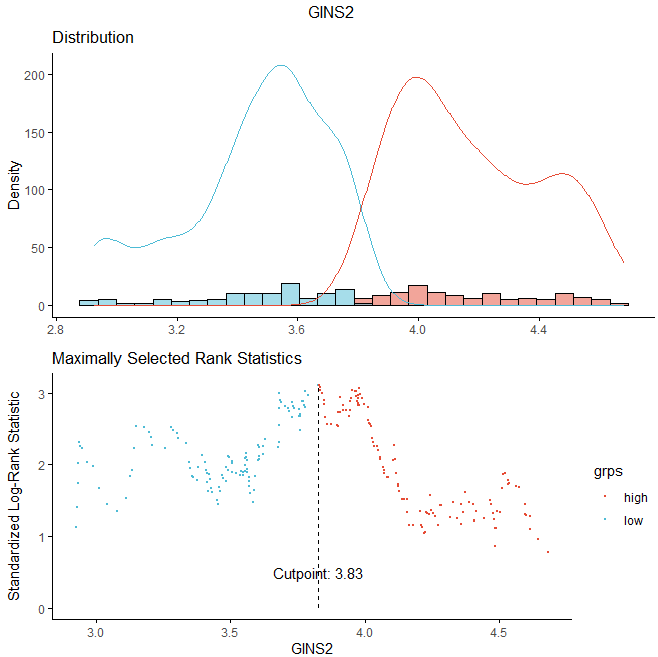
#下面这里小果展示数据的分布进行可视化,以GINS2为例:plot(res.cut, "GINS2", palette = "npg")res.cat <- surv_categorize(res.cut)head(res.cat)
fit <- survfit(Surv(time, event) ~GINS2, data = res.cat)library(survminer)ggsurvplot(fit,data = surv,pval = T,conf.int = TRUE,risk.table = TRUE,risk.table.col = "strata",palette = "jco",size = 1,xlim = c(0,2000),break.time.by = 500,surv.median.line = "hv",ylab = "Survival probability (%)",xlab = "Time (Days)",ncensor.plot = TRUE,ncensor.plot.height = 0.25,risk.table.y.text = FALSE)
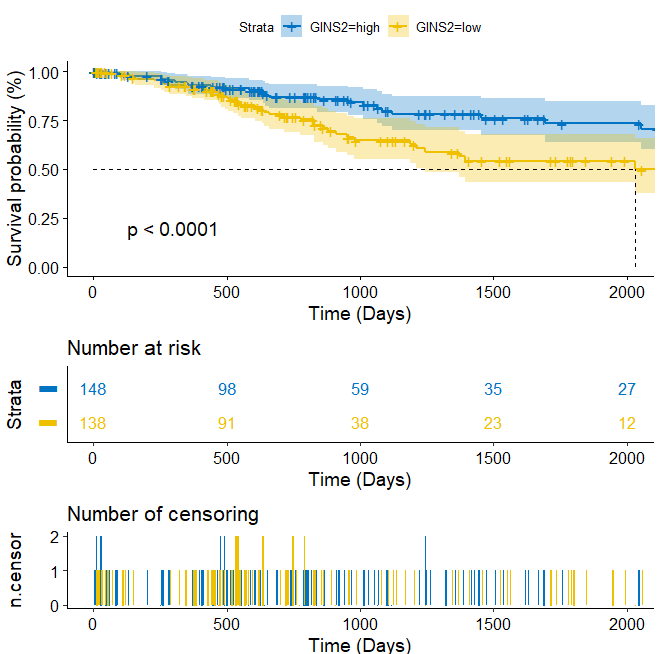
至此,单基因有差异的生存曲线图绘制完成了。图中可以看出p<0.0001,有统计学意义。小果在这里也介绍一下生存曲线中的参数,在上述代码中都有标注。小伙伴们多多研究参数的意义,可以根据自己的数据,和想要的样式修改参数,得到自己想要的结果图片。
小果还提供思路设计、定制生信分析、文献思路复现;有需要的小伙伴欢迎直接扫码咨询小果,竭诚为您的科研助力!

定制生信分析
服务器租赁
扫码咨询小果


往期回顾
|
01 |
|
02 |
|
03 |
|
04 |