生信必备分析内容,cox模型评估-决策曲线分析(DCA)
点击蓝字
关注小图
今天小图为小伙伴们带来的分享为利用DCA分析评价模型的临床净获益率,该分析在模型评价中是缺一不可的分析,小图将通过该推文介绍如何进行模型评价和模型中指标的临床净获益率,该分析非常值得小伙伴学习,小图强烈推荐,接下来和小图一起开始今天的学习吧!
1.何为DCA分析?
在开始该分析之前,小图先为小伙伴们简单介绍一下何为DCA分析?DCA曲线,即决策曲线分析法(Decision Curve Analysis),是用来帮助确定高风险患者进行干预,而低风险患者避免干预(避免过度医疗),即评价患者获益程度的一种评估方法。这就是小图对DCA分析的简单介绍,小伙伴们是不是很容易理解呀!有不理解的小伙伴,可以自行到网上查询深入学习呀!马上和小图一起开启今天的实操。
2.准备需要的R包
#安装需要的R包install.packages("survival")#加载需要的R包library(survival)library(ggDCA)library(tidyverse)source("stdca.R")
3.读取输入数据
#行名为样本名,其他列为临床因素,主要包括生存时间,生存状态等信息。pbc<-read.table("easy_input.txt")
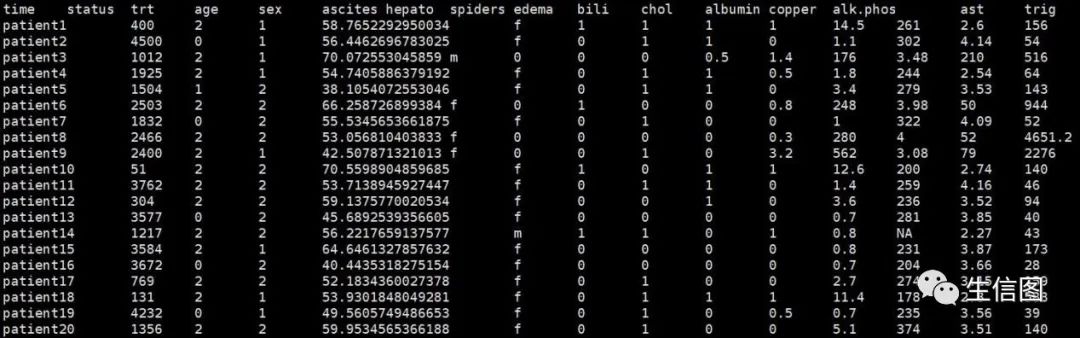
#删掉缺失数据pbc <- na.omit(pbc)#先把bili分为三类:低、中、高pbc$catbili <- cut(pbc$bili,breaks=c(-Inf, 2, 4, Inf),labels=c("low","medium","high"))#把status分为两类,原来的2为变成1,原来的1和0变成0pbc$died <- pbc$status==2pbc$status =ifelse(pbc$died=="TRUE",1,0)
4.DCA分析评价模型的临床净获益率
#多因素回归分析Srv = Surv(pbc$time, pbc$died)#此处选择5年的时间节点,输入文件的time列的单位是天,5年是1825天。#下面每两行计算1种cox模型的系数,后面将画图对比
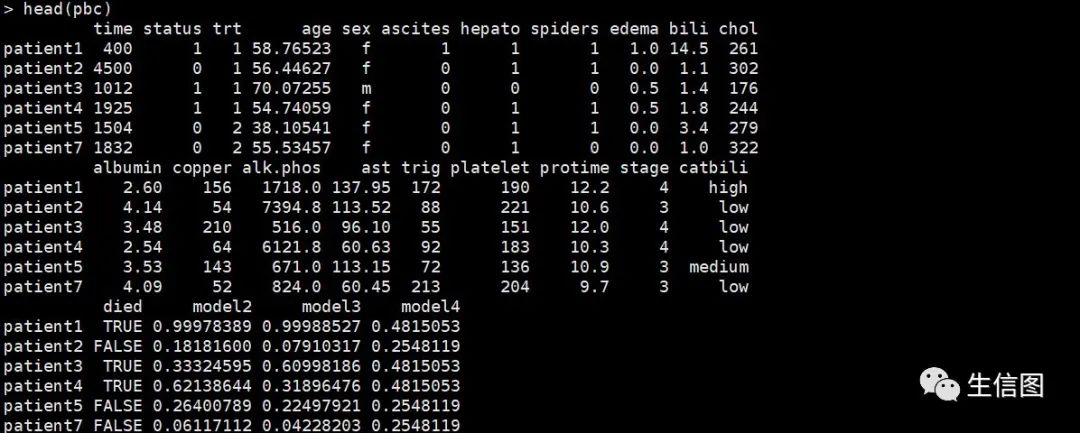
#模型一coxmod1 = coxph(Srv ~ trt + age + sex + hepato + catbili + copper + stage, data=pbc)pbc$model1 = c(1 - (summary(survfit(coxmod1,newdata=pbc), times=1825)$surv))
#模型二coxmod2 = coxph(Srv ~ ascites + spiders + edema + chol + albumin + alk.phos + ast + trig + platelet + protime, data=pbc)pbc$model2 = c(1 - (summary(survfit(coxmod2,newdata=pbc), times=1825)$surv))
#模型三coxmod3 = coxph(Srv ~ trt + age + sex + hepato + catbili + copper + stage + ascites + spiders + edema + chol + albumin + alk.phos + ast + trig + platelet + protime, data=pbc)pbc$model3 = c(1 - (summary(survfit(coxmod3,newdata=pbc), times=1825)$surv))
#模型四coxmod4 = coxph(Srv ~ stage, data=pbc)pbc$model4 = c(1 - (summary(survfit(coxmod4,newdata=pbc), times=1825)$surv))
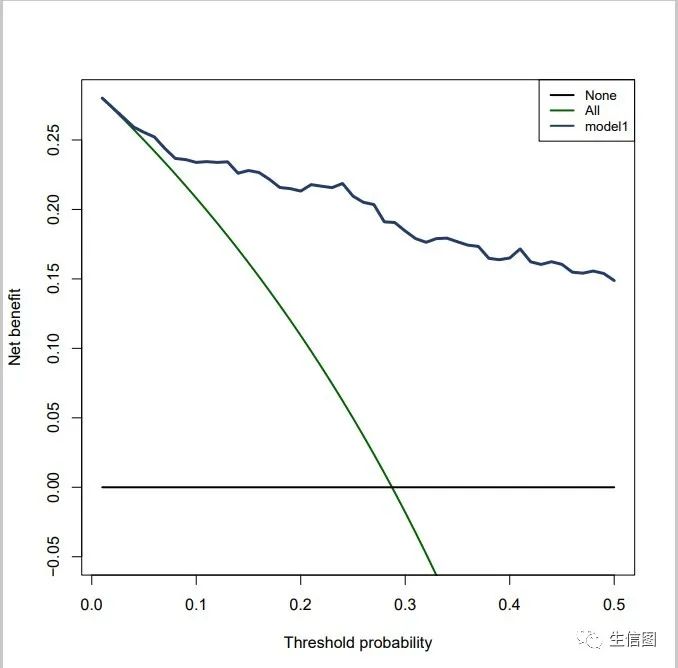
#画一条临床净获益率mod1<-stdca(data=pbc, outcome="status", ttoutcome="time", timepoint=1825,predictors="model1",cmprsk=TRUE,smooth=TRUE, xstop=0.5,intervention="FALSE")
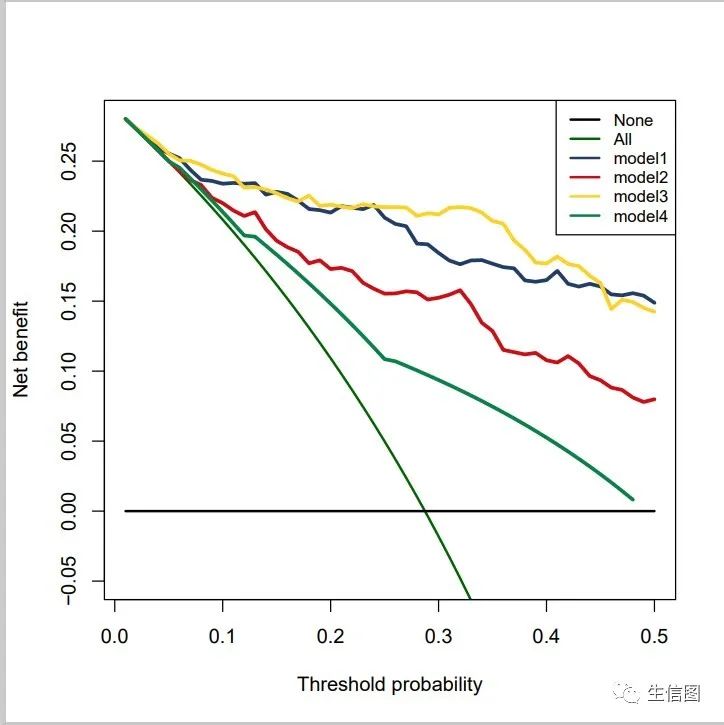
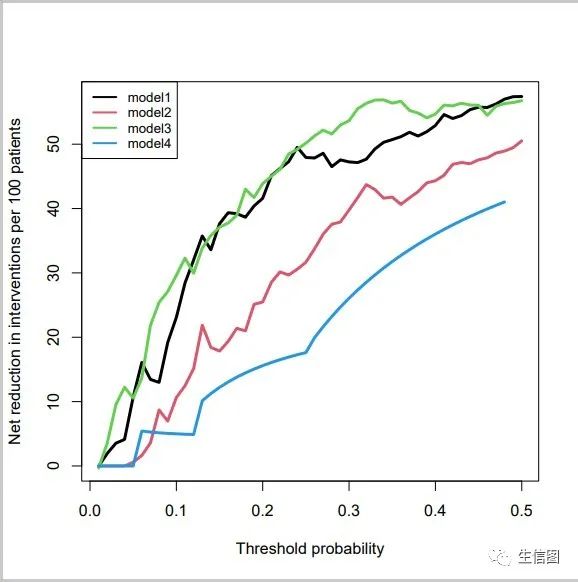
#多个模型比较pdf("net_benefit.pdf",width = 6,height = 6)stdca(data=pbc, outcome="status", ttoutcome="time", timepoint=1825,predictors=c("model1","model2","model3","model4"),cmprsk=TRUE, smooth=TRUE,xstop=0.5,intervention="FALSE")dev.off()
5.DCA分析评价模型的干预临床净获益率
#画一条干预临床净获益率pdf("3red.pdf")mod1 <- stdca(data=pbc, outcome="status", ttoutcome="time", timepoint=1825,predictors="model1",cmprsk=TRUE,smooth=TRUE, xstop=0.5,intervention="TRUE")dev.off()
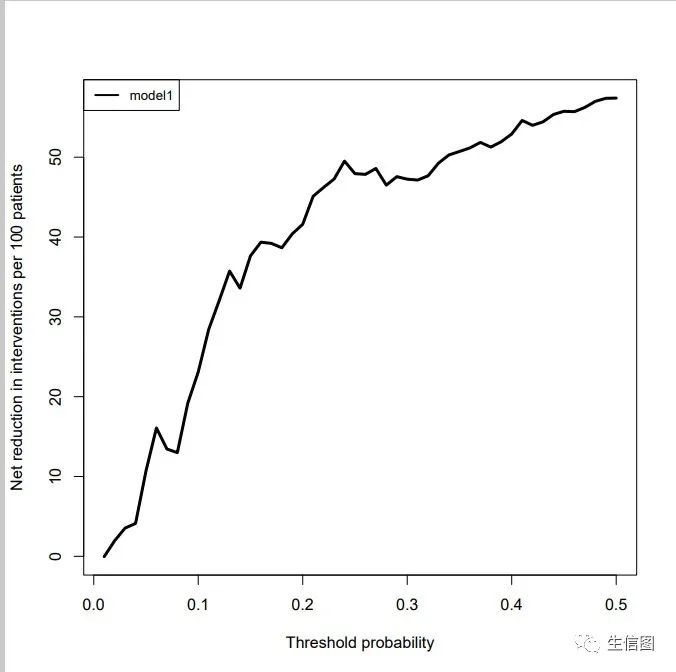
#多条比对pdf("net_reduction.pdf",width = 6,height = 6)stdca(data=pbc, outcome="status", ttoutcome="time", timepoint=1825,predictors=c("model1","model2","model3","model4"),cmprsk=TRUE, smooth=TRUE,xstop=0.5,intervention="TRUE")
6.ggDCA包绘制DCA曲线
#构建两个模型fit1 <- coxph(Surv(time,status)~age,data = pbc)fit2 <- coxph(Surv(time,status)~trt + age + sex + hepato + catbili + copper + stage,data = pbc)#绘制单个模型的DCA曲线plot1 <- dca(fit1,times = 365)ggplot(plot1)ggsave("dca1.pdf")
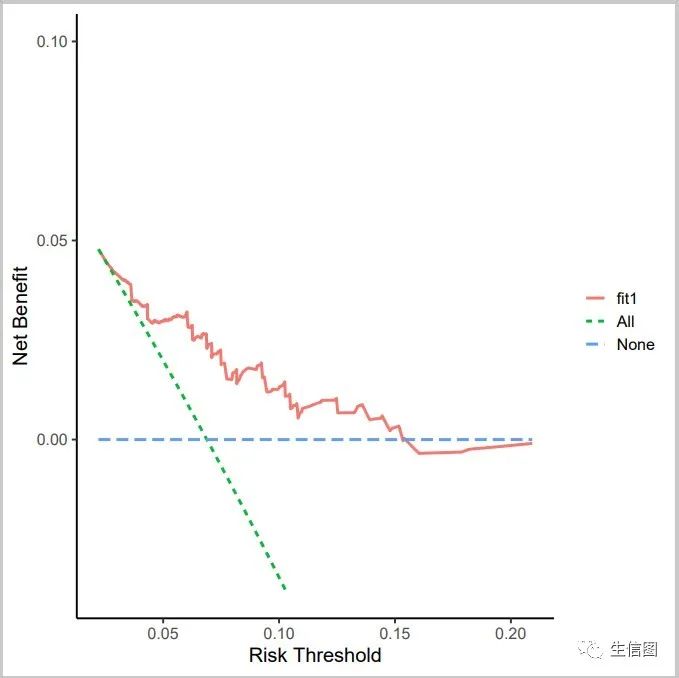
#绘制两个模型的DCA曲线plot2 <- dca(fit1,fit2,times = 365)ggplot(plot2)ggsave("dca2.pdf")
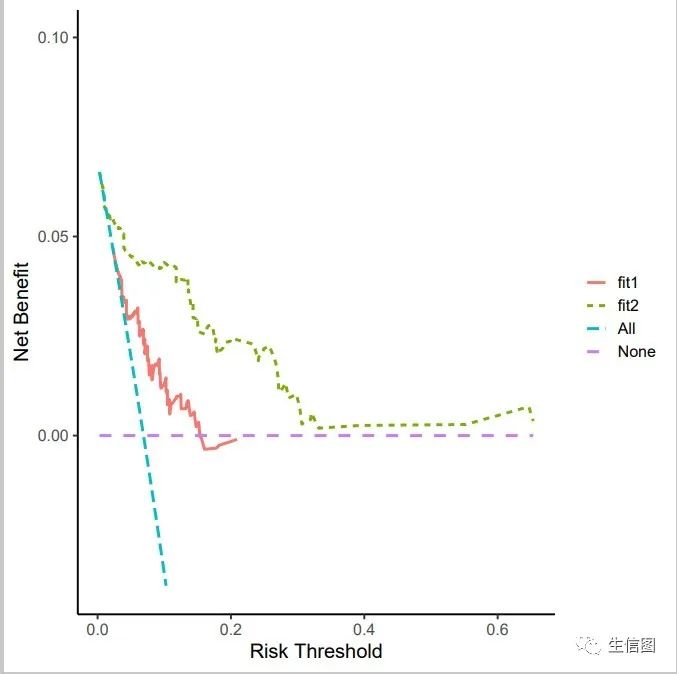
PCA分析相关其他分析内容欢迎尝试本公司新开发的云平台生物信息分析小工具,零代码完成分析,云平台网址:
http://www.biocloudservice.com/home.html
欢迎使用:云生信平台 ( http://www.biocloudservice.com/home.html)

|
往期推荐 |
|
|
|
|
|
|
👇点击阅读原文进入网址