使用renv管理项目,让R包不再冲突!
{ 点击蓝字,关注我们 }
不知道各位的小伙伴在日常的科研学习中是否遇到过这种情况呢?不同的项目需要的R包不太相同,当我们在同一台电脑的环境中运行的话R包会发生版本冲突,因为这种原因项目无法继续进行。
项目的运行,环境的配置是第一道难关,这个就相当于我们进行日常的科研实验中的实验材料,试想一下材料工具都没准备好,我们要如何继续我们的实验呢?同时,这也是众多生物信息学的研究者共同的难关,并不是说有经验的研究者就可以轻松的越过这道难关,因为这个问题出现的原因是写各种R包的作者用的环境,R语言的版本并不一样,换言之就是我们所需要的实验材料不一样呢。
但这个也并不是解决不了的事情哦,各位小伙伴只要把每个实验所需要的实验材料分门别类的分开,我们就可以避免这个问题啦~小师妹今天就想教给大家如何将我们的“实验材料”,我们的R语言环境给分开!我们今天所需要的R包便是renv!
简单的介绍一下,renv 包可帮助我们为 R 项目创建可重现的环境。使用 renv 使您的 R 项目更加独立、可移植和可重复。
隔离:为一个项目安装新的或更新的包不会破坏其他项目,反之亦然。这是因为 renv 为每个项目提供了自己的私有的library。便携式:轻松地将项目从一台计算机传输到另一台计算机,甚至跨不同平台。renv 可以轻松安装项目所依赖的软件包。可重现:renv 记录所依赖的确切软件包版本,并确保无论走到哪里,这些确切版本都是安装的版本。
小师妹在这为大家总结了一份renv的流程图~
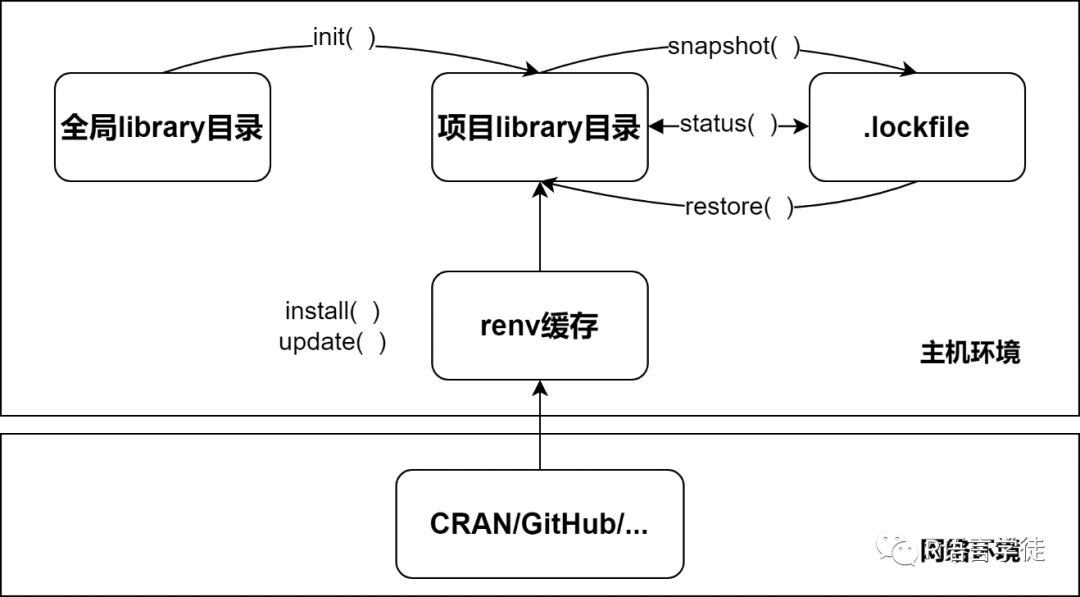
下面小师妹就开始带大家体验一下renv的基础功能啦~
首先,我们第一步还是得先安装R包
install.packages(“renv”)library(renv)
step 1
第一次使用时,需要先使用renv::init()函数,激活新的项目
env_dir <- "E:/SCP_env" # 设置项目的路径dir.create(env_dir)renv::init(project = env_dir)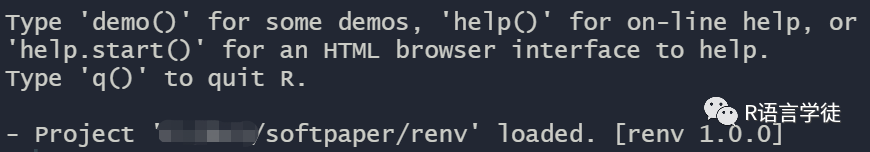
当我们的的Rstudio中出现以上提示时,就证明我们已经成功加载安装成功啦~
运行以上的代码过后,便会新建项目的library,其中会包含当前正在使用的所有R包。软件包会被记录在renv.lock文件中,且.Rprofile确保每次打开该项目时都会使用这个library。文件目录长这样哦
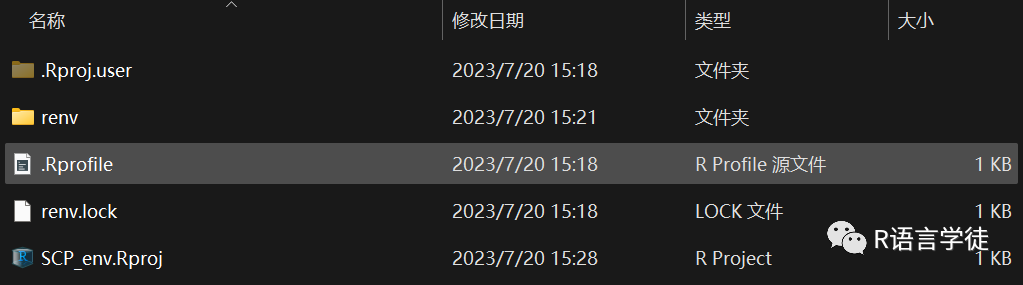
step 2
激活renv环境,当我们创建项目后我们需要继续安装和更新R包,可以使用
renv::install()or renv::update()renv::activate()renv::install()# 在我们下载好所有的R包之后,我们可以使用renv::snapshot()锁定项目中记录的包及其下载的源(CRAN/GitHub等等)renv::snapshot()

这个就是小师妹进行安装时候出现的界面,renv会帮我们储存好R包及其下载的镜像。
我们也可以观察一下下面的两个R包的路径,这样就可以直观的体验出renv的作用啦!
这一张图是使用renv包时,library的路径

而这一张是小师妹笔记本上的library路径

step 3
如果小伙伴们需要与其他人共享代码或在新环境中上运行这个的代码,可以调用 renv::restore() 来重新安装锁定文件中记录的特定软件包版本。
renv::restore()step 4
最后一步,当我们不需要使用renv啦,我们就可以使用renv::deactivate()函数关闭renv包,继续使用主体的R语言的环境哦~
renv::deactivate()小师妹为大家总结一下~renv包为大家在使用不同项目的过程中,为大家创建不同的library路径,以便于减少R包的版本冲突,替大家分开“实验材料”,也是便于后期对项目的复现哦,要知道,在计算机领域,哪怕是一样的代码,时间不一样也会导致结果的不一致哦!
今天小师妹的分享就到这里啦!大家在以后创建新的科研项目时可以用上renv这个R包,减少基础的问题,跨过环境配置这道难关呢!如果小伙伴有其他数据分析需求,可以尝试使用本公司新开发的生信分析小工具云平台,零代码完成分析,非常方便奥,云平台网址为:http://www.biocloudservice.com/home.html。
1
END
1

