手把手教你用DESeq2 进行基因表达差异分析
{ 点击蓝字,关注我们 }
clusterProfiler是一个用于生物信息学和功能富集分析的R语言包,它可以对基因集合进行功能注释和富集分析,从而帮助研究人员理解大规模基因表达数据的生物学意义。
clusterProfiler可以用于对多个物种的基因集合进行功能注释和富集分析,包括基因本体(生物过程、细胞组分、分子功能)以及基因通路(KEGG、Reactome等)。该软件包通过统计方法来确定哪些功能和通路在给定基因集合中过于富集,从而帮助用户了解这些基因的功能和调控机制。今天小师妹主要给大家介绍如何利用这个R包来进行Gene Ontology的富集分析。
01
安装加载包
首先我们需要安装并载入ClusterProfiler 包
代码:
if (!require("BiocManager", quietly = TRUE))install.packages("BiocManager")BiocManager::install("clusterProfiler")
02
载入数据
准备一个基因列表,该列表包含您感兴趣的基因。基因列表可以是基因符号、Ensembl ID或其他常见的基因识别符。例如,通过差异表达基因分析获得的显著上调/或下调基因,将这些基因名称作为输入。
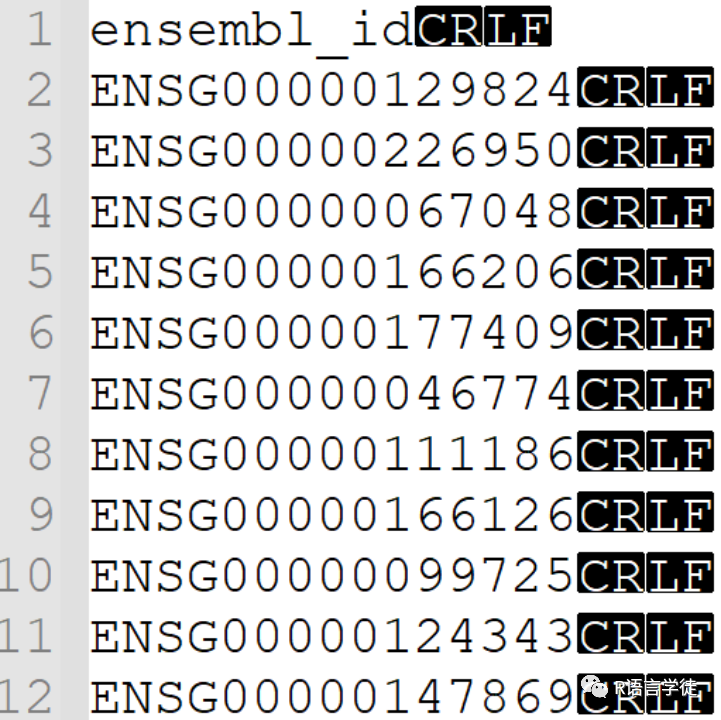
 图1 基因列表文件内容示例
图1 基因列表文件内容示例代码:
genes <- read.delim('gene.txt', header = TRUE, stringsAsFactors = FALSE)[[1]]03
进行基因注释
使用bitr函数对基因列表进行基因注释。该函数可以将基因识别符映射到GO注释信息。进行GO分析时,需要考虑的一个基础因素就是基因的GO注释信息从何处获取。Bioconductor上提供了以下19个物种的Org类型的包,包含了这些物种的GO注释信息。
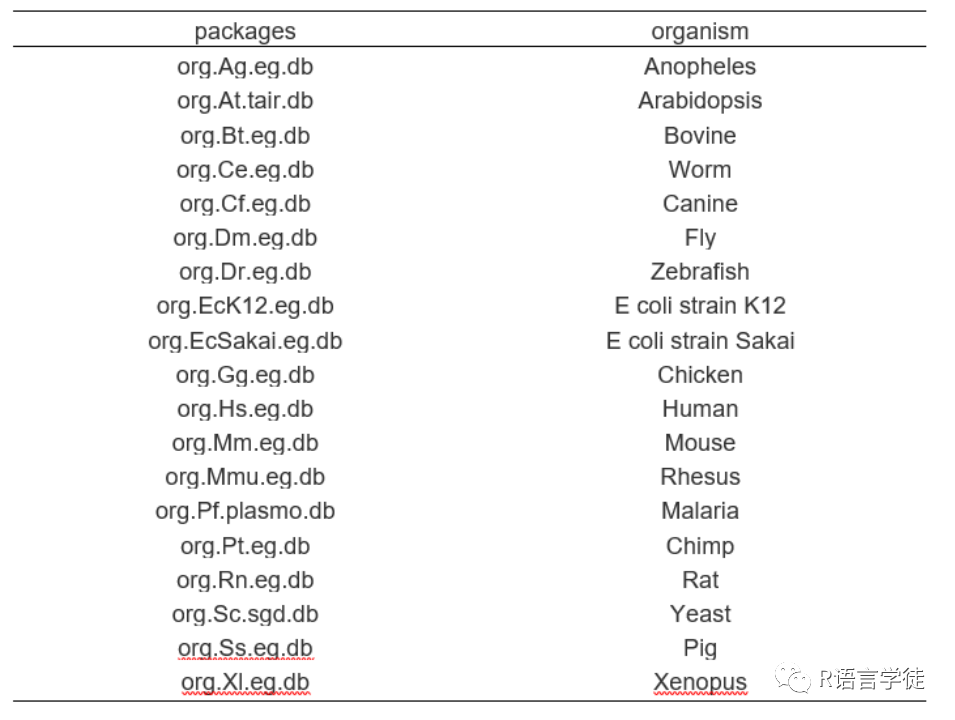
 图2 Bioconductor提供GO注释的物种与R包名称
图2 Bioconductor提供GO注释的物种与R包名称对于以上19个物种,只需要安装对应的org包,clusterProfile就会自动从中获取GO注释信息,我们只需要差异基因的列表就可以了,使用起来非常方便。
示例代码:
library(clusterProfiler)library(org.Hs.eg.db)# 将基因符号映射到GO注释(以人类为例)gene_list <- c("GENE1", "GENE2", "GENE3", ...)gene2go <- bitr(gene_list, fromType = "SYMBOL", toType = "ENTREZID", OrgDb = org.Hs.eg.db)
04
进行GO富集分析
使用enrichGO函数对注释后的基因进行GO富集分析。该函数根据基因在特定GO术语中的富集程度计算富集分数和调整的p值。您可以选择不同的统计方法和参数设置。
代码:
#GO富集分析enrich.go <- enrichGO(gene = genes, #待富集的基因列表OrgDb = 'org.Hs.eg.db', #指定物种的基因数据库,示例物种是人类keyType = 'ENSEMBL', #指定给定的基因名称类型,例如这里以 ensembl id 为例ont = 'ALL', #GO Ontology,可选 BP、MF、CC,也可以指定 ALL 同时计算 3 者pAdjustMethod = 'fdr', #指定 p 值校正方法pvalueCutoff = 0.05, #指定 p 值阈值(可指定 1 以输出全部)qvalueCutoff = 0.2, #指定 q 值阈值(可指定 1 以输出全部)readable = FALSE)#输出write.table(enrich.go, 'enrich.go.txt', sep = 't', row.names = FALSE, quote = FALSE)
在上述代码中,使用了人类的基因注释数据库(org.Hs.eg.db),设置了p值和q值的阈值。
05
结果解释和可视化
查看富集分析的结果,了解在给定基因列表中富集的GO术语。可以使用summary函数和barplot、dotplot等函数对结果进行摘要和可视化。
summary函数:summary(enrich.go)

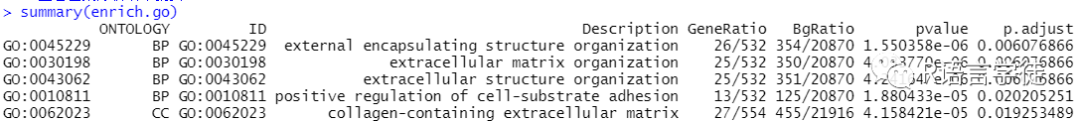 图3 查看富集分析结果摘要
图3 查看富集分析结果摘要barplot函数:barplot(enrich.go)
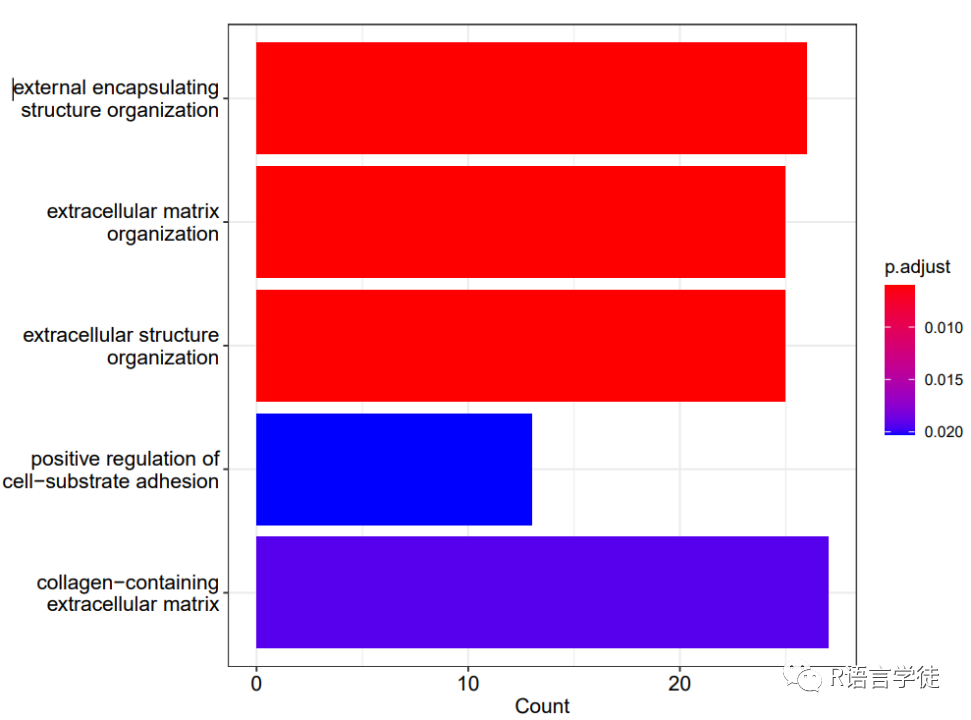
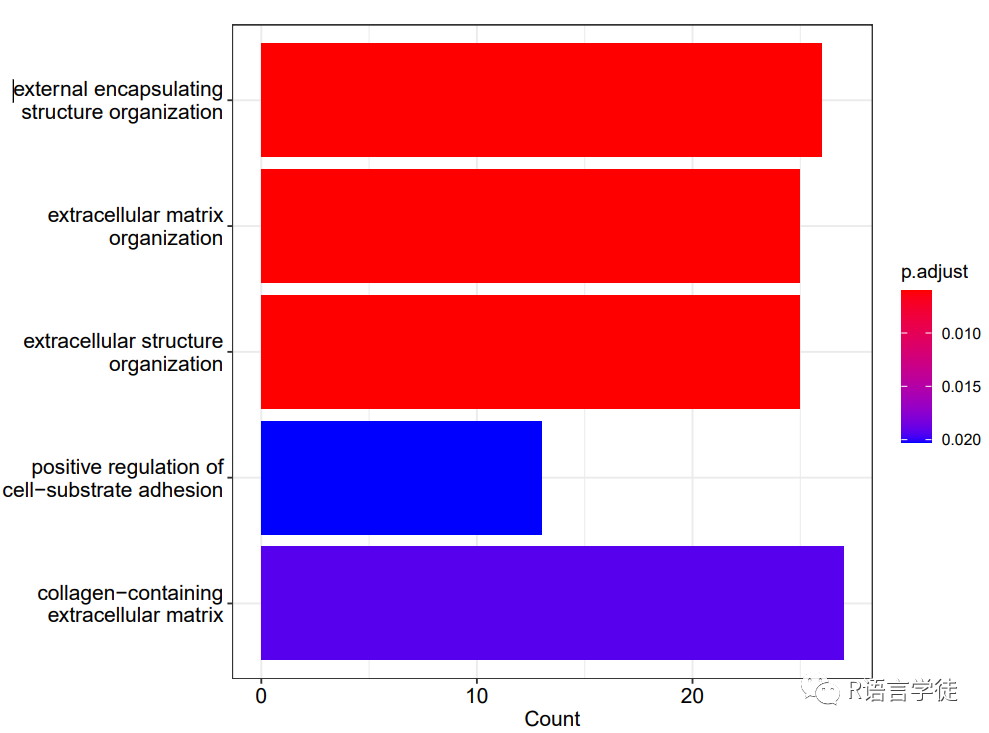 图4 富集柱形图
图4 富集柱形图dotplot函数:dotplot(enrich.go)
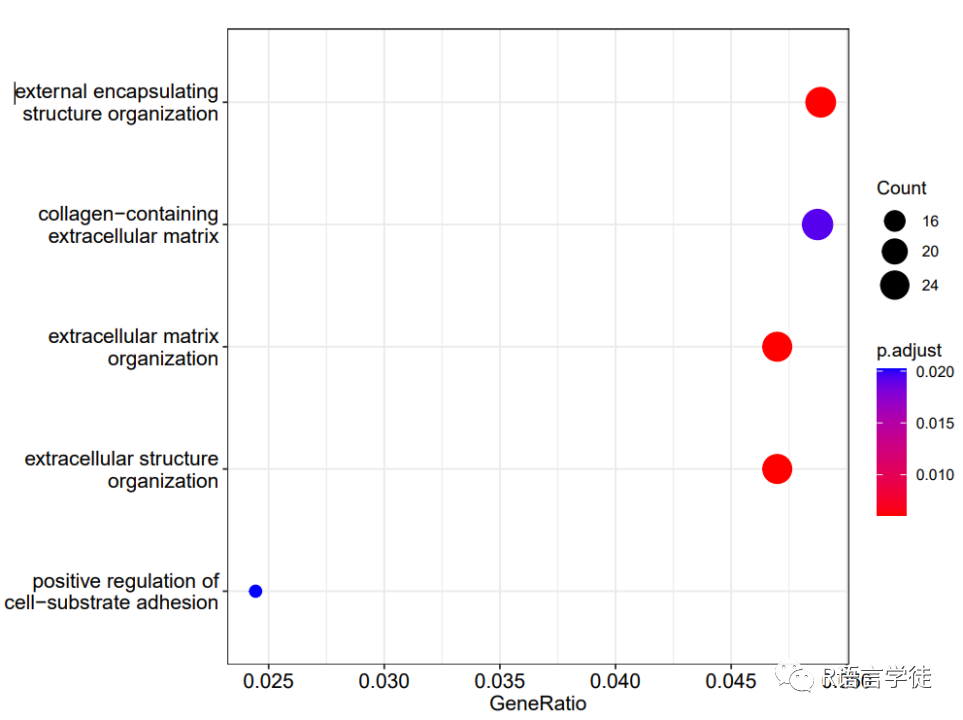
以上步骤是小师妹提供的使用clusterProfiler进行基因本体(GO)分析的一般流程,您可以根据您的具体需求和数据进行相应的调整和扩展。总之,clusterProfiler是一个强大的R语言包,可用于生物信息学和功能富集分析,提供了丰富的功能注释和富集分析工具,有助于研究人员对基因集合的生物学意义进行深入分析。更多学习资源请大家移步小师妹专属云生信平台(云生信 – 学生物信息学 (biocloudservice.com))搜索更多资源哦!
E
N
D
