「付费」【代码分享-18】oncoPredict,分析速度更快更简单的肿瘤样本药物敏感性分析R包
 今天小果带来的分享属于肿瘤生信文章必不可缺的分析内容—肿瘤样本药物敏感分析,小果今天利用oncoPredict包进行预测,相比与pRRopetic包,该包运行速度会更快,可以使用自定义的细胞系表达和药敏数据进行分析;感兴趣的小伙伴可以跟着小果一起进行实操学习。
今天小果带来的分享属于肿瘤生信文章必不可缺的分析内容—肿瘤样本药物敏感分析,小果今天利用oncoPredict包进行预测,相比与pRRopetic包,该包运行速度会更快,可以使用自定义的细胞系表达和药敏数据进行分析;感兴趣的小伙伴可以跟着小果一起进行实操学习。
1.准备需要的R包
BiocManager::install("sva")install.packages("ggplot2")install.packages("tidyverse")install.packages("cowplot")BiocManager::install("preprocessCore")#加载需要的R包library(preprocessCore)library(ridge)library(glmnet)library(car)library(tidyverse)library(impute)library(ggplot2)library(cowplot)library(sva)
2.数据读取与处理
#表达矩阵,行为基因名,列为样本名dat <- read.table("easy_input_expr.txt",sep = "t",row.names = 1,header = T,stringsAsFactors = F,check.names = F)
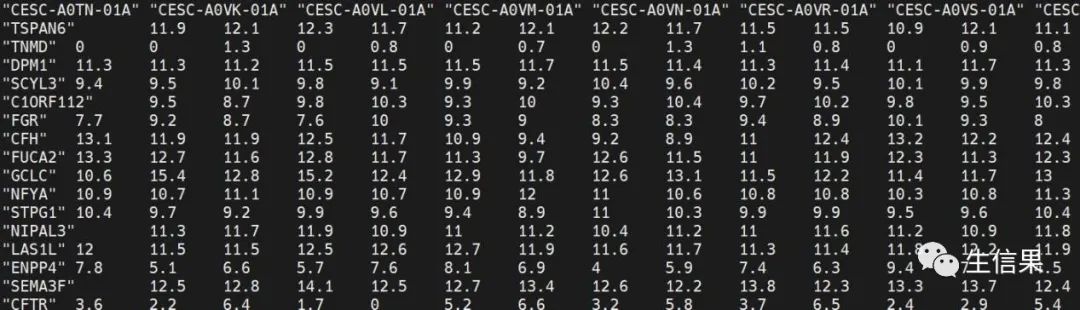
#分组信息,行为样本名,列为分组信息ann <- read.table("easy_input_anno.txt",sep = "t",row.names = 1,header = T,stringsAsFactors = F,check.names = F)

# 构建测试集数据testingExprData <- as.matrix(dat[,rownames(ann)])# 细胞系表达矩阵# 数据类型为矩阵,细胞系表达是行为基因,列为细胞系,入值为表达谱的矩阵,表达谱需做log2标准化trainingExprData <- readRDS(file='GDSC2_Expr_short.rds')# 细胞系对应药敏数据# 数据类型为矩阵,细胞系药敏是行为细胞系,列为药物,入值为药物敏感性的矩阵,数据一般存在NA,需要过滤或进行填补trainingPtype <- readRDS(file = "GDSC2_Res.rds") # 数据类型trainingPtype <- trainingPtype[,apply(trainingPtype, 2, function(x) {sum(is.na(x)) < 0.2 * nrow(trainingPtype)})] # 去除在超过20%样本中都缺失的药物trainingPtype <- trainingPtype[apply(trainingPtype, 1, function(x) {sum(is.na(x)) < 0.2 * ncol(trainingPtype)}),] # 去除在超过20%药物中都缺失的样本trainingPtype <- t(impute.knn(t(trainingPtype))$data) # KNN填补缺失值trainingPtype <- exp(trainingPtype) # 这里根据数据
3.结果文件
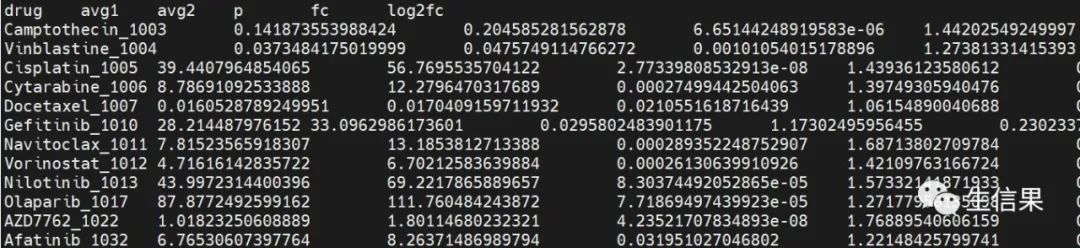
3.1 output_wilcox_test_for_potential_drugs.txt
该结果为计算的不同分组中药物敏感的显著差异分析结果文件。
3.2 boxplot_of_predicted_drug_sensitivity.pdf
该结果图片为绘制的不同分组中显著差异的药物敏感箱线图

最终小果利用oncoPredict包成功的预测了肿瘤样本药物敏感性,欢迎大家和小果一起讨论学习呀!今天小果的分享就到这里,下期在见奥。

往期推荐
如果需要完整的代码可以点击付费获取哦!今天小果的分享就到这里,如果小伙伴有其他数据分析需求,可以尝试本公司新开发的生信分析小工具云平台,季代码完成分析,非常方便奥!
(扫码领取正好的输入文件,代码文件及示例结果)