预处理,降维和聚类分析RNA测序数据的好帮手 — R语言包scran

R语言在生物信息学和生物统计学中被广泛应用,有许多优秀的软件包可以帮助研究人员处理和分析生物学数据。其中一个重要的R语言包就是”scran”,它是一个专为单细胞RNA测序数据分析而设计的工具。“scran”包提供了一系列强大的功能,用于预处理、降维和聚类分析,有助于研究人员更好地理解细胞间的差异和相似性。
单细胞RNA测序技术的出现在生物学领域掀起了一股革命,它为研究人员提供了窥视单个细胞内部基因表达的机会。通过单细胞RNA测序,我们可以深入研究组织和器官中每一个微小的细胞,从而揭示细胞类型、状态和功能之间微妙的差异。然而,这种技术也带来了巨大的数据挑战,因为从每个细胞中获取的基因表达数据呈现高维性和噪声。
正是在这种背景下,”scran”包崭露头角,成为处理和解释单细胞RNA测序数据的不可或缺的工具。这个包在数据处理方面表现出色,通过支持高级的基因表达数据预处理,例如过滤低表达基因和校正技术偏差,有助于减少数据中的噪声,提高数据质量。这使得后续的分析更准确,更具有可解释性,为细胞内部的基因表达模式提供了更清晰的图景。
“scran”的内建降维技术如PCA和t-SNE,充分利用了数学和统计学的原理,将高维的基因表达数据转化为二维或三维的表示。这个降维过程不仅便于数据的可视化,也为聚类和细胞亚型的划分提供了基础。而聚类功能,正是”scran”包的又一亮点。它可以将表达模式相似的细胞归为一类,帮助研究人员发现不同细胞类型和状态。此外,对批次效应的校正功能也使得数据更具可比性,为不同样本之间的差异提供了更准确的评估。
公众号后台回复“111”领取代码,代码编号:231105
也就是说,”scran”包不仅填补了单细胞RNA测序数据分析工具的空白,更成为生物学家们揭示细胞间差异和功能的得力助手。这个包的多样功能和强大性能,无疑为单细胞RNA测序技术的应用拓宽了道路,推动了生物学领域的前沿研究。
要使用scran包,可以在R中使用以下命令进行安装和加载:
> install.packages("scran") #安装scran语言包> library(scran) #加载语言包
首先,“scran”包提供了用于数据预处理的函数,如去除低表达基因和高度变异基因,从而减少噪声的影响,提高数据质量。接着,“scran”包利用了高级降维技术,如主成分分析(PCA)和t分布随机邻域嵌入(t-SNE),将高维数据映射到低维空间,使得数据更易于可视化和分析。这有助于研究人员发现细胞之间的模式和聚类。
另一个“scran”包的关键功能是聚类分析。它提供了多种聚类方法,如层次聚类和K均值聚类,可以将细胞分成不同的群集,每个群集代表一个细胞类型或状态。这有助于确定组织或器官中不同细胞种类的存在以及它们之间的相对比例。
示例:
当使用scran语言包时,一个简单的示例可以是对单细胞RNA测序数据的预处理、降维和聚类分析。首先,你需要安装并加载scran包以及其他所需的包。
> install.packages("scran")> library(scran)# 假设你已经有一个名为'sce'的SingleCellExperiment对象# 这里假设'sce'对象已经包含了基因表达数据和样本信息
使用scran包的函数对数据进行预处理,比如去除低表达基因和高度变异基因,以及归一化处理。
# 过滤低表达基因和高度变异基因> sce <- quickCluster(sce)# 归一化处理> sce <- computeSumFactors(sce)> sce <- logNormCounts(sce)
利用PCA(主成分分析)将高维数据降维,以便进行可视化。
# 执行PCA降维> sce <- runPCA(sce)
绘制PCA降维后的数据分布,可以用散点图展示不同细胞类型或样本之间的差异。
# 绘制PCA图> plotPCA(sce, colour_by = "cell_type")
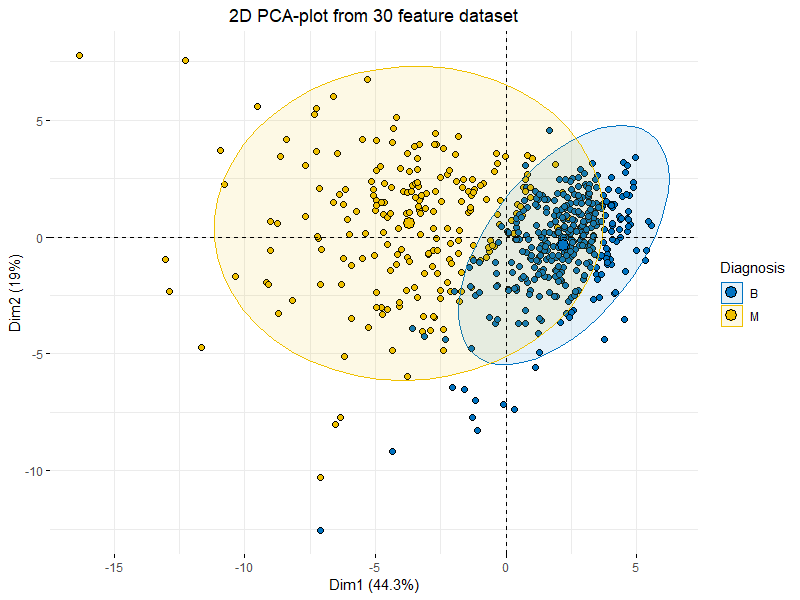
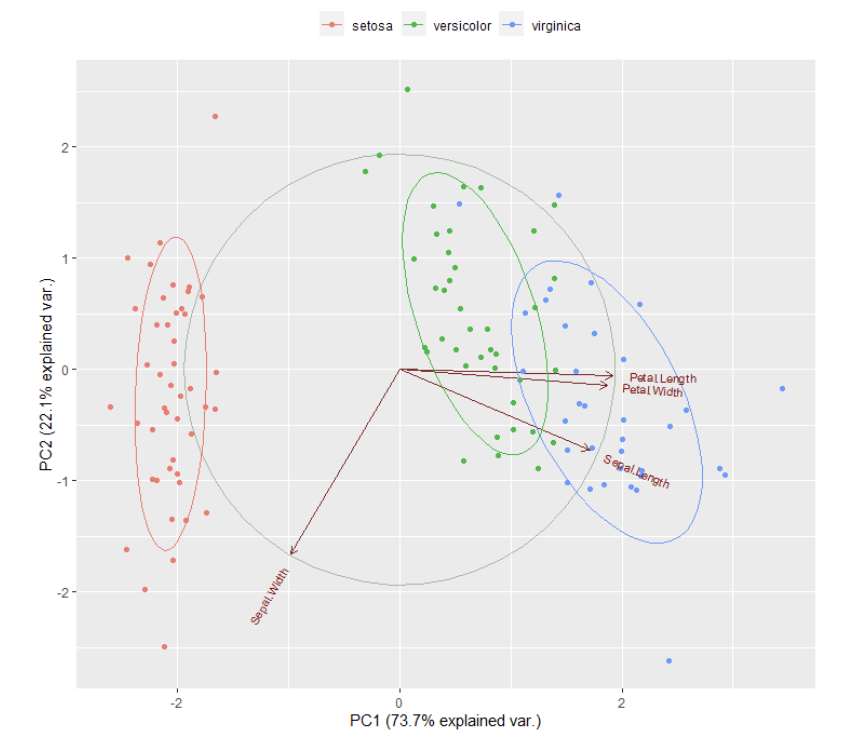
使用scran包中的聚类函数,将细胞分成不同的群集。
# 进行聚类分析,假设你要分为3个聚类> sce <- buildSNNGraph(sce, use.dimred = "PCA")> sce <- clusterCells(sce, method = "igraph", k = 3)
绘制聚类结果,将不同聚类用不同颜色标注在降维图上。
# 绘制带聚类标签的PCA图> plotPCA(sce, colour_by = "cluster")
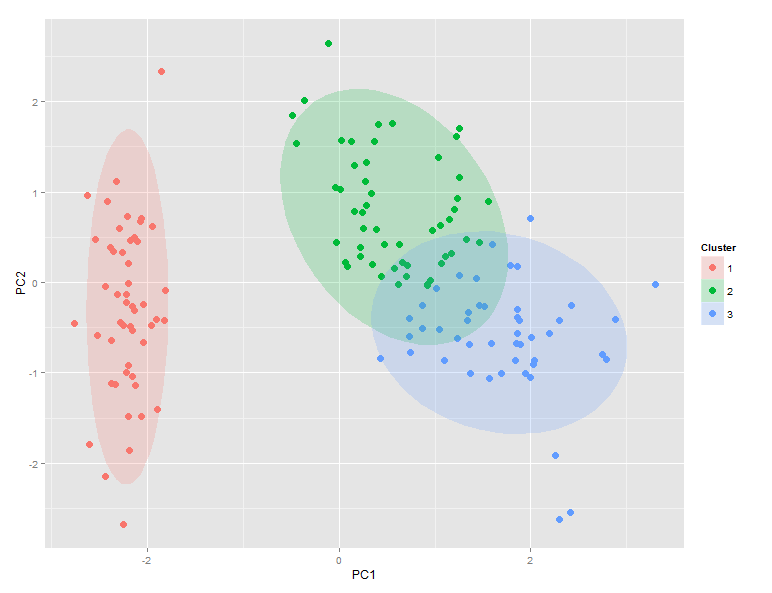
通过这个简单的示例,可以使用scran包对单细胞RNA测序数据进行预处理、降维和聚类分析,并在降维图上可视化不同细胞类型或聚类的分布情况。这有助于更好地理解数据中的细胞多样性和差异。
此外,”scran”包还具备在单细胞RNA测序数据分析中至关重要的批次效应校正能力。在单细胞RNA测序实验中,数据可能来自不同的实验批次,甚至来自不同的实验室或测序平台,这可能导致技术性的偏差。这些批次效应会对数据的结果产生显著影响,可能掩盖或误导研究人员对细胞间真实生物学差异的理解。
批次效应校正是一个关键步骤,旨在消除不同批次之间的技术偏差,使得数据更具有一致性和可比性。”scran”包通过提供多种方法,赋予了研究人员处理批次效应的灵活性,以适应不同数据集和研究设计的需求。这些方法可以根据数据的特点自动或手动调整参数,从而使得校正过程更加精准和准确。
通过应用”scran”包的批次效应校正功能,研究人员可以消除数据中的批次偏差,从而得到更准确、更可靠的分析结果。这为不同实验条件下细胞的比较和细胞亚型的发现提供了更有力的支持。同时,这也为跨实验室和跨平台的数据整合提供了便利,使得数据的可比性得以维持。
以上就是对R语言包scran的简单介绍啦,”scran”包在生物学研究中发挥着重要作用,特别是在单细胞RNA测序数据的分析领域。它为研究人员提供了强大的工具,用于数据预处理、降维、聚类和批次效应校正,帮助他们更好地理解细胞的多样性和功能。通过“scran”包,研究人员能够从海量的单细胞基因表达数据中提取有价值的信息,推动生物学领域的深入研究。
小伙伴们,今天有没有学到新知识呢,想要继续了解R语言内容可以持续关注小果哦~
往期推荐
