小果震惊!利用PyMol比对蛋白质三维结构原来如此简单!
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

PyMol是一款强大的分子可视化工具,可用于分子结构的可视化、编辑、分析和比对等方面。其功能包括:分子可视化、电子密度图、分子对接、分子编辑、分子动力学模拟、分子结构比对和脚本编写等。
上一期,小果教大家安装了PyMol,本期小果就带大家一起学习用PyMol进行蛋白质结构,一些基础操作可以查看官网给出的教程(start [PyMOL Documentation])
一、在使用之前我们需要下载蛋白结构,以下是几个常用的数据库:
1.Protein Data Bank (PDB) (www.rcsb.org):是世界上最大的蛋白质结构数据库之一,包含数万个蛋白质结构的三维坐标数据。
2.Protein Data Bank Japan (PDBj) (https://pdbj.org/?lang=zh-CN):是日本的蛋白质结构数据库,提供了与PDB类似的蛋白质结构数据和搜索功能。
3.Worldwide Protein Data Bank (wwPDB) (www.wwpdb.org):wwPDB是一个由PDB、PDBj和欧洲蛋白质数据银行(PDBe)组成的联合数据库,提供了全球范围内的蛋白质结构数据和搜索功能。
4.Protein Model Portal(www.proteinmodelportal.org):是一个综合性的蛋白质结构数据库,包含了来自PDB和其他数据库的数千个蛋白质结构模型。
5.SWISS-MODEL Repository(SWISS-MODEL Repository (expasy.org)):SWISS-MODEL Repository是一个蛋白质结构模型数据库,包含了来自SWISS-MODEL和其他来源的数百万个结构模型。
二、小果下载了两个肌红蛋白(myoglobin)的pdb文件。
三、下载成功后我们在PyMol打开,可以在展示窗口看到蛋白的三维结构
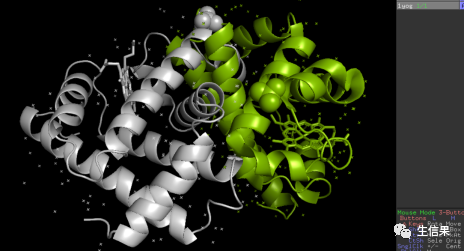
四、在Pymol命令行中输入以下命令以选择需要对齐的部分,为了更直观,可以在最右侧C栏改变蛋白展示出的颜色。Name是你文件的名称
align name1, name2如图,两个蛋白重叠在了一起,因为都是肌红蛋白所以重叠度很高,我们可以放大查看细节。
再打开一个蛋白结构

输入align name3, name1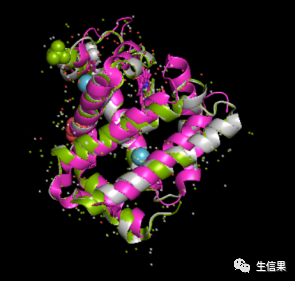
可以看到三个蛋白都重叠在了一起,通过点击右侧栏的蛋白名称可以隐藏其中某个或某几个蛋白
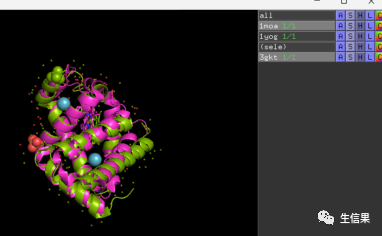
是不是很简单呢~
如果有生信分析的需要可以看看我们推出的生信云平台哦
网址:云生信 – 学生物信息学 (biocloudservice.com)
在这之后还有非常多进阶且美观的代码,小果的笔记本有所限制就不一一展示啦,把代码展示给大家,后续的结果图小果会打包成压缩文件夹在文章结尾分享给大家哦!
今天的分享就到这啦~小果希望大家也可以像学习popsicleR一样,先将代码运行成功,然后有能力的同学可以可以研究一下源代码,进行深层次的学习哦!
如果小伙伴有其他数据分析需求,可以尝试使用本公司新开发的生信分析小工具云平台,零代码完成分析,非常方便奥,云平台网址为:(http://www.biocloudservice.com/home.html),其中也包括了通路表达分析(http://www.biocloudservice.com/313/313.php),单细胞的基因共表达分析(http://www.biocloudservice.com/906/906.php)等各种小工具哦~,有兴趣的小伙伴可以登录网站进行了解。
点击“阅读原文”立刻拥有
↓↓↓