保姆级教程!小果教你学习曼哈顿图!Manhattan图!
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

有的小伙伴可能听说过曼哈顿图,但是并不了解,也不知道是来做什么的,其实,曼哈顿因为其形似曼哈顿摩天大楼,所以被称为曼哈顿图。本质上就是散点图,我们一般用来展示大量非零的波动数据。Y轴高点是用来显示具有相关性的位点。平时我们用的最多的就是全基因组关联分析研究中也就是GWAS,因为曼哈顿图可以一目了然的查看基因的频率和分布。
小伙伴是不是了解了很多,下面小果就带大家去学习一下如何绘制曼哈顿图。
曼哈顿图绘制方法有很多种,今天就带大家了解一下:
install.packages(‘qqman’)
library(qqman)#这里主要用到qqman这个R包,这个很好下载,普通方式就可以下载。
这里我们使用R包qqman自带的实例数据集qwas 去为大家展示,
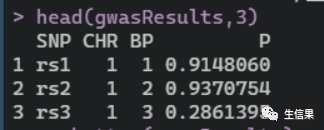
小伙伴可以按照这个形式去设置自己的数据
其中SNP是snp名称,CHR是染色体的编号,BP是碱基的位置,P就是P值啦
我们先绘制简单的曼哈顿图manhattan:
manhattan(gwasResults, col = c("blue4", "orange3"), main = "Results from simulated trait",genomewideline = FALSE, suggestiveline = FALSE,highlight = snpsOfInterest[1:10])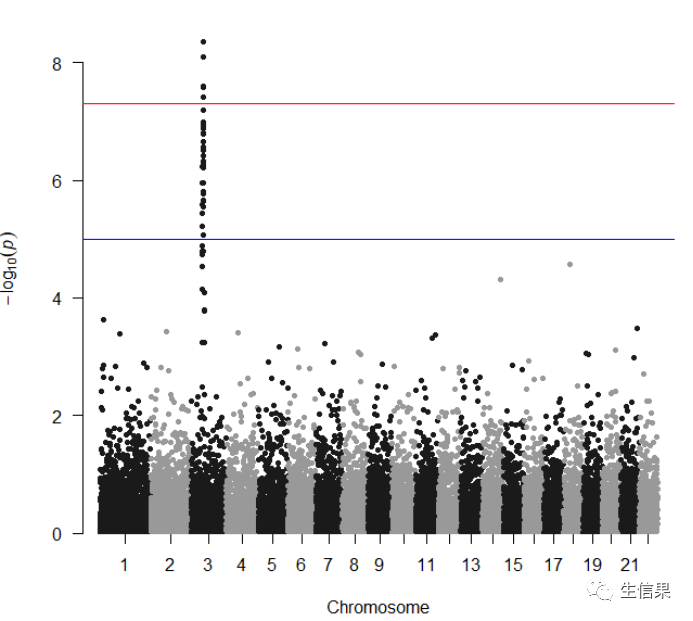
但是这个图看起来平平无奇,我们加点细节,加入标题,调整颜色,把部分SNP高亮等一些细节。
head(snpOfInterest) #这里我们先查看内置高亮snp数据, snpOfInterest可自行设置
manhattan(gwasResults, col = c("blue4", "orange3"), main = "Results from simulated trait",genomewideline = FALSE, suggestiveline = FALSE,highlight = snpsOfInterest[1:10])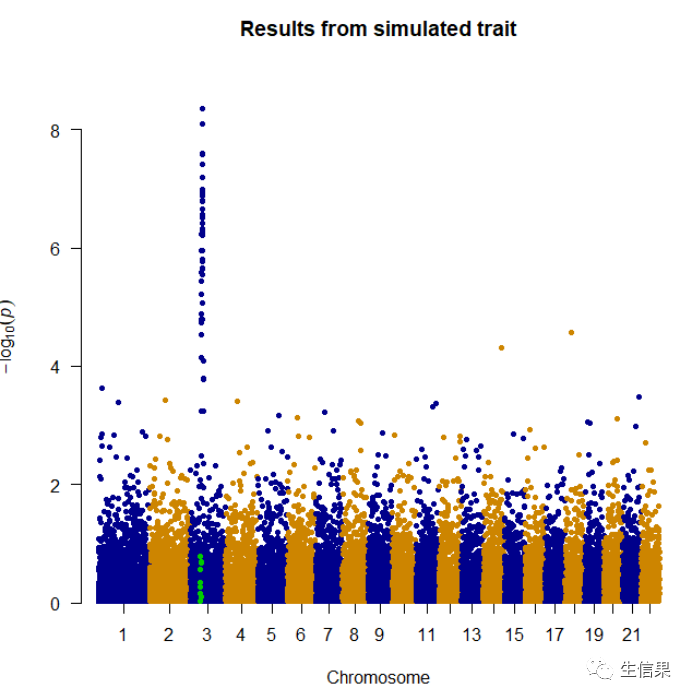
里面参数 CHR3的绿点是来自snpsOflnterest,highlight参数控制。
蓝色横线是由参数suggestiveline控制,红色使用genomewideline控制
小伙伴可以自信设置。
我们还可以把基因名加上去,这里我们去教大家如何批量表示基因名:
gwasResults[3057,1] <- “AA” #这里将将最显著的点,自定义,可看出改变
manhattan(gwasResults,suggestiveline =FALSE,genomewideline =FALSE,col=c("#FF6A6A","#43CD80","#EE7600"),annotatePval=0.05,annotateTop=TRUE)
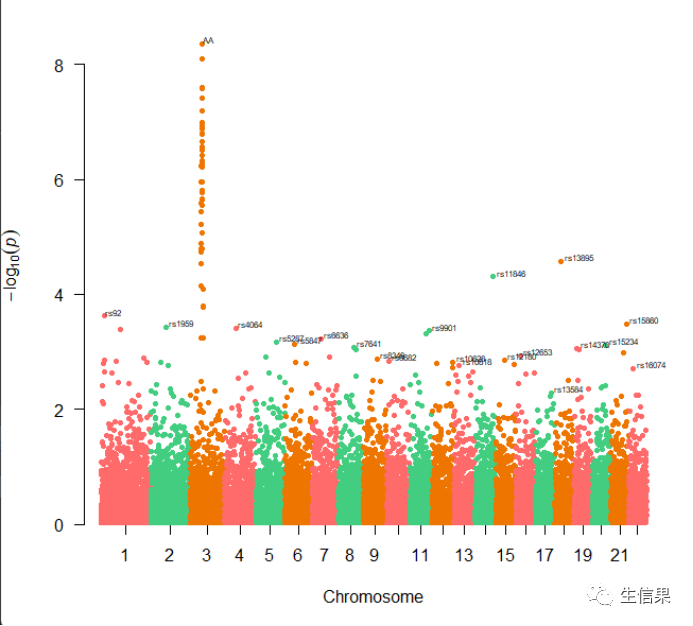
这样就绘制完成啦,上述代码中,annotatePval可以设置p阈值,低于该值的散点将会在图中被标记,annotateTop默认为True,即仅标记p值最小的点,所以该图中各条染色体只有一个snp被标记,倘若annotateTop设置为False,则所有低于annotatePval的点均会被标记。
上述是使用qqman包绘制曼哈顿图,小果这里还有一种方法,CMplot包绘制曼哈顿图,
这个包参数会更多,而且图会更好看。并且可以绘制多表型的曼哈顿图,以及circos状展示。是不是很心动呢?
library(CMplot)#我们首先载入这个包CMplot(pig60K,plot.type="m",LOG10=TRUE,threshold=NULL,chr.den.col=NULL,file="jpg",dpi=300)这里使用内部的数据集pig60K,小伙伴根据数据集形式自行去设置:
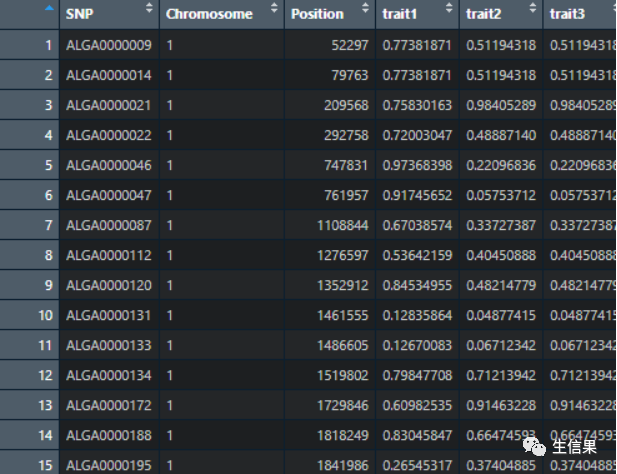
这个图绘制完会会出现在设置好的文件夹里,来看看效果:
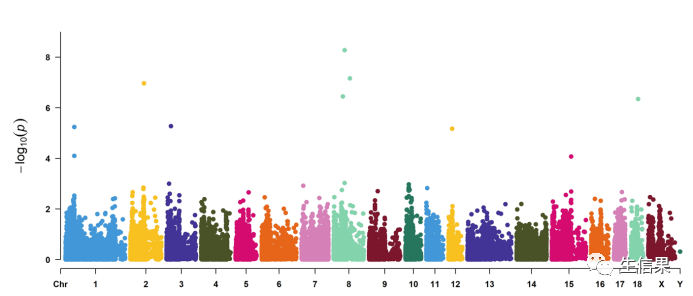
我们绘制SNP的密度图:
CMplot(pig60K, plot.type="m", LOG10=TRUE, ylim=NULL, threshold=c(1e-6,1e-4),threshold.lty=c(1,2), threshold.lwd=c(1,1), threshold.col=c("black","grey"),amplify=TRUE,chr.den.col=c("darkgreen","yellow","red"),bin.size=1e6,signal.col=c("red","green"),signal.cex=c(1,1),signal.pch=c(19,19),file="jpg",dpi=300)
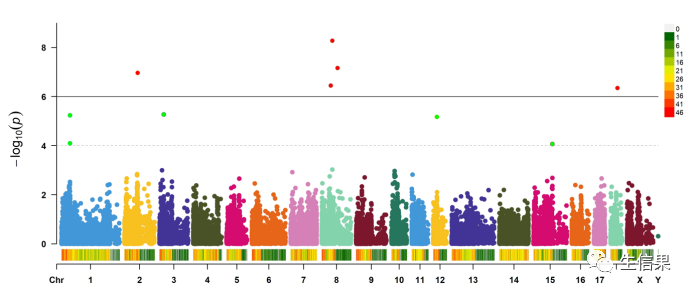
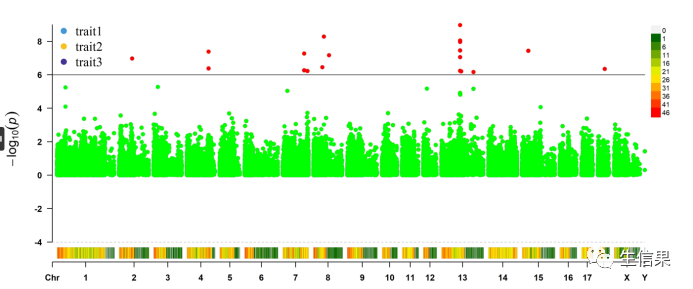
接下来我们还可以对多表型的数据展示:
CMplot(pig60K, plot.type="m", multracks=TRUE, threshold=c(1e-6,1e4),threshold.lty=c(1,2),threshold.lwd=c(1,1), threshold.col=c("black","grey"), amplify=TRUE,bin.size=1e6,chr.den.col=c("darkgreen", "yellow", "red"), signal.col=c("red","green"),signal.cex=c(1,1),file="jpg",memo="",dpi=300)
我们还可以绘制圈图也就是circos展示
CMplot(pig60K,plot.type="c",chr.labels=paste("Chr",c(1:18,"X"),sep=""),r=0.4,cir.legend=TRUE,outward=FALSE,cir.legend.col="black",cir.chr.h=1.3,chr.den.col="black",file="jpg",dpi=300)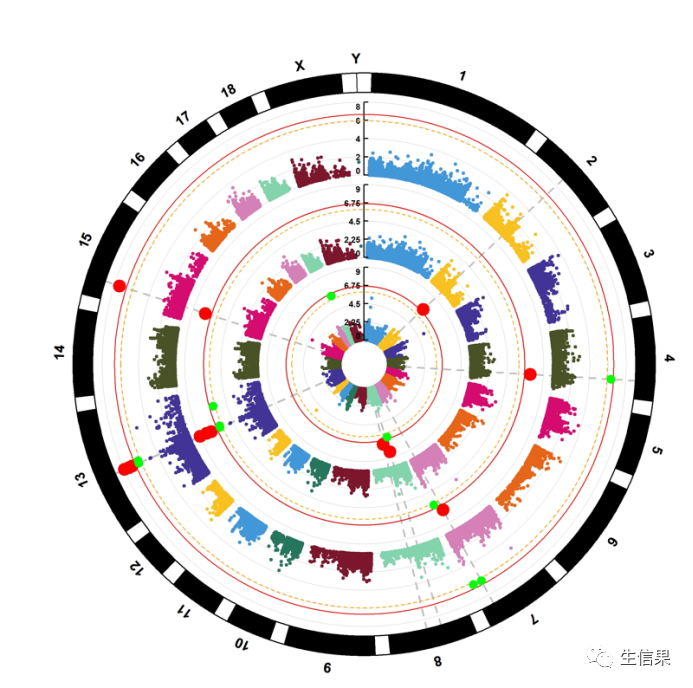
上述都是使用自带的示例数据pig60K,小果还是喜欢circos的图片,图片好看精美。
上述就是我们对曼哈顿的学习,小伙伴有没有学会呢,去准备一下自己的示例数据,绘制好看的曼哈顿图。小果要提醒大家,要多多理解代码参数的意义,上述小果已经标注了很多,小伙伴们可以根据自己数据内容套用进去,但是也不要忘记是什么含义,这样才能绘制出自己想要的曼哈顿图!
下期将为你带来更多R语言的骚操作技巧,以下推荐的是一个多功能的生信平台。
云生信平台链接:http://www.biocloudservice.com/home.html。【点击文末阅读原文查看哦】
其他相关分析内容,例如预测肿瘤样本药物敏感性分析(http://www.biocloudservice.com/712/712.php),预测某样本亚型对免疫治疗的反应(http://www.biocloudservice.com/292/292.php),单样本富集算法分析免疫浸润丰度(http://www.biocloudservice.com/106/106.php),计算64种免疫细胞相对含量(http://www.biocloudservice.com/107/107.php)等都可以用本公司新开发的零代码云平台生信分析小工具,一键完成该分析奥,感兴趣的小伙伴欢迎来尝试奥,网址:http://www.biocloudservice.com/home.html。今天小果的分享就到这里,下期在见奥。
小果友情推荐
好用又免费的工具安利



点击“阅读原文”立刻拥有
↓↓↓