Seurat RunPCA报错?小果教大家,遇到报错不要怕
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

-
各位小伙伴们,大家好呀,我是你们的老朋友小果,最近小果在分析单细胞数据时遇到了一个问题,在这里和大家分享一下呀。
首先小果从10x Genomics支持网站上的一个公开数据集下载FASTQ文件。这是来自人类外周血单个核细胞(PBMC)的1,000个PBMC数据集,包括淋巴细胞(T细胞、B细胞和NK杀伤细胞)和单核细胞。
下载代码为:
wget https://cf.10xgenomics.com/samples/cell-exp/3.0.0/pbmc_1k_v3/pbmc_1k_v3_fastqs.tar此数据集大小为5.17G,不是很大,大概几分钟就可以下载完成喽。
之后对此压缩文件进行解压。因为这是一个tar文件而不是tar.gz文件,所以不需要使用-z参数来提取它。
代码:
tar -xvf pbmc_1k_v3_fastqs.tar使用cellranger进行定量后,得到三个文件夹如下所示:

小果以此三个文件夹作为输入文件,进行seurat的分析。
在进行下游分析时遇到一个问题,数据缩放之后进行PCA线性降维,代码如下,出现下图的错误。
#数据缩放all.genes <- rownames(scdata)scdata <- ScaleData(scdata, features = all.genes)#降维scdatascdata <- RunPCA(scdata, features = VariableFeatures(object = scdata))DimPlot(scdata, reduction = "pca")
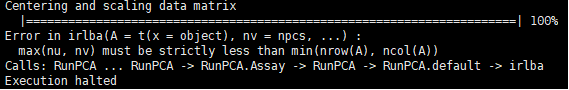
我们可以发现,主要问题是RunPCA过程出现错误,导致流程中断。发现报错提示:最大的npcs必须严格遵循小于最小行列数的规则。
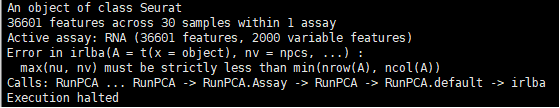
npcs必须要小于最小行列数,默认情况下npcs = 50,scdata查看矩阵行列数,发现最小值为30,将其改为29后,可以成功运行。
这边小果总结一下:
1.在学习某个软件或者某个包的使用时,我们可以详细查看每一个函数的使用手册,了解函数的原理、功能以及每个参数的作用,这样在我们之后实际操作过程中也比较方便快捷。
2.在遇到问题时,我们要仔细查看报错内容,根据报错提示找寻答案。
在做数据分析时,不可避免的会遇到降维分析,在这里小果给大家推荐一个做降维分析非常方便的小工具:PCA-UMAP和tSNE降维分析(http://www.biocloudservice.com/794/794.php),可以在线网页运行,运行速度很快,大家可以用起来哦。
以后大家遇到问题,也欢迎来和小果讨论呀。

微信号 | 18502195490
知乎 | 生信果
点击“阅读原文”立刻拥有
↓↓↓