一键解锁单细胞空间转录组可视化的神奇技巧!
{ 点击蓝字,关注我们 }
单细胞空间转录组(single-cell spatial transcriptomics)是一种新兴的单细胞RNA测序技术,它可以将细胞的RNA分布信息与其形态和位置信息相结合,实现对细胞在空间上的定位和分析。传统的单细胞RNA测序技术只能获得每个细胞的RNA测序数据,而无法获取它们在组织中的位置信息。而单细胞空间转录组技术则可以通过空间成像技术(如光学或质谱成像)将细胞的RNA分布信息映射到组织结构上,实现对单个细胞在组织或器官中的位置和空间关系的精确定位和分析。
对单细胞空间转录进行可视化可以帮助我们更好地理解细胞在组织结构中的分布、类型和功能等信息。单细胞空间转录技术可以提供每个细胞在组织结构中的位置和RNA表达信息,而可视化技术可以将这些信息以图形化的方式呈现出来,使得数据更加直观、易于理解和解释。通过绘制细胞在空间上的分布图,我们可以了解细胞在组织结构中的分布情况,探究细胞类型和功能在组织结构中的空间分布模式,以及不同细胞类型之间的空间关系,还可以比较不同基因在不同细胞类型和位置上的表达模式。
今天小师妹使用的是SeuratData中的数据。SeuratData是Seurat软件包提供的一个数据集仓库,包含了许多公开的单细胞RNA测序数据集。SeuratData可以通过Seurat软件包中的函数进行下载和使用,也可以通过SeuratData包进行访问。SeuratData包提供了一个简单的接口,可以通过函数调用快速加载SeuratData中的数据集,并进行数据探索、可视化和分析。此外,SeuratData还提供了一些示例代码和教程,帮助用户了解Seurat软件包的各种功能,从而更好地应用于自己的研究中。
01
#导入需要的R包
library(ggplot2)library(patchwork)library(dplyr)library(SeuratData)#导入数据集InstallData("stxBrain")df <- LoadData("stxBrain", type = "anterior1")
通过绘制单个细胞在空间中检测到的RNA分子数的小提琴图,观察数据分布情况。
#绘制关于nCount_Spatial的小提琴图#nCount_Spatial:单个细胞在空间中检测到的RNA分子数VlnPlot(df, features = "nCount_Spatial", pt.size = 0.1,cols ="#56B6A0" ) + NoLegend()
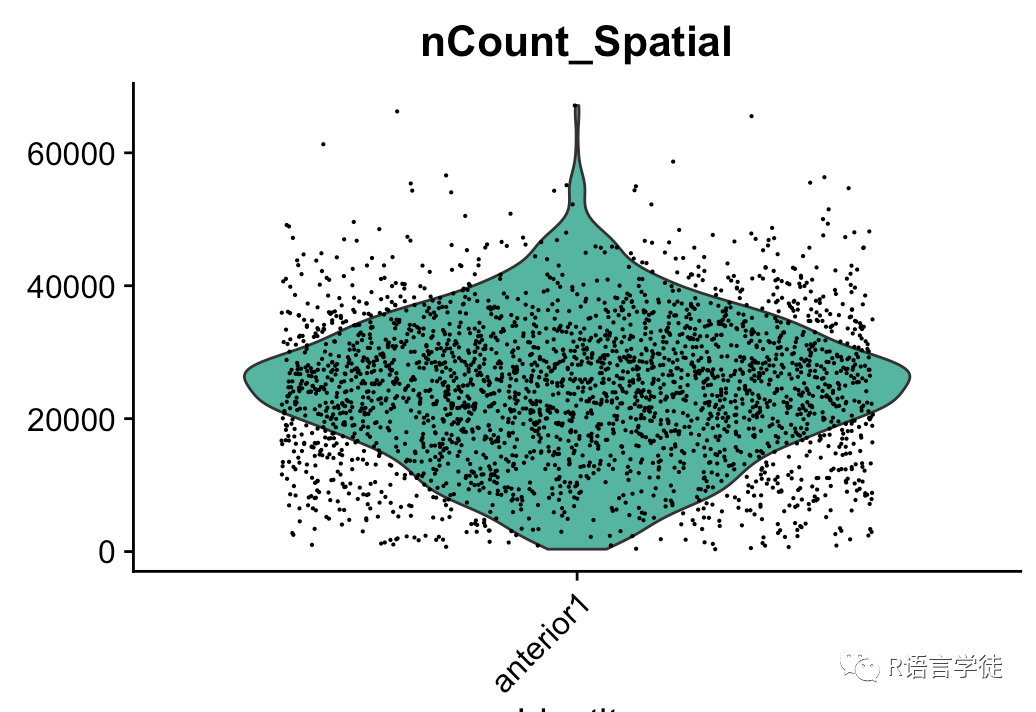
可以看到数据非常分散,需要进行标准化。这里小师妹采用的是SCTransform进行标准化。SCTransform是一个用于单细胞RNA测序数据的批次校正和基因表达值变换的函数,它可以通过消除批次效应和技术噪声,提高不同样本之间的可比性和准确性,从而更好地挖掘单细胞数据中的生物学信息。
#构建基因表达的正则化负二项式模型df <- SCTransform(df, assay = "Spatial", verbose = FALSE)
接下来就可以使用SpatialFeaturePlot函数绘制单细胞数据集中每个细胞在空间中的分布图,其中features参数表示需要绘制的特征。SpatialFeaturePlot可以将单细胞空间转录数据中的位置信息与RNA表达信息相结合,将每个细胞在组织结构中的位置和RNA表达信息以散点图或热图的方式呈现出来,方便我们直观地观察和比较不同细胞类型的空间分布模式和基因表达情况。
#SpatialFeaturePlot函数用于绘制单细胞数据集中每个细胞在空间中的分布图SpatialFeaturePlot(df, features = c("Hpca", "Ttr"))

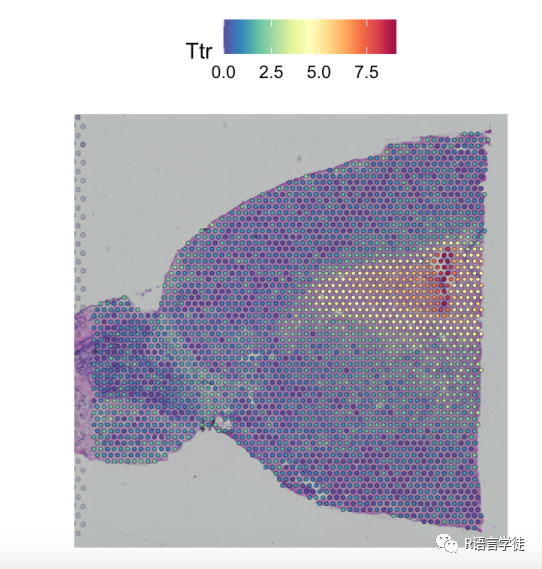
还可以调整斑点的大小、透明度等提高组织图像的可视化效果
#调整斑点的大小和透明度SpatialFeaturePlot(df, features = "Ttr", pt.size.factor = 1)
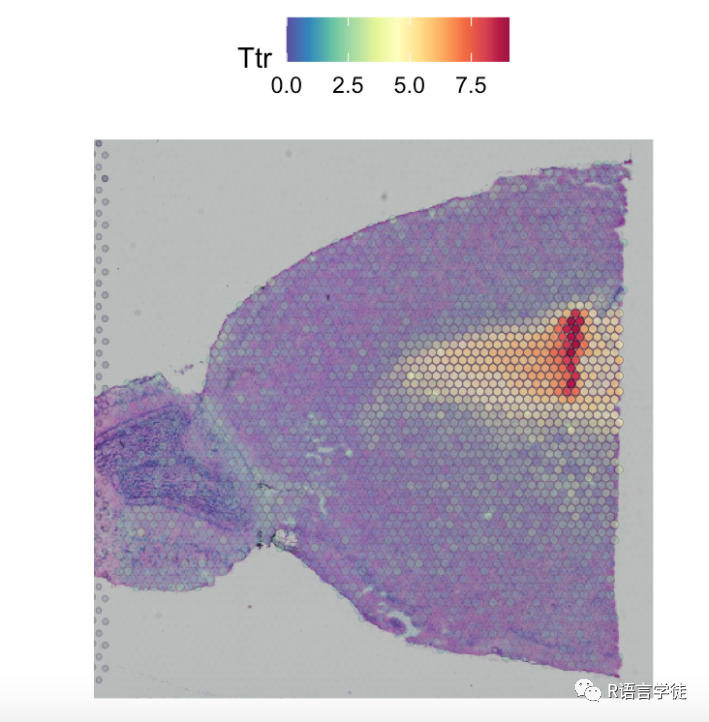
#降低低表达式点的透明度SpatialFeaturePlot(df, features = "Ttr", alpha = c(0.1, 1))
与scRNA-seq分析的工作流程相同,继续对RNA表达数据进行降维和聚类
#对RNA表达数据进行降维和聚类df <- RunPCA(df, assay = "SCT", verbose = FALSE)df <- FindNeighbors(df, reduction = "pca", dims = 1:30)df <- FindClusters(df, verbose = FALSE)df <- RunUMAP(df, reduction = "pca", dims = 1:30)
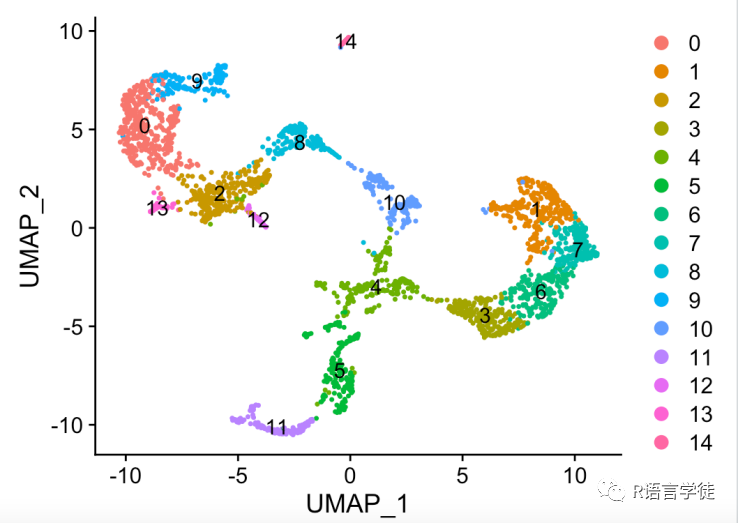
对聚类结果进行可视化
#可视化聚类结果DimPlot(df, reduction = "umap", label = TRUE)
还可以使用SpatialDimPlot()在原本的图像上叠加聚类结果
SpatialDimPlot(df, label = TRUE, label.size = 3)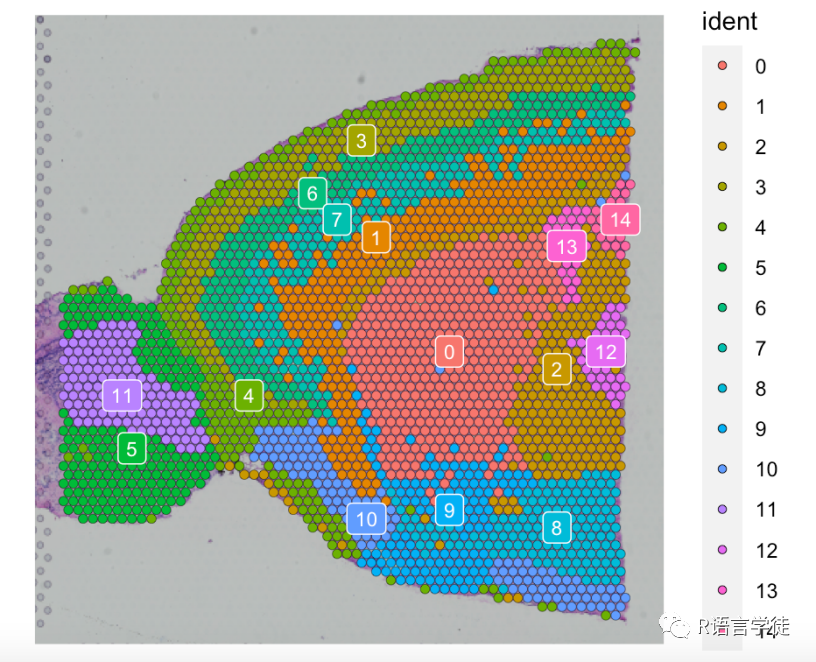
还可以只突出单个簇的细胞的位置情况
SpatialDimPlot(df, cells.highlight = CellsByIdentities(object = df, idents = c(0,1,2,3,4,5,6,7,8,9)), facet.highlight = TRUE, ncol = 5)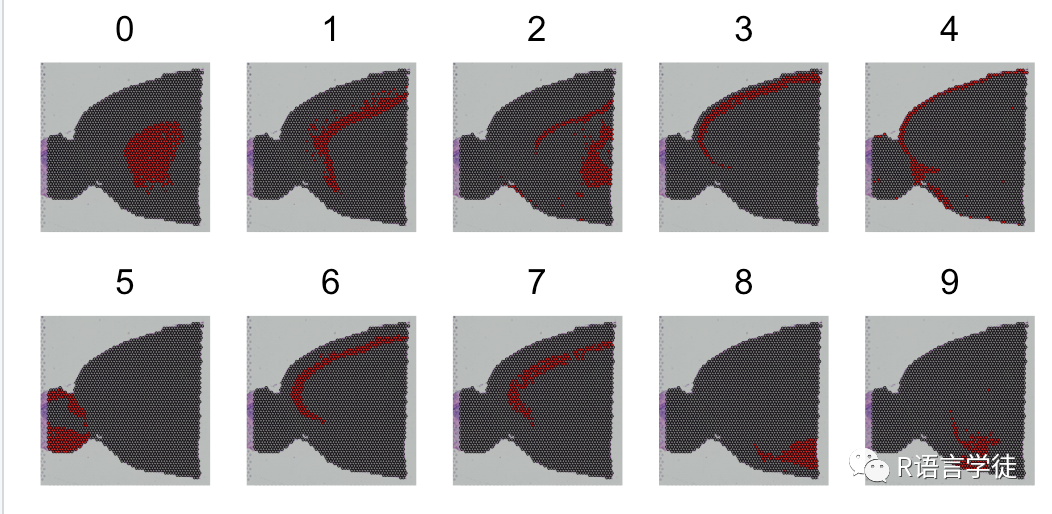
通过小师妹介绍的一键解锁单细胞空间转录组可视化的神奇技巧,我们可以更好地了解单个细胞在组织结构中的分布、类型和功能等信息。通过绘制细胞在空间上的分布图还可以探究细胞类型和功能在组织结构中的空间分布模式、空间关系。这就是今天小师妹带来的可视化技巧啦~快点去试试吧!
还有更多单细胞分析的小工具:单细胞分析(http://www.biocloudservice.com/366/366.php),可以直接在线输入数据,快速得到输出结果!大家一起来使用吧~
