三分钟解决分组合并问题
{ 点击蓝字,关注我们 }
在做生信分析过程中,我们经常会遇到想要把丰度的数据按照分组进行合并分析的情况,那对于刚刚入门生信的小白来讲还是有一定难度的。如果你在做数据分析的过程中遇到了类似的问题,请仔细阅读以下案例,帮助你解决这类问题。
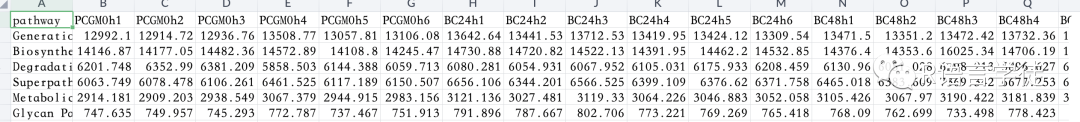
问题描述:有一个分组的文件和一个丰度表的文件,如下图
这两个文件,存放在附件中,供读者分析使用。想要根据第一张图片中分组的信息Treat1列的信息(有三组),对第二张图中的分度数据进行合并。
在数据分析处理的过程中,不论对后续的差异基因表达的挖掘还是对于通路统计的分析,都需要按组进行分析,这就面临上述讲述的问题的解决方案。

问题已经明确了,那通过小师妹的一个脚本来解决上述阐明的问题吧。
以下这个脚本可以实现上述的操作,一起来解读以下吧~
脚本解读
argv <- commandArgs(TRUE)in_file <- argv[1]map_file <- argv[2]#这里argv主要是可以读取shell交互页面的参数,这里的参数为两个文件,第一个为丰度表,另外一个为分组表#read.table读取相关的数据文件map <- read.table(map_file,header = F,quote = "",sep = "t",stringsAsFactors = F)table <- read.table(in_file,header = T,sep = "t",quote = "",stringsAsFactors = F)#提取分组信息group <- unique(map$V4)result <- data.frame(table[,1])#用一个for循环来进行合并(注意这里是按第四列即分组的列)for(i in group){samples <- map[which(map$V4==i),][[1]]tmp_samples <- table[,colnames(table) %in% samples]tmp_samples$result <- rowSums(tmp_samples)/length(samples)result <- cbind(result,tmp_samples$result)}##计算相对丰度colsum <- colSums(result[,-1])for(i in 2:ncol(result)){result[,i] <- result[,i]/colsum[[i-1]]}#修改一下列名,就可以啦~colnames(result) <- c(colnames(table)[1],group)#最后再把文件返写出write.table(result,file = "result.xls",row.names = F ,col.names = T,sep = "t", quote = F)
整个脚本虽然代码不多,主要用到了for循环来实现的。
脚本的用法
1、读者可以一行行复制到自己R环境中进行测试操作,这里我的分组文件的有效列为第四列,根据自己的数据分组来调整相应的位置。
2、调节好后,我们可以把这个脚本保存一下例如另存为samole2group.R,那下次运行时候可以在终端操作以下命令Rscript samole2group.R map_file table_file , 跑完结束会返回名字为result.xls的结果文件。查看文件即按组合并好的文件。

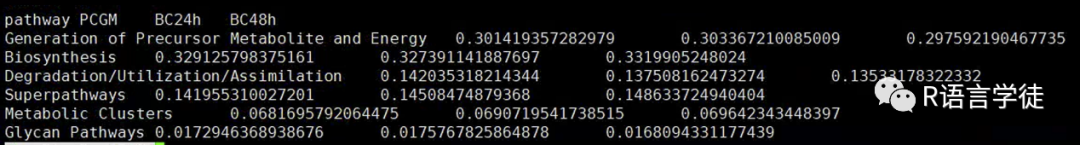
总结
如果读者熟悉shell编程语言,也可以采用三剑客中的一员awk完成此部分的操作并且操作简便,但是需要生信工程师对于awk的用法掌握熟练;此外如果都觉得有困难也可以用excel协助解决。
小师妹认为这种简单的问题也许就会困扰我们很久(小师妹曾经也吃过这方面的苦),因此建议在处理问题的过程中我们应该借助自己最熟悉的工具或是编程语言来协助我们解决问题,并且借鉴前人的经验,解决后也需要保存相应写好的脚本以及脚本的使用说明,这都是生信小白应该养成的良好的习惯。
生信小工具有时候也可以达到你想要的目的哦~http://www.biocloudservice.com/home.html
今天的分享就到这里啦,如果还有什么问题可以和小师妹进行讨论哦~
