想用无监督聚类预测分子亚型?确定不考虑ConsensusClusterPlus包吗?
点击蓝字 关注我们
最近大海哥发现很多小伙伴都通过基因表达数据来对样本进行聚类,划分为多个分子亚型来展开分析,并且都发了高分SCI,这让其他的小伙伴纷纷表示,我也要试一下这个方法,但是要怎么操作呢?别担心,大海哥来替你排忧解难!今天大海哥将介绍一个R包,叫做ConsensusClusterPlus,名字真长,好用也是真好用。这个R包是专门用于无监督一致性聚类,其可根据不同组学数据集将样本区分成几个亚型,从而发现新的疾病亚型或者对不同亚型进行比较分析,为了简化过程,这个R包把所有的步骤都封装到了一个函数里,所以使用起来非常简单,只需要三步
1、 加载R包
2、 导入数据
3、 运行聚类的函数
是不是和徒手劈榴莲一样简单?是不是也正好符合你的想法?那就让我们开始吧!
#首先安装R包,需要使用BiocManager来安装哦# if (!require("BiocManager", quietly = TRUE))# install.packages("BiocManager")# BiocManager::install("ConsensusClusterPlus")#安装过程中会安装官方依赖包“ALL”#本次分析使用的样例数据来源于官方提供的ALL包中的探针表达数据#导入R包library(ALL)library(ConsensusClusterPlus)#导入数据data(ALL)d=exprs(ALL)#要求行为基因,列为样本,行列相反的小伙伴用t()进行转置哦d[1:5,1:5]

#这里为了让效果更佳,我们对数据进行一些预处理#取中位数绝对偏差(Median Absolute Deviation)较大的前5000个探针mads=apply(d,1,mad)df=d [rev(order(mads))[1:5000],]# order(mads):从小到大排序,返回索引# rev(order(mads):从大到小排序#计算每个值减去每行的中位数df = sweep(df,1, apply(d,1,median,na.rm=T))#处理结束,看一下数据df[1:5,1:5]

#然后我们定义一下输出文件夹的名字outdir="clusterResult"#开始聚类!results = ConsensusClusterPlus(df,maxK=6,reps=50,pItem=0.8,pFeature=1,title=outdir,clusterAlg="hc",distance="pearson",seed=1213,plot="pdf")
#这里大海哥稍微介绍一下函数中包含的参数
df:就是我们的数据,要求行为基因,列为样本
maxK:聚类的最大簇数,要求为整数
reps:重采样的次数
pItem:抽样比例
pFeature:样本特征的比例,一般就是1
title:指定输出文件夹名以及输出文件的前缀
clusterAlg:聚类采用的算法,默认是“hc”指层次聚类,还可以选择“km”指K-means聚类,或者一个返回聚类的函数
distance:计算基因距离的方式,有pearson、spearman、euclidean、binary、maximum、canberra、minkowski等,大家自行选择
seed:随机的程度,就随意设置就好了
plot:可以选择png或者pdf,还有pngBMP
#主要就是这些参数,更多参数大家可以上官网瞧瞧哦!
#运行完上面那行代码,就可以得到基本的聚类结果了,我们来看看吧
#下面为主要的聚类结果图
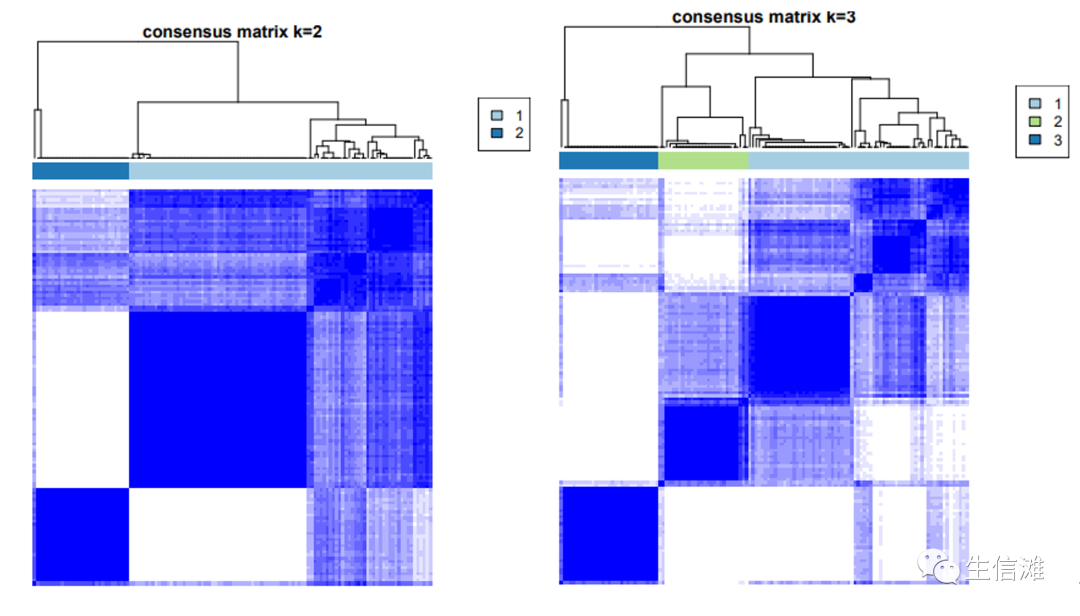
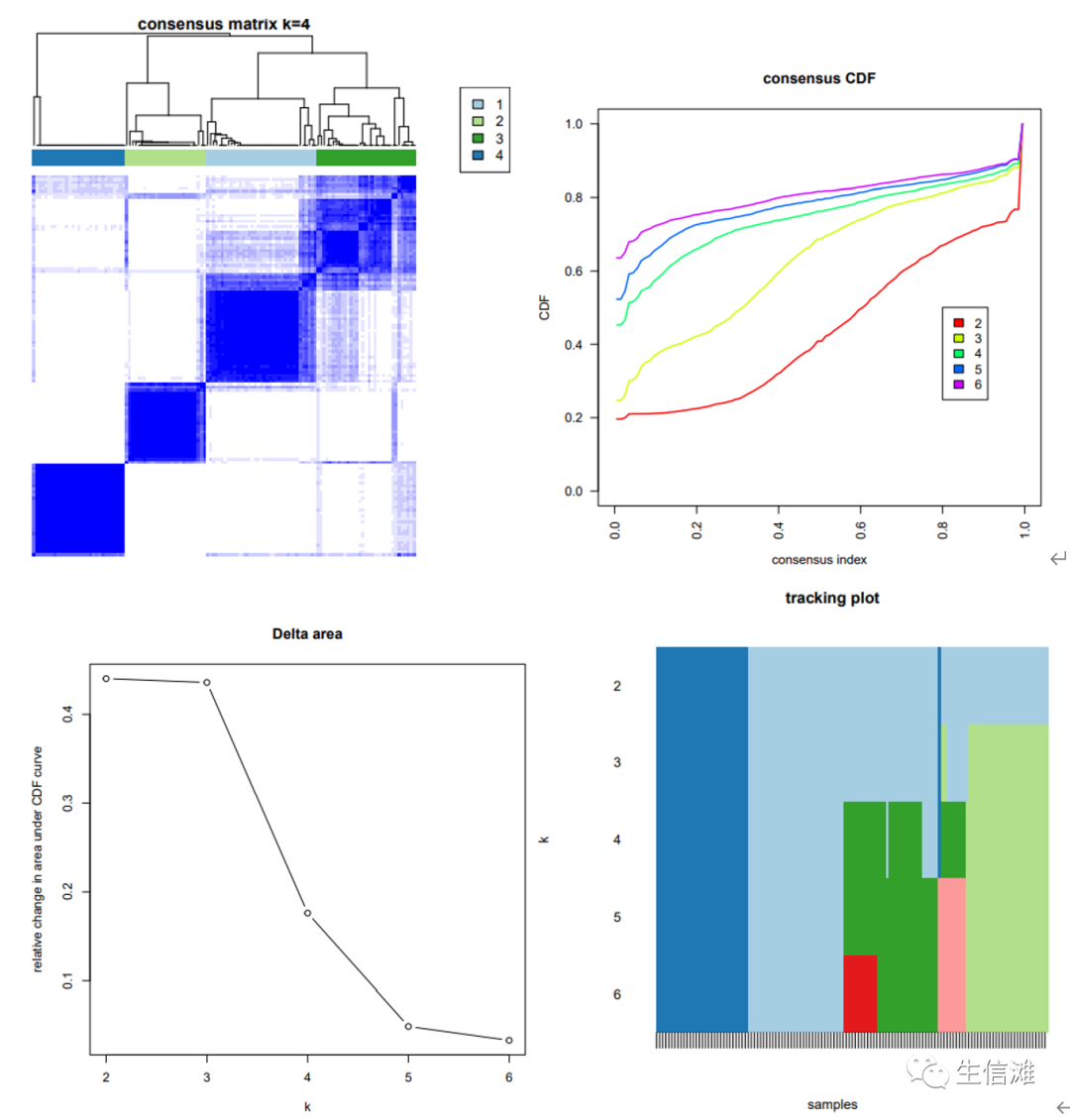
#上图中consensus matrix为样本聚类热图,一共有5张,大海哥这里展示了3张,一般杂乱线条越少说明聚类结果越好;#consensus CDF为累计分布图,我们一般看consensus CDF的那个曲线于X轴更平行,就选k为几,或者通过手肘法观察Delta area来确定,然后选择输出结果中的对应k为几的表格文件就好了#tracling plot中行为样本,列为每个K,一般用于定性评估不稳定的聚类和不稳定的样本#有的小伙伴还想看看组间一致性和组类一致性,就用下面的代码icl = calcICL(results,title=outdir,plot="pdf")#然后就可以得到下面的图
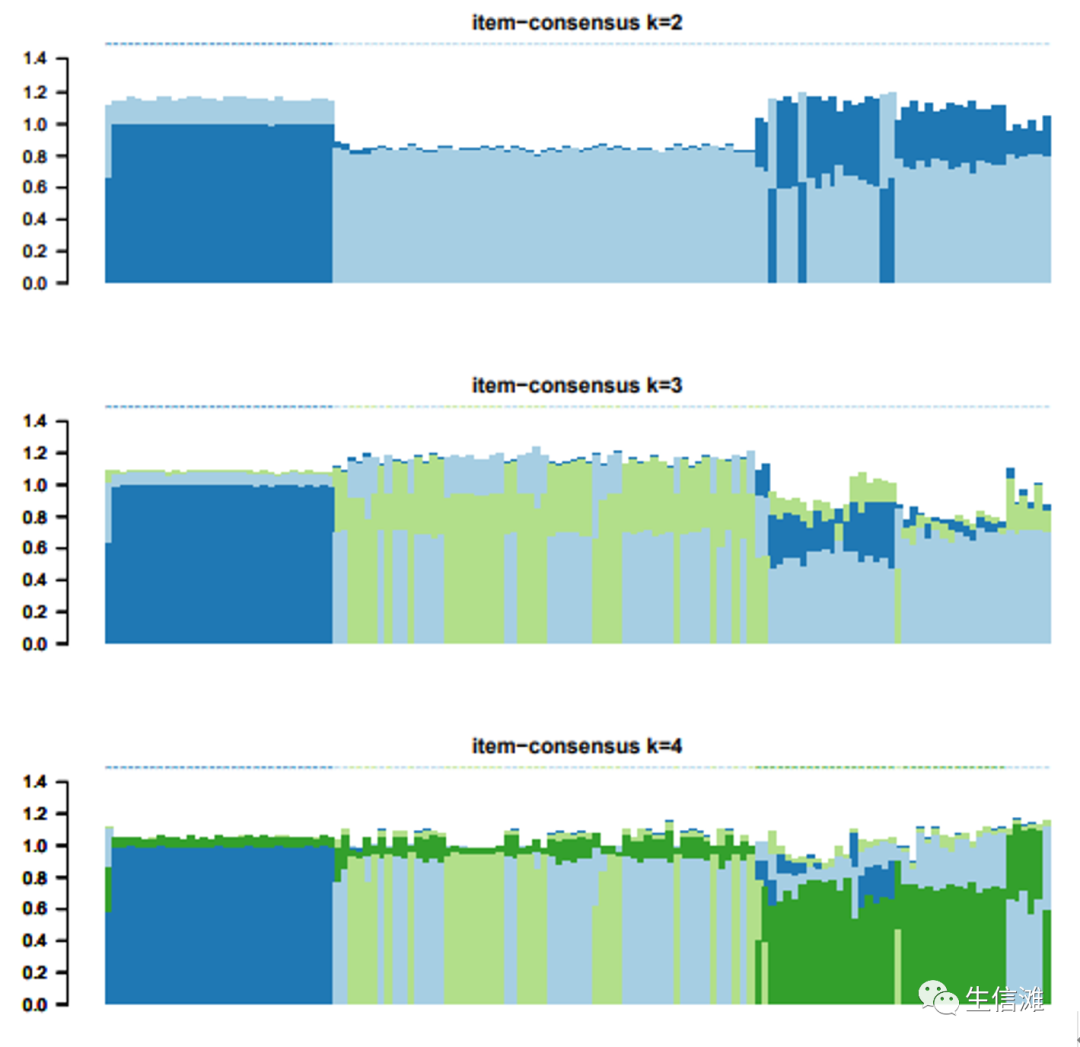

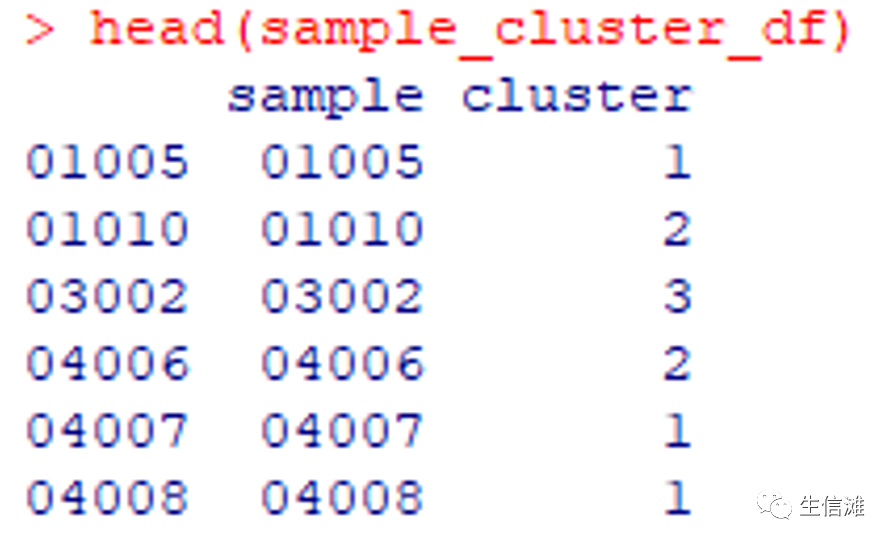
#选择合适的k值后,可以得到对应聚类结果的数据框sample_cluster <- results[[6]]$consensusClasssample_cluster_df <- data.frame(sample = names(sample_cluster), cluster = sample_cluster)head(sample_cluster_df)
点击“阅读原文”进入网址