三分钟教你入门生存分析之Cox比例风险模型
{ 点击蓝字,关注我们 }
Cox比例风险模型(Cox proportional hazards model)是一种常用的生存分析方法,用于评估不同变量对于生存时间的预测能力。它是由David Cox于1972年首次提出的。Cox比例风险模型在生物信息学中有广泛的应用,主要用于生存分析和预测。在生存分析中,Cox比例风险模型可以用于评估不同基因或其他生物学因素对生存时间的影响。例如,对于癌症研究,研究人员可以使用Cox比例风险模型来探索不同基因表达水平与癌症患者生存时间的关系,并识别与生存时间相关的预测因子。此外,Cox比例风险模型还可以用于基于基因表达数据进行生存预测。例如,在肿瘤研究中,研究人员可以根据某些基因的表达水平,使用Cox比例风险模型来预测患者未来的生存时间,并评估不同基因对预测能力的贡献。这种方法可以帮助医生为患者制定个性化的治疗方案,提高治疗效果。在生物信息学中,Cox比例风险模型通常与高通量数据分析方法相结合,如基因芯片、RNA测序等,可以进一步提高生存分析和预测的准确性和可靠性。
Cox比例风险模型基于风险比(hazard ratio)的概念,即不同个体之间生存时间的比较。它假设生存时间是由两个部分组成:基准风险(baseline hazard)和可调整风险(adjustable hazard)。其中,基准风险是所有个体都具有的风险,可以看作未受到任何影响的基础风险;可调整风险是个体之间不同的风险,可以由一些可调整的协变量(covariates)解释。
Cox比例风险模型的基本形式为:
h(t|X) = h0(t) * exp(Xβ)其中,h(t|X)是在给定协变量X的条件下,时间t的风险比;h0(t)是时间t的基准风险;exp(Xβ)是可调整风险的比率,也称为协变量的风险比(hazard ratio);X是协变量的矩阵;β是协变量的系数向量。Cox比例风险模型的核心假设是协变量的风险比在时间轴上是恒定的,即不随时间变化。
那么现在就让小师妹带大家一起在R中实践一下吧~
我们可以先对数据做一些可视化,以检视得出的结果和直观上的数据是否一致
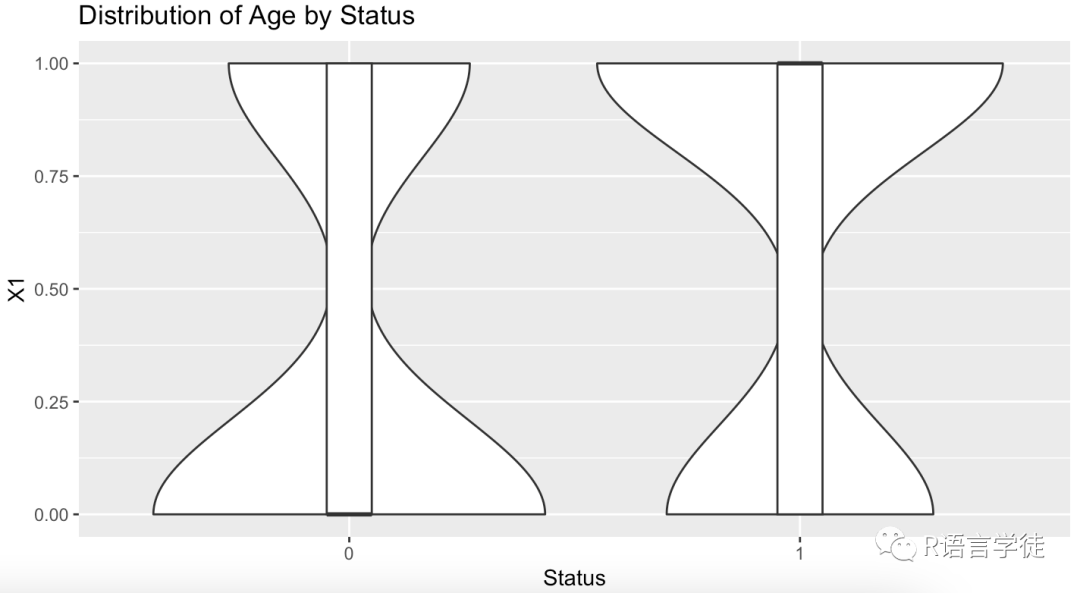
比如,通过小提琴图观察X1在不同status下的分布情况
ggplot(d, aes(x = factor(status), y = X1)) +geom_violin() +geom_boxplot(width = 0.1, fill = "white") +labs(title = "Distribution of Age by Status", x = "Status", y = "X1")
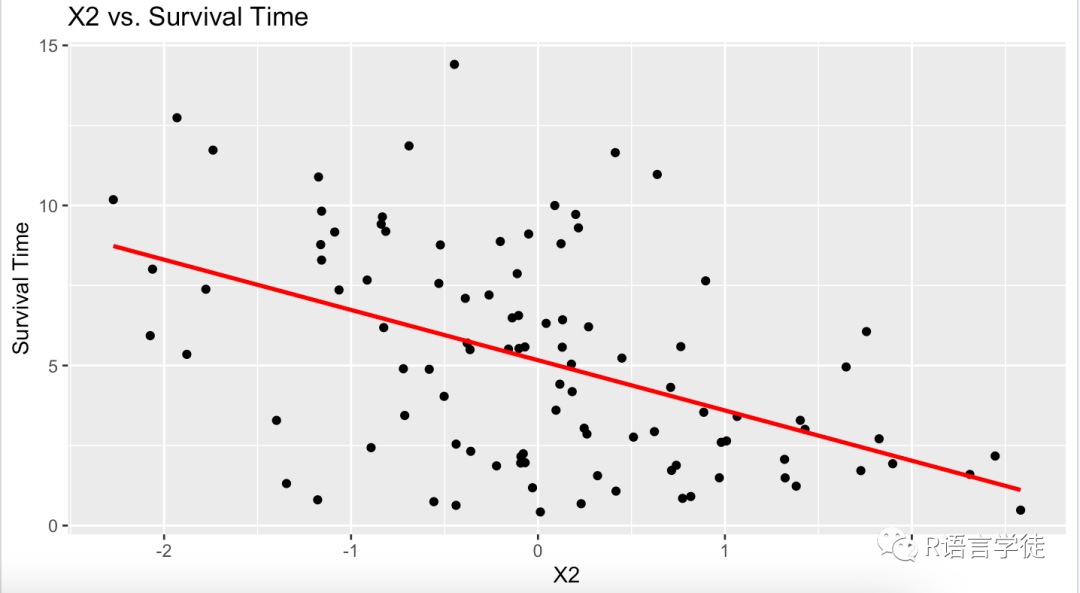
绘制散点图,显示X2和生存时间的关系
ggplot(d, aes(x = X2, y = time)) +geom_point() +geom_smooth(method = "lm", se = FALSE, color = "red") +labs(title = "X2 vs. Survival Time", x = "X2", y = "Survival Time")
## 载入相关的工具包:
## 载入相关的工具包:library(rms)library(riskRegression)library(survival)## 使用 prodlim 包生成生存数据set.seed(100)d <- SimSurv(100)## 构建Cox回归模型并计算预测生存概率coxmodel <- cph(Surv(time, status) ~ X1 + X2, data = d, surv = TRUE)coxmodel
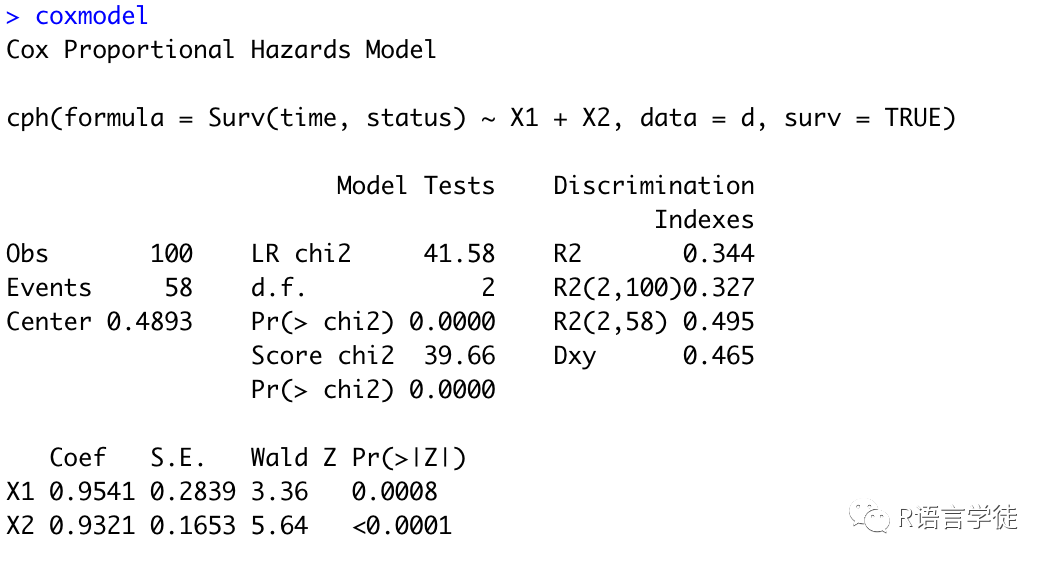
这个结果中的模型系数表示自变量对生存率的影响,系数越大表示影响越大,系数为正表示该变量与生存率正相关,系数为负表示该变量与生存率负相关。当模型系数显著时,P值通常小于0.05,表示变量对生存率的影响是显著的;当模型系数不显著时,P值通常大于0.05,表示变量对生存率的影响是不显著的。
## 提取在选定时间点的预测生存概率ttt <- quantile(d$time)ndat <- data.frame(X1 = c(0.25, 0.25, -0.05, 0.05), X2 = c(0, 1, 0, 1))predictSurvProb(coxmodel, newdata = ndat, times = ttt)
首先使用quantile()函数计算原始数据集d中生存时间变量time的5个分位数,将结果存储在向量ttt中。然后,创建一个新的数据框ndat,其中包含两个自变量X1和X2,每个自变量有4个不同的取值。接着,使用predictSurvProb()函数对ndat进行生存概率预测,其中coxmodel是先前拟合的Cox回归模型,newdata = ndat表示对ndat中的数据进行预测,times = ttt表示计算在5个分位数时间点下的生存概率。最终,函数将预测结果输出到一个矩阵中,其中每一行表示一个样本在不同时间点下的生存概率预测值。

在Cox比例风险模型中,常常存在某些自变量的效应可能与风险群体有关,即不同风险群体可能具有不同的自变量效应。例如,在一个研究中,我们发现X1在不同年龄段的患者中可能具有不同的影响,因此我们需要根据年龄将样本分为不同的群体,然后在每个群体中分别拟合Cox模型。
在这种情况下,我们可以使用strata()函数来实现分层。strata()函数将可以被用于为Cox模型中的自变量指定分层,这样就可以在每个分层中分别拟合Cox模型。在这里,我们将X1变量作为分层变量,即将数据集中的样本按照X1变量的不同取值分成不同的组,然后在每个组中拟合Cox模型,以便更好地捕捉X1变量的影响。
library(ggplot2)library(tidyverse)library(GSVA)

predictSurvProb(sfit, newdata = d[1:3, ], times = c(1, 3, 5, 10))
使用predictSurvProb()函数对数据集d中的前3个观测值进行生存概率预测,其中newdata = d[1:3, ]表示对数据集d中的前3个观测值进行预测,times = c(1, 3, 5, 10)表示预测在1、3、5和10个时间点下的生存概率。
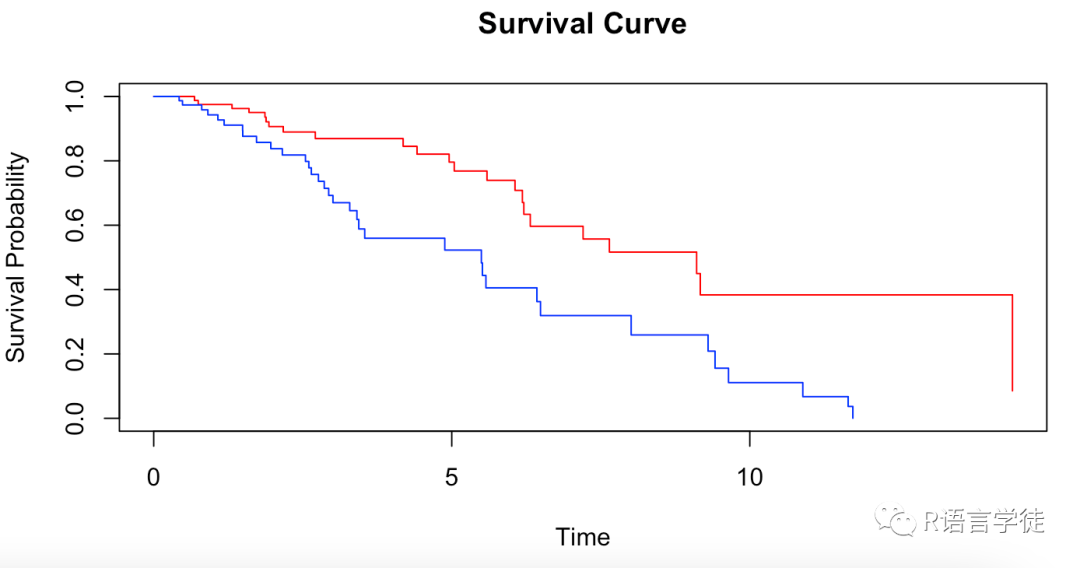
## 使用plot()函数对生存曲线进行可视化sfit <- survfit(sfit)plot(sfit, col=c("red", "blue"), main="Survival Curve", xlab="Time", ylab="Survival Probability")
还可以绘制森林图:森林图是一种用于显示 Cox 回归模型中各个变量的系数和置信区间的可视化方法。它通常用于显示多个变量的结果,并将它们按照重要性排序。
coxm <- coxph(Surv(time, status) ~ X1 + X2, data = d)library(ggplot2)library(forestmodel)forest_model(coxm,theme = theme_forest(),factor_separate_line=TRUE)
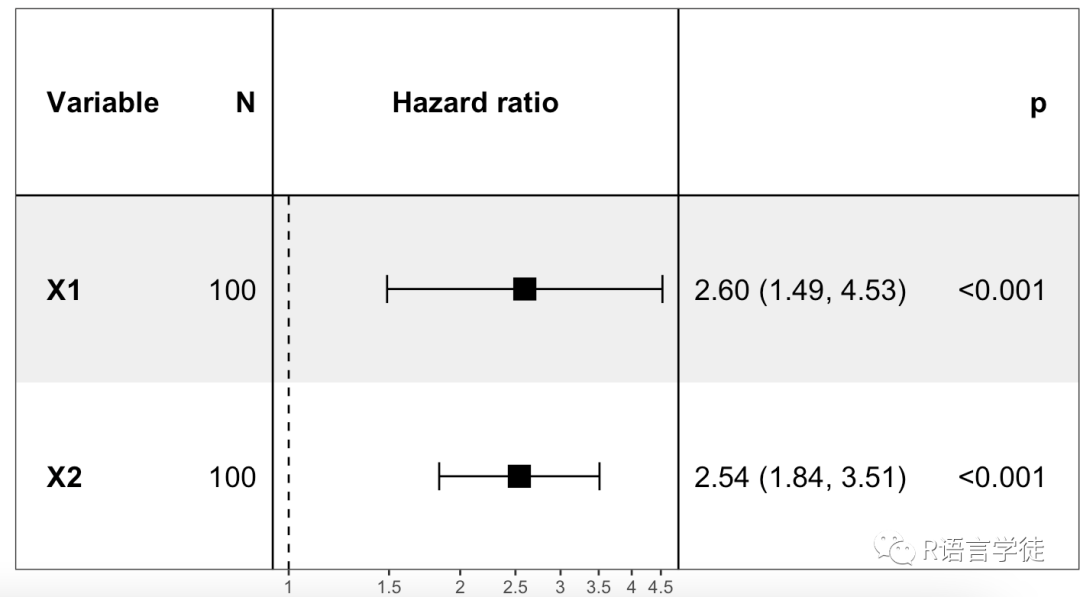
这就是Cox比例风险模型的应用过程啦~大家学会了吗~有疑问的小伙伴欢迎留言和小师妹交流哦~
E
N
D

更多方便实用的小工具在云生信平台等着大家哦!
http://www.biocloudservice.com/home.html
