为什么高分文章都喜欢用lasso回归来提取特征,这里有答案!
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

小果昨天下班后,想着看看有啥生信好文章,看看能不能发现一些好套路来给小伙伴们谋福利,功夫不负有心人,还真的发现了一个。
小果发现很多文章都喜欢用lasso回归来进行特征选择。这是为什么呢?今天小果就以一篇Journal of Clinical Oncology杂志(IF=26.3)中的文章为例来说明这个原因。
这篇文章采用影响组学方法在结直肠癌淋巴结转移方面进行预测研究,目的是为了预测结直肠癌术前淋巴结转移的风险,进而为临床医生提供决策参考,具有重要的临床价值。好文章一定要有好数据!这篇文章便意识到了这一点,于是作者在提取原始图片数据的特征后,便考虑到了特征选择。那么用什么方法最好呢?Lasso回归!作者利用Lasso回归从150个特征中筛选出24个回归系数非零的具有预测作用的特征,在此基础上完成后续实验。今天小果就用一个案例分析来带大家认识这个高分生信必备算法——Lasso回归。
今天使用的数据集是一个前列腺癌数据集,该数据集有97个样本,每个样本包含9个特征。数据样例如下图所示。
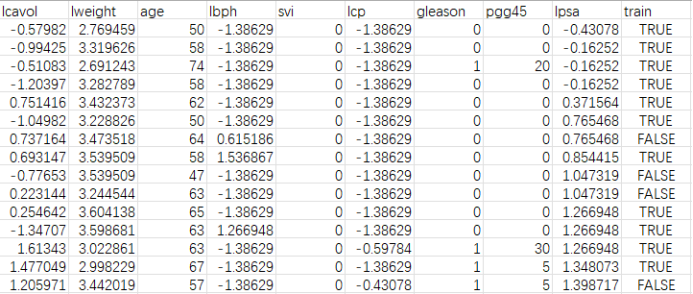
其中train中的TRUE和FALSE表示该行样本是否为训练集,lpsa为结果变量,PSA表示病人前列腺癌特异性抗原水平,lpsa即为其对数。本次分析即为利用lasso回归提取出可以表现lpsa表达水平的特征。
1、加载R包及数据
library(caret)library(glmnet)prostate1<-read.csv("prostate.csv")head(prostate)#测试集训练集划分train<-subset(prostate, train ==TRUE)[, 1:9]str(train)test <-subset(prostate, train==FALSE)[,1:9]str(test)#构建Lasso回归模型x <-as.matrix(train[, 1:8])y <-train[, 9]#Lasso回归,指定模型类型,这里采用gaussian类型lasso<- glmnet(x, y, family =“gaussian”, alpha =1)print(lasso)#查看系数和L1范数的关系plot(ridge, label =TRUE)
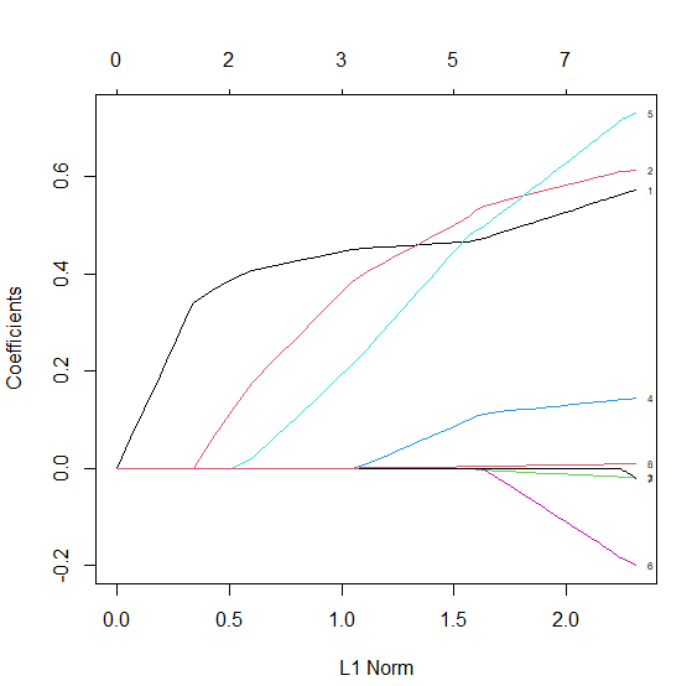
plot(lasso, xvar =“lambda”, label =TRUE)
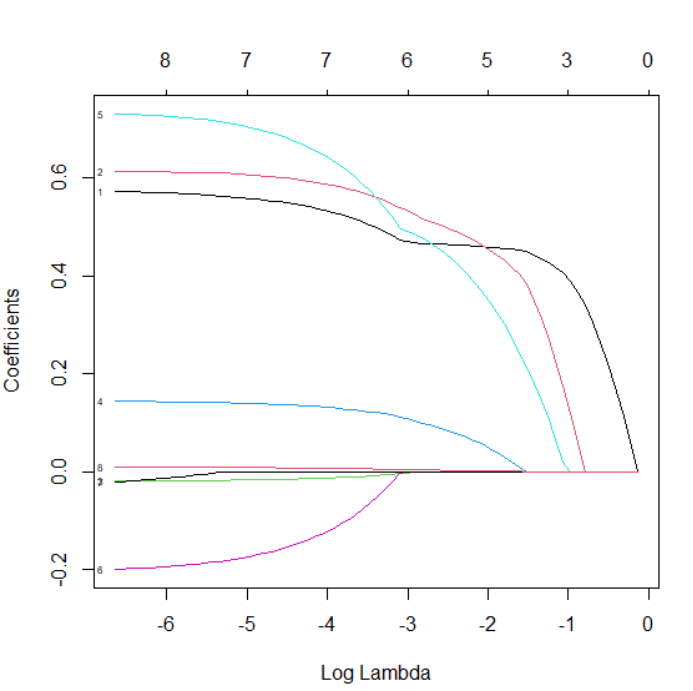
#十折交叉验证,选择合适的lambda值cvfit <- cv.glmnet(x, y,nfolds = 10)plot(cvfit)#这里选择lambda.lse,得到参数最少且性能优良的模型print(cvfit$lambda.1se) #0.3158532#可以查看模型参数估计结果coefficients<-coef(lasso,s= cvfit$lambda.1se)Active.Index<-which(coefficients!=0) #系数不为0的特征索引Active.coefficients<-coefficients[Active.Index] #系数不为0的特征系数值
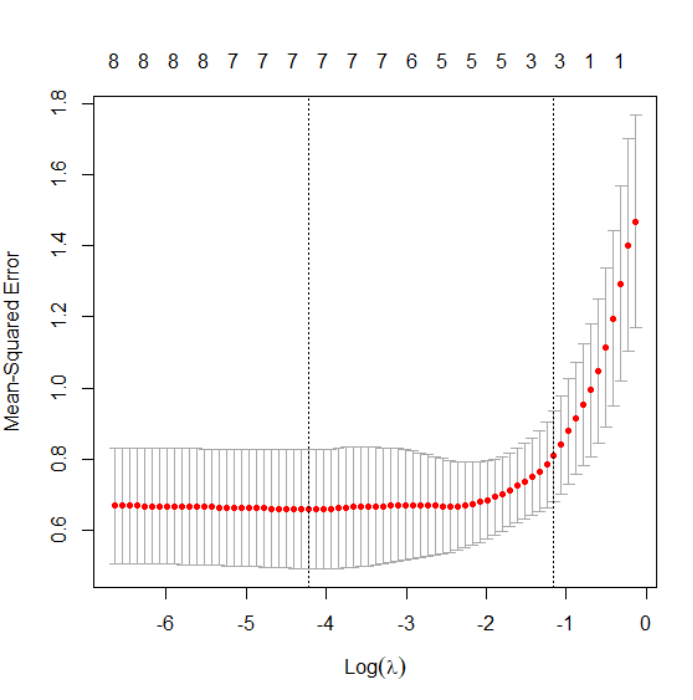
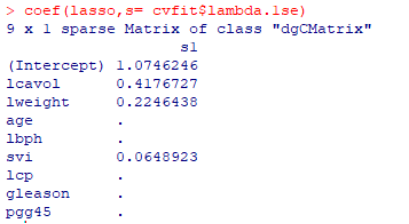
结果可以看到根据lasso回归构建模型, 发现lcavol、lweight、svi三个特征系数不为0,可以作为有效特征来代替病人样本PSA表达水平,进而展开后续分析。小伙伴们也可以将样例数据换成自己的数据,利用lasso回归提取出能够替代病人临床结果的有限特征,进而展开更有意义的分析。高分文章近在咫尺!心动的小伙伴一定不要错过!
这里推荐一下小果新开发的零代码云生信分析工具平台包含超多零代码小工具,上传数据一键出图,感兴趣的小伙伴欢迎了解~
网址:http://www.biocloudservice.com/home.html
今天小果的分享就到这里,欢迎大家和小果一起讨论学习,下期再见哦!
小果友情推荐
好用又免费的工具安利


