Igraph包:三分钟帮你实现蛋白互作网络中的网络拓扑性质分析
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

还在为如何入门生信而彷徨吗?还在为Linux系统而感到无助吗?快关注小果的微信公众号,小果手把手带你入门生信,没有门槛、没有难度,跟着效果走,啥问题都没有。
蛋白互作网络是由蛋白质相互作用所构成的网络结构。网络拓扑结构分析是对蛋白互作网络中节点和边的特征进行分析,以揭示网络的基本特性和功能模式。
常用的网络拓扑结构分析方法包括度分布分析、聚类系数分析、介数中心性分析、网络中心性分析等。其中度分布分析可以揭示网络中节点的连接情况,聚类系数分析可以揭示网络中节点的聚集程度,介数中心性分析可以揭示网络中节点在信息传递中的重要性,网络中心性分析可以揭示网络中节点的重要性和影响力。
通过网络拓扑结构分析,可以深入了解蛋白互作网络的特性和功能,为进一步研究蛋白质相互作用提供重要参考。
今天,小果手把手教学,三分钟带你学会igraph包,导师再也不用担心你不会做ppi下游分析啦!
1、加载R包及数据
要进行网络拓扑性质分析,首先要构建蛋白互作网络。常用的构建蛋白互作网络的方式有使用cytoscape、string。小果今天介绍的igraph包,也可以帮助我们构建蛋白互作网络。首先,我们需要准备蛋白互作的数据,即互作的蛋白对。小果选用了人类18210个蛋白产生的431696互作数据(小果将初试蛋白对数据命名为protein_pair_label.csv文件,大家也可以在hippie数据库中直接下载,hippie数据库是ppi数据库之一)
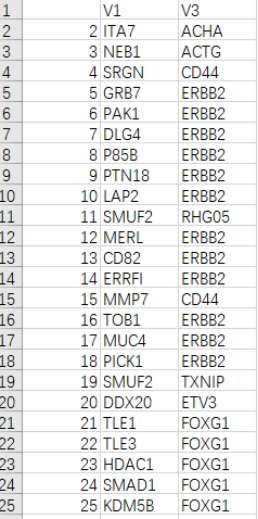
图 1 蛋白互作对
2、Igrpah包实战,构建单板互作网络
Install.packages(“igraph”) #下载igraph包library(“igraph”) #导入igraph包human<-read.csv(file.choose()) #导入数据human<-human[,c(2,3)] #数据格式处理,只保留互作的两列蛋白HumanPro<-graph.data.frame(human, directed=FALSE)summary(HumanPro)
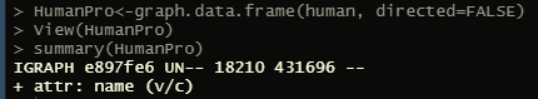
图 2summary之后的结果
建立的人类蛋白质相互作用网络中含有18210个节点,431696条边,为无向图
plot(HumanPro) 可以展示蛋白质网络图,由于小果选用的蛋白对太多,网络图十分复杂。
图 3网络图
如果大家想要更加精美的网络图,小果之前有发过cytoscape的内容哦!
图 4cytoscape构建的网络图
3、Igrpah实战,度分析、介度分析
Degr<-degree(HumanPro)write.table(Degr,"D:\sj\Degree.txt") #将计算出的度保存在文件Degree.txt中。简单地画一下网络的度分布图:DegrDis<-degree.distribution(HumanPro)plot(DegrDis)
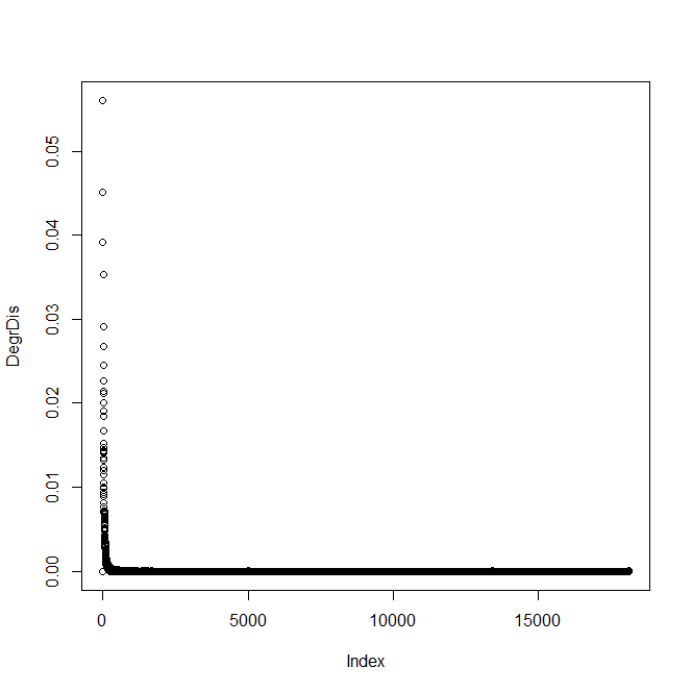
图 5 度分布图
网络的介度计算也是同理:
Betw<-betweenness(HumanPro,directed=FALSE,normalized=TURE,以上就是今天的内容啦!
这里推荐一下小果新开发的零代码云生信分析工具平台包含超多零代码小工具,上传数据一键出图,感兴趣的小伙伴欢迎了解~
网址:http://www.biocloudservice.com/home.html
今天小果的分享就到这里,欢迎大家和小果一起讨论学习,下期再见哦!
小果友情推荐
好用又免费的工具安利


