五分钟教会你使用R语言包nlme快速建立线性和非线性混合效应模型
{ 点击蓝字,关注我们 }
R语言包nlme是一个用于实现线性和非线性混合效应模型的强大工具。混合效应模型是一种广泛应用于统计建模的技术,特别适用于分析具有重复测量或层次结构的数据。该包提供了一系列功能,可以帮助研究人员更好地理解和解释数据中的随机变异和固定效应。
要使用nlme包,可以在R中使用以下命令进行安装和加载:
> install.packages("nlme") #安装nlme语言包> library(nlme) #加载语言包
线性混合效应模型是混合效应模型的一种特殊情况,它假设数据中的随机变异是通过一个线性模型来描述的。lme()函数可以处理这类模型,同时可以指定固定效应和随机效应,从而更好地捕捉数据的变异性。此外,nlme包还提供了一系列用于诊断模型拟合效果的函数,例如通过绘制残差图、Q-Q图和相关图来检验模型是否满足假设。
示例:
假设我们有一组生物学实验数据,其中测量了不同个体的某个生物指标(例如体重)在不同时间点下的数值。这种数据通常包含多个个体的多次测量,具有层次结构,因为同一组个体在不同时间点下有多次观测。
首先,我们需要安装和加载nlme包,并准备示例数据。这里使用一个虚构的示例,假设我们有一个名为”biological_data”的数据框,其中包含三列:”个体ID”表示不同个体的标识,”时间”表示测量的时间点,”体重”表示对应时间点下的体重数值。
#安装并加载nlme语言包> install.packages(“nlme”)> library(nlme)# 创建虚拟的生物学数据# 假设有5个个体,每个个体有3个时间点的测量> individuals <- factor(rep(1:5, each = 3))> time_points <- factor(rep(1:3, times = 5))> weights <- c(50, 52, 55, 60, 58, 62, 65, 63, 68, 70, 72, 75, 80, 78, 82)# 创建数据> biological_data <- data.frame(个体ID = individuals, 时间 = time_points, 体重 = weights)
现在,我们使用nlme包来拟合一个线性混合效应模型,将体重作为响应变量,时间作为固定效应,个体ID作为随机效应。我们可以使用lme()函数来实现这个模型。在这个模型中,”体重”是响应变量,”时间”是固定效应,”个体ID”是随机效应。通过将个体ID作为随机效应,我们考虑了个体之间的随机变异,同时利用时间作为固定效应,来考虑不同时间点对体重的影响。
# 拟合线性混合效应模型> linear_mixed_model <- lme(体重 ~ 时间, random = ~1 | 个体ID, data = biological_data)# 查看模型的摘要> summary(linear_mixed_model)

我们可以绘制模型的残差图,检验模型是否满足线性假设。
# 绘制残差图> plot(resid(linear_mixed_model) ~ fitted(linear_mixed_model),+ xlab = "Fitted Values",+ ylab = "Residuals",+ main = "Residual Plot")
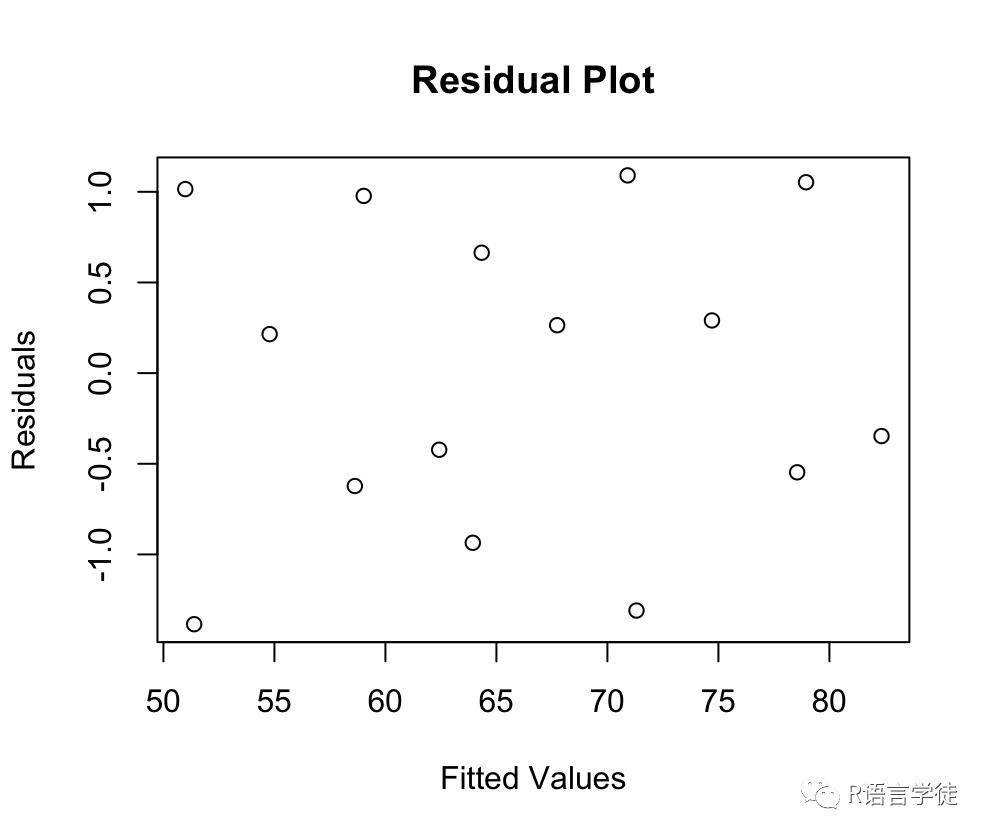
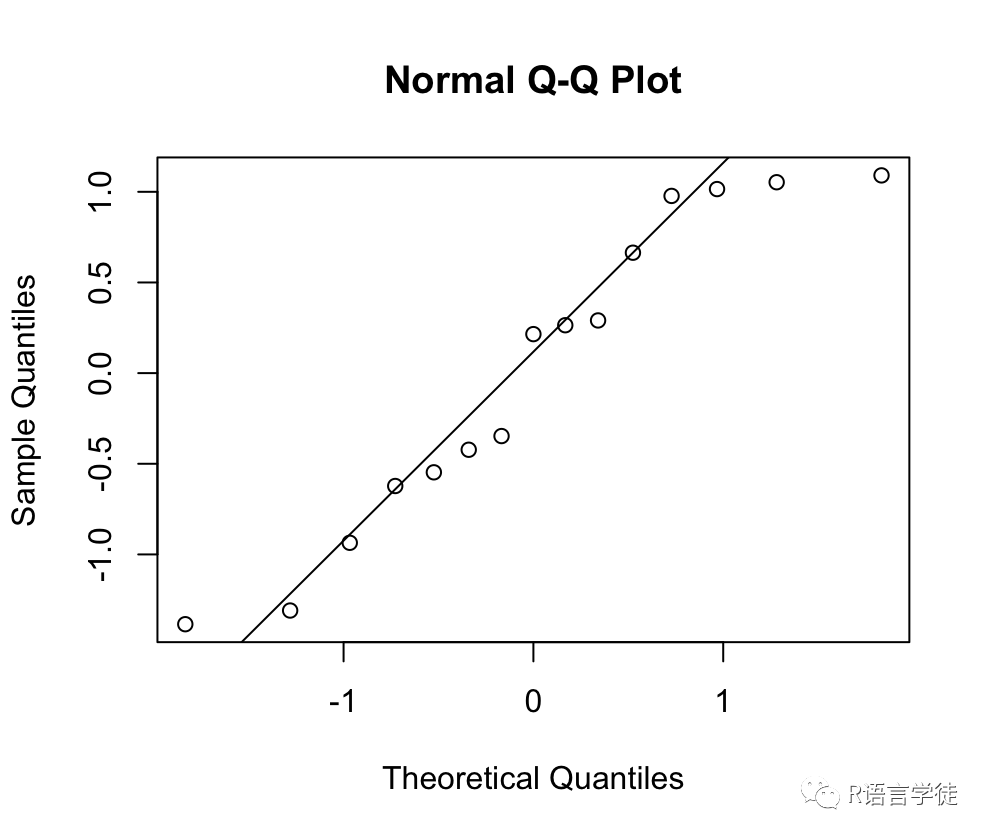
除了残差图,我们还可以绘制Q-Q图和相关图来进一步检验模型是否满足假设。
# 绘制Q-Q图> qqnorm(resid(linear_mixed_model))> qqline(resid(linear_mixed_model))
# 绘制相关图> plot(time_points, resid(linear_mixed_model),+ xlab = "Time",+ ylab = "Residuals",+ main = "Residuals vs. Time")
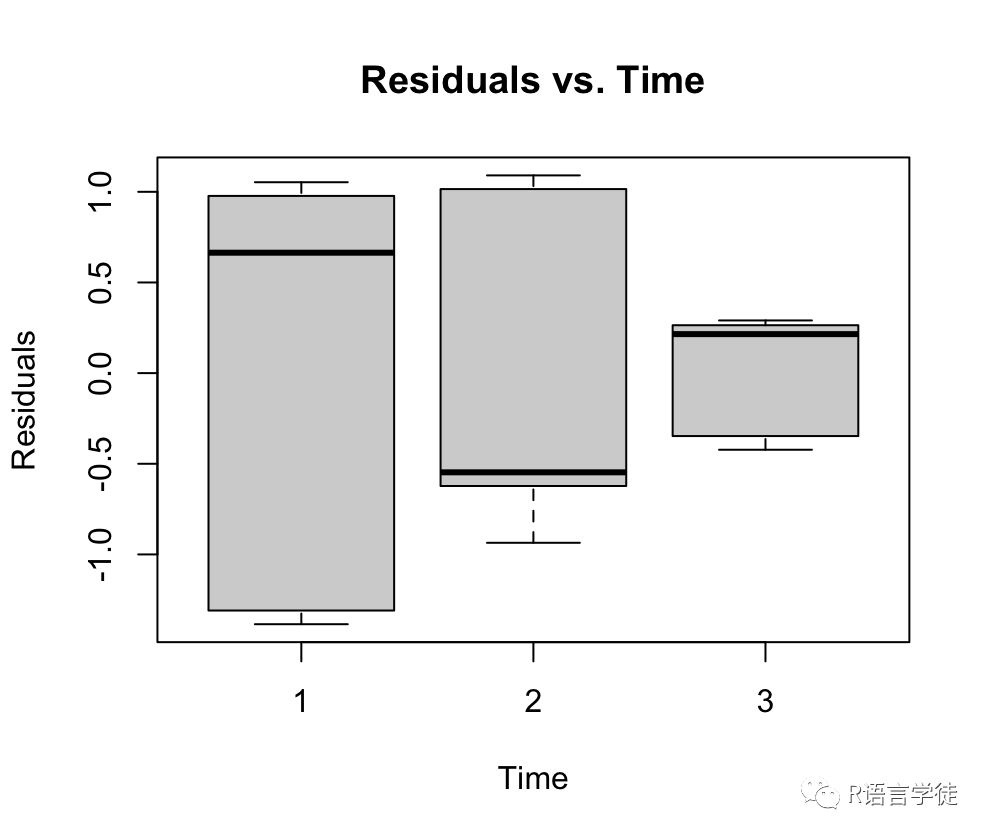
通过这些诊断图,我们可以检查模型是否满足线性假设,以及随机误差是否符合正态分布和独立性的假设。
相比之下,非线性混合效应模型更加灵活,因为它允许数据中的随机变异通过非线性函数来描述。nlme()函数提供了拟合非线性混合效应模型的功能,它可以用于解决许多实际问题,例如药物代谢动力学的建模、生长曲线的拟合等。非线性混合效应模型通常需要更多的计算资源和更复杂的模型设定,但在某些情况下,它可以更好地拟合数据和预测未知数据。
示例:
假设我们有一个生物学实验数据集,其中包含了一组植物的生长数据。我们希望使用非线性混合效应模型来拟合这些数据,以探索植物的生长曲线模式。假设我们有一个名为”plant_growth_data”的数据框,其中包含三列:”时间”表示测量的时间点,”个体ID”表示不同植物个体的标识,”生长量”表示对应时间点下植物的生长量。
# 假设我们有一个名为"plant_growth_data"的数据框# 创建虚拟的生物学数据> time_points <- c(1, 2, 3, 4, 5, 1, 2, 3, 4, 5) # 5个时间点> individuals <- factor(rep(1:2, each = 5)) # 2个植物个体> growth <- c(2.5, 3.8, 4.2, 5.1, 6.0, 1.8, 2.9, 3.5, 4.0, 4.8) # 生长量# 创建数据框> plant_growth_data <- data.frame(时间 = time_points, 个体ID = individuals, 生长量 = growth)
在这个例子中,我们将使用Logistic生长曲线来描述植物的生长模式,该曲线是非线性的。我们将时间作为固定效应,个体ID作为随机效应,并使用Logistic函数作为非线性的生长模型。
# 定义Logistic生长模型> logistic_model <- function(t, b0, b1, b2) {+ y <- b0 / (1 + exp(-b1 * (t - b2)))+ return(y)+ }> # 拟合非线性混合效应模型> nonlinear_mixed_model <- nlme(生长量 ~ logistic_model(时间, b0, b1, b2),+ fixed = b0 + b1 + b2 ~ 1,+ random = b0 + b1 + b2 ~ 1 | 个体ID,+ start = c(b0 = 1, b1 = 1, b2 = 1),+ data = plant_growth_data)# 查看模型的摘要> summary(nonlinear_mixed_model)
在这个模型中,我们通过自定义的Logistic生长模型logistic_model()来描述植物的生长模式。模型中有三个参数b0、b1、b2,分别代表生长曲线的最大值、生长速率和中点位置。通过拟合非线性混合效应模型,我们可以得到这些参数的估计值,从而获得了关于植物生长曲线的信息。
除了模型拟合功能,nlme包还提供了一系列用于预测和可视化模型结果的函数。研究人员可以使用这些函数来从已拟合的模型中提取参数估计值、预测新数据点以及绘制拟合曲线和效应图。这些工具为研究人员提供了更好地理解数据和模型的能力,从而帮助他们做出更合理的统计推断。
以上就是对R语言包nlme的简单介绍啦,nlme包是一个功能强大且灵活的R语言工具,适用于处理复杂的统计建模问题。它允许研究人员在数据中引入随机效应,更好地捕捉数据的变异性,并且可以适用于线性和非线性模型。通过nlme包,研究人员可以更深入地挖掘数据中的信息,从而为各种学科领域的研究提供有力的支持。无论是在医学、生态学、心理学还是社会科学等领域,nlme包都能发挥重要的作用,帮助研究人员做出准确可靠的统计推断,推动科学的发展与进步。
小伙伴们,今天有没有学到新知识呢,想要继续了解R语言内容可以持续关注小师妹哦~~或者也可以关注我们的官网也会持续更新的哦~ http://www.biocloudservice.com/home.html

