纯代码实操,师妹带你做基因与临床的生存分析与校准
点击蓝字,关注我们
我们经常对基因表达数据与临床数据进行生存分析,今天小师妹给大家带来不一样的分析预测,他就是Nomogram。我们先了解一下概念理论再进行操作。
Nomogram,也常称为诺莫图或者列线图,在医学领域的期刊出现频率越来愈多,常用于评估肿瘤学和医学的预后情况,可将Logistic回归或Cox回归的结果进行可视化呈现。
校准曲线(Calibration Curve)量度着预测准确性。有助于确定在不同时间点上模型的预测能力如何。这样的分析对于基因与临床因素在肿瘤研究中的应用具有重要意义。
言归正传,实操部分来了。下面是示例样本的表达量信息和临床数据文件。

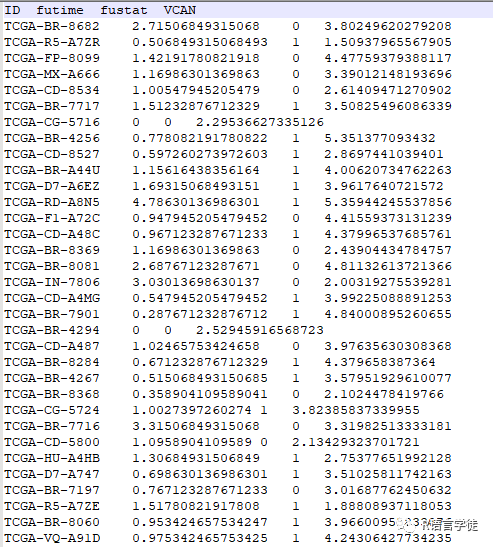
#install.packages("survival")#install.packages("regplot")#install.packages("rms")#引用包library(survival)library(regplot)library(rms)expFile="expTime.txt" #表达数据文件cliFile="clinical.txt" #临床数据文件setwd() #设置工作目录#定义了几个变量,包括"expFile"和"cliFile",分别为表达数据文件和临床数据文件的文件名。#另外,通过"setwd"函数设置了工作目录exp=read.table(expFile, header=T, sep="t", check.names=F, row.names=1)#读取名为"expFile"的表达数据文件,该文件是一个以制表符分隔的文本文件,包含基因表达矩阵。#"header=T"表示第一行是列名,"sep="t""表示使用制表符作为分隔符,"check.names=F"表示不检查列名的合法性,#"row.names=1"表示使用第一列作为行名。读取后的数据被保存在变量"exp"中,是一个包含基因表达矩阵的数据框。cli=read.table(cliFile, header=T, sep="t", check.names=F, row.names=1)cli=cli[apply(cli,1,function(x)any(is.na(match('unknow',x)))),,drop=F]cli$Age=as.numeric(cli$Age)#读取名为"cliFile"的临床数据文件,该文件也是一个以制表符分隔的文本文件,包含与每个样本相关的临床信息。#"header=T"表示第一行是列名,"sep="t""表示使用制表符作为分隔符,"check.names=F"表示不检查列名的合法性,#"row.names=1"表示使用第一列作为行名。#然后,代码使用apply函数过滤出含有"unknow"的行,再将"Age"列转换为数值类型。samSample=intersect(row.names(exp), row.names(cli))exp1=exp[samSample,,drop=F]cli=cli[samSample,,drop=F]rt=cbind(exp1, cli)#合并表达数据"exp"和临床数据"cli"。首先,找出两个数据框中共同具有的样本(行名),并将这些样本作为"exp"和"cli"的子集。#然后使用cbind函数将"exp1"和"cli"按列合并,得到名为"rt"的新数据框,其中包含了基因表达数据和临床信息。res.cox=coxph(Surv(futime, fustat) ~ . , data = rt)nom1=regplot(res.cox,plots = c("density", "boxes"),clickable=F,title="",points=TRUE,droplines=TRUE,observation=rt[2,],rank="sd",failtime = c(1,3,5),prfail = F)#进行生存分析。首先,使用coxph函数进行Cox比例风险模型拟合,其中"Surv(futime, fustat)"是生存时间和事件(死亡或事件)的生存对象。#拟合结果保存在"res.cox"变量中。#然后,使用"regplot"函数绘制列线图(Nomogram图),显示基因表达和临床因素与生存风险的关系。nomoRisk=predict(res.cox, data=rt, type="risk")rt=cbind(exp1, Nomogram=nomoRisk)outTab=rbind(ID=colnames(rt), rt)write.table(outTab, file="nomoRisk.txt", sep="t", col.names=F, quote=F)

#输出列线图的风险打分文件,计算并输出列线图(Nomogram图)中的风险得分。#使用predict函数基于Cox模型计算每个样本的风险得分,并将结果保存在"nomoRisk"变量中。#接着,将风险得分与表达数据合并为新的数据框"rt",再将数据框的列名和数据写入名为"nomoRisk.txt"的文本文件中。#校准曲线pdf(file="calibration.pdf", width=5, height=5)#绘制了校准曲线(Calibration Curve),用于评估Nomogram模型的预测性能#1年校准曲线f <- cph(Surv(futime, fustat) ~ Nomogram, x=T, y=T, surv=T, data=rt, time.inc=1)cal <- calibrate(f, cmethod="KM", method="boot", u=1, m=(nrow(rt)/3), B=1000)plot(cal, xlim=c(0,1), ylim=c(0,1),xlab="Nomogram-predicted OS (%)", ylab="Observed OS (%)", lwd=1.5, col="green", sub=F)#3年校准曲线f <- cph(Surv(futime, fustat) ~ Nomogram, x=T, y=T, surv=T, data=rt, time.inc=3)cal <- calibrate(f, cmethod="KM", method="boot", u=3, m=(nrow(rt)/3), B=1000)plot(cal, xlim=c(0,1), ylim=c(0,1), xlab="", ylab="", lwd=1.5, col="blue", sub=F, add=T)#5年校准曲线f <- cph(Surv(futime, fustat) ~ Nomogram, x=T, y=T, surv=T, data=rt, time.inc=5)cal <- calibrate(f, cmethod="KM", method="boot", u=5, m=(nrow(rt)/3), B=1000)plot(cal, xlim=c(0,1), ylim=c(0,1), xlab="", ylab="", lwd=1.5, col="red", sub=F, add=T)legend('bottomright', c('1-year', '3-year', '5-year'),col=c("green","blue","red"), lwd=1.5, bty = 'n')dev.off()#使用cph函数建立Cox模型,将Nomogram作为解释变量,并设定不同的时间间隔。#然后,使用calibrate函数生成校准曲线。代码将校准曲线绘制在一个PDF文件中,#分别绘制1年、3年和5年的校准曲线,并在图例中显示不同时间间隔的颜色。最后,通过dev.off()函数关闭PDF绘图设备。
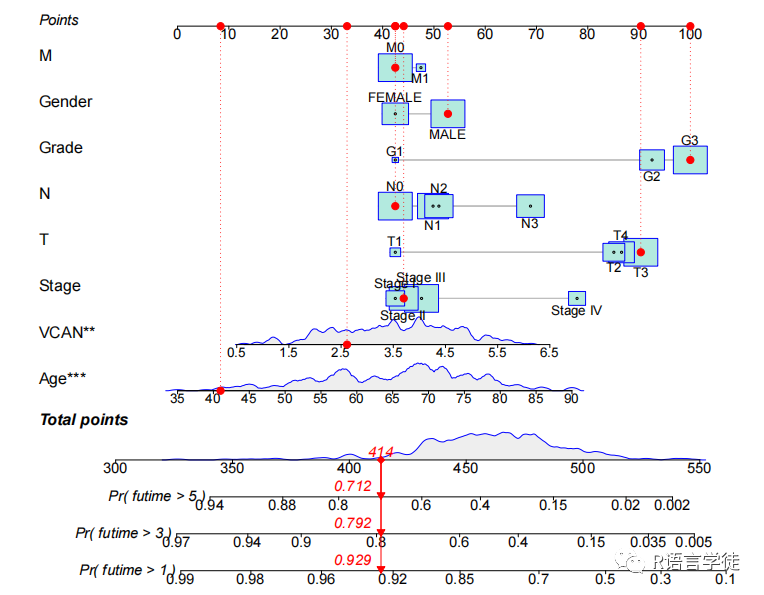

综合分析基因表达数据和临床数据,然后利用生存分析和校准曲线评估基因表达和临床信息对肿瘤患者预后的预测能力,预测患者预后以及发现潜在的生物标志物。下期将为你带来更多R语言的骚操作技巧,以下推荐的是一个多功能的生信平台。
★

