图表配色过于平庸?ggsci–为科研图表增色添彩
{ 点击蓝字,关注我们 }
大家好,今天小师妹要为大家介绍一个强大而美妙的R包——ggsci!想必各位学习生信的小伙伴都听说过SCI吧,能发一篇SCI的文章,那可是做生物科研的研究者梦寐以求的一件事呀!今天我们或许还没能够发SCI,但今天让小师妹带大家先来用用SCI的配色!
ggsci 是一个基于ggplot2的R包,旨在为数据可视化提供丰富多样的专业级颜色主题。ggplot2是R 中最受欢迎的数据可视化包,而ggsci则为ggplot2增添了更多的配色方案。默认的ggplot2颜色主题相对较少,而ggsci则为我们提供了多种专业设计的配色方案,为科研图表增色添彩,让我们的数据可视化更加吸引人和易读。无论是生物信息学研究者、医学科学家还是其他领域的数据分析人员,ggsci 都会是小伙伴们的得力助手。让小师妹带着大家一起来探索 ggsci 的魅力和应用场景吧!
使用 ggsci 包非常简单,只需要几行代码,我们就能将其应用到 ggplot2 图表中。
首先,小伙伴们需要安装并加载 ggsci 包:
if(!require("ggsci"))install.packages("ggsci")## 载入需要的程辑包:ggsci## Warning: 程辑包'ggsci'是用R版本4.2.3 来建造的if(!require("ggplot2"))install.packages("ggplot2")## 载入需要的程辑包:ggplot2## Warning: 程辑包'ggplot2'是用R版本4.2.3 来建造的安装完成后,我们就可以开始使用 ggsci 包了。让我们来看几个具体的应用示例,展示 ggsci 在科研图表中的魅力吧!
示例1:生物信息学领域
小师妹为大家使用代码模拟了一个基因表达矩阵,通过比较绘制基因的表达水平展示我们ggsci配色的优越性哦~
首先,大家可以先模拟数据:
# 加载所需的包library(matrixStats)# 设置随机种子以保证结果的可复现性set.seed(123)# 创建模拟基因表达矩阵num_samples <- 5num_genes <- 10# 使用正态分布随机生成基因的表达值expression_matrix <- matrix(rnorm(num_samples * num_genes, mean = 10, sd = 2), nrow = num_genes)# 添加行和列名称rownames(expression_matrix) <- paste0("Gene", 1:num_genes)colnames(expression_matrix) <- paste0("Sample", 1:num_samples)# 预览基因表达矩阵print(head(expression_matrix))## Sample1 Sample2 Sample3 Sample4 Sample5## Gene1 8.879049 12.448164 7.864353 10.852928 8.610586## Gene2 9.539645 10.719628 9.564050 9.409857 9.584165## Gene3 13.117417 10.801543 7.947991 11.790251 7.469207## Gene4 10.141017 10.221365 8.542218 11.756267 14.337912## Gene5 10.258575 8.888318 8.749921 11.643162 12.415924## Gene6 13.430130 13.573826 6.626613 11.377281 7.753783# 将基因表达矩阵转换成长格式(long format)expression_df <- as.data.frame(expression_matrix)expression_df$Gene <- rownames(expression_df)expression_long <- tidyr::gather(expression_df, key = "Sample", value = "Expression", -Gene)接下来,我们可以使用 ggplot2 来绘制基因的表达水平,并使用 ggsci 提供的配色方案:# 绘制默认配色的热图ggplot(expression_long, aes(x = Sample, y = Gene, fill = Expression)) + geom_tile() + scale_fill_gradient(low = "white", high = "blue") + theme_minimal() + theme(axis.text.x = element_text(angle = 45, hjust = 1))
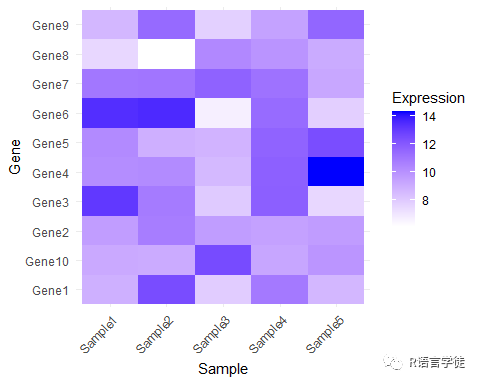
# 绘制使用 ggsci 配色的热图ggplot(expression_long, aes(x = Sample, y = Gene, fill = Expression)) + geom_tile() + ggsci::scale_fill_gsea() + theme_minimal() + theme(axis.text.x = element_text(angle = 45, hjust = 1))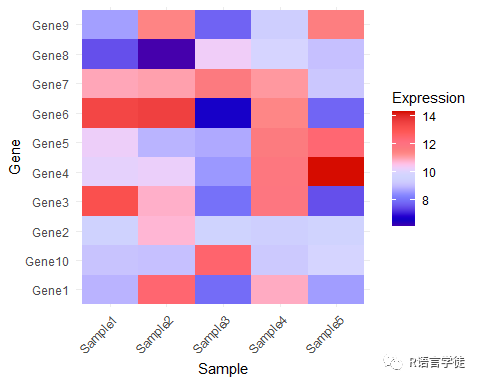
示例2:医学科学领域
在医学科学领域,我们经常需要绘制生存曲线来分析患者的生存时间。我们可以使用 ggplot2 和 ggsci 包来绘制生存曲线,并选择合适的配色方案
。
首先小师妹先为大家准备一套生存曲线的模拟数据。我们假设有两组数据,每组数据包含了一些观测时间点和事件(生存或死亡)信息。为了模拟数据,我们使用 rnorm() 函数生成随机的生存时间,并使用 rbinom() 函数生成随机的事件信息(0代表生存,1代表死亡)。
# 加载所需的包library(survival)library(survminer)## Warning: 程辑包'survminer'是用R版本4.2.3 来建造的## 载入需要的程辑包:ggpubr## ## 载入程辑包:'survminer'## The following object is masked from 'package:survival':## ## myeloma# 设置随机种子以保证结果的可复现性set.seed(123)# 创建模拟数据num_samples <- 100# 第一组数据group1_time <- rnorm(num_samples, mean = 50, sd = 10)group1_event <- rbinom(num_samples, size = 1, prob = 0.7)# 第二组数据group2_time <- rnorm(num_samples, mean = 70, sd = 15)group2_event <- rbinom(num_samples, size = 1, prob = 0.5)# 合并数据data <- data.frame(time = c(group1_time, group2_time), event = c(group1_event, group2_event), group = factor(rep(c("Group1", "Group2"), each = num_samples)))# 预览模拟的生存曲线数据print(head(data))## time event group## 1 44.39524 1 Group1## 2 47.69823 0 Group1## 3 65.58708 1 Group1## 4 50.70508 1 Group1## 5 51.29288 1 Group1## 6 67.15065 0 Group1接下来,我们可以使用 ggplot2 和 ggsci 来绘制生存曲线,并使用 ggsci 提供的配色方案:# 使用 Kaplan-Meier 方法计算生存曲线survfit_obj <- survfit(Surv(time, event) ~ group, data = data)# 绘制生存曲线图,使用 ggplot2 默认配色ggsurvplot(survfit_obj, data = data, palette = "default")
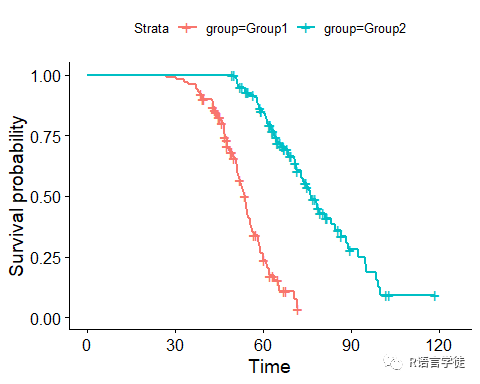
# 绘制生存曲线图,使用 ggsci 提供的配色方案ggsurvplot(survfit_obj, data = data, palette = "jama")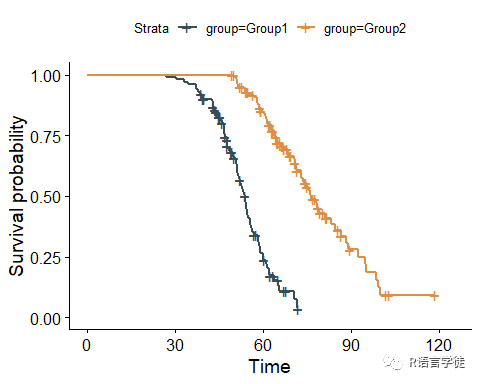
ggsurvplot(survfit_obj, data = data, palette = "igv")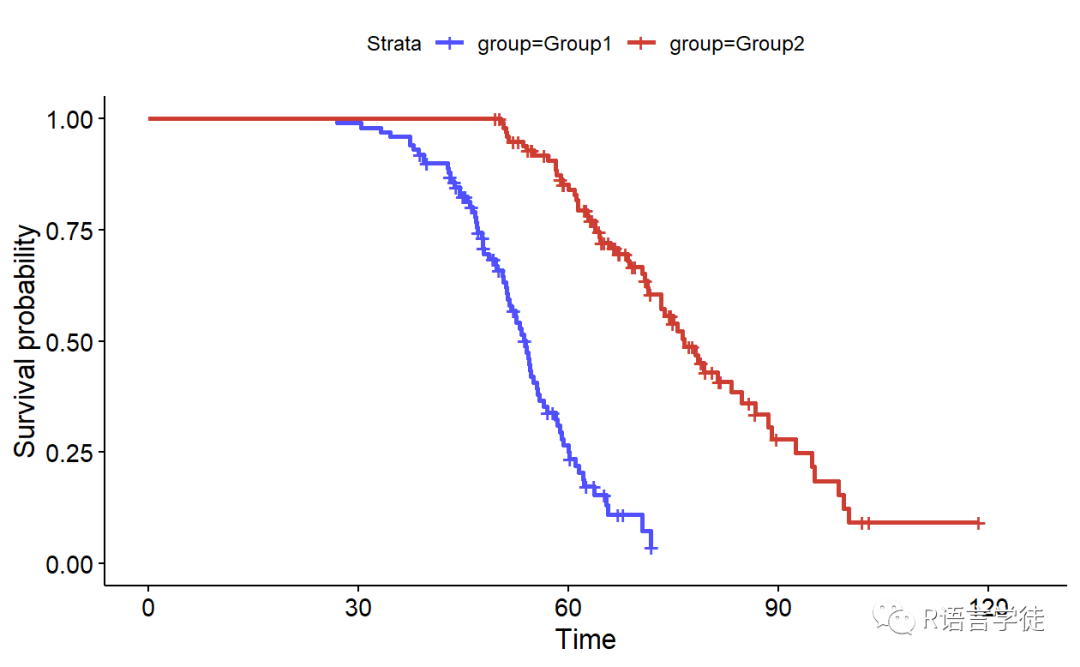
在这个示例中,我们使用了 ggsci 提供的 “jama”和“igv” 配色方案,这个方案在生存曲线中具有很好的辨识度,能够帮助我们更好地理解患者的生存情况。
今天小师妹的分享就到这里啦!有兴趣的小伙伴可以进行这个网站
https://www.rdocumentation.org/packages/ggsci/versions/3.0.0查看所有配色及参数哦!不知道ggsci包是否对你的配色有帮助呢?希望小伙伴们以后都可以有属于自己的一套配色,绘制出只属于自己的图表!加油!
如果小伙伴有其他数据分析需求,可以尝试使用本公司新开发的生信分析小工具云平台,零代码完成分析,非常方便奥,
云平台网址为:
http://www.biocloudservice.com/home.html。


END

