基因探险:用WGCNA解密基因表达之谜
点击蓝字 关注我们
hello,今天小师妹将教大家学习如何使用WGCNA包对基因表达数据进行分析。感兴趣的话就和小师妹一起看下去吧!
什么是WGCNA?
WGCNA(Weighted Gene Co-expression Network Analysis)是一个基于R语言的生物信息学软件包,用于分析基因共表达网络。它可以帮助我们理解基因之间的相互作用、功能模块和调控机制。
WGCNA包提供了一套丰富而全面的工具,用于构建、可视化和解释基因共表达网络。它通过计算基因表达数据中基因之间的相关性,将高相关性的基因聚类成模块,并识别出模块与特定生物学过程或性状之间的关联。
使用WGCNA包,你可以执行以下主要任务:
1构建基因共表达网络:
WGCNA通过计算基因间的相关系数来构建基因共表达网络。它可以根据不同的相关系数方法(如Pearson相关系数、Spearman相关系数等)进行计算,并根据用户的选择创建一个加权网络。
2模块检测:
WGCNA将高相关性的基因聚类成模块,每个模块代表一个具有类似表达模式的基因集合。这些模块可以帮助我们识别生物学功能相关的基因组。
3模块与性状关联:
WGCNA可以通过计算模块与给定性状之间的关联来鉴定与性状相关的模块。这有助于我们理解基因与特定性状(如疾病状态、表型变化等)之间的关系。
4数据可视化:
WGCNA提供了多种可视化工具,使用户能够直观地探索和呈现基因共表达网络、模块和模块与性状的关联。这些工具包括热图、网络图、散点图等。
WGCNA在生物学研究中广泛应用,特别是在发现和解释基因调控网络方面。它可以帮助揭示疾病机制、鉴定生物标志物、预测药物响应等。通过使用WGCNA包,研究人员可以更好地理解基因表达数据,并从中获得有关生物学过程的深入洞察。
WGCNA进行基因表达数据分析
安装与导入WGCNA包
BiocManager::install(“WGCNA”) library(‘WGCNA’)# 在读入数据时,遇到字符串之后,不将其转换为factors,仍然保留为字符串格式options(stringsAsFactors = FALSE)
数据下载与导入
首先我们来看一下数据的文件格式:

然后,我们需要导入数据:
# 导入示例数据,这里填写自己存放表达矩阵的路径femData = read.csv("LiverFemale3600.csv") # femData代表该文件你可以使用dim函数查看数据的形状(行数和列数):
dim(femData) 你可以使用names函数查看每一列的标题(列标):
names(femData) 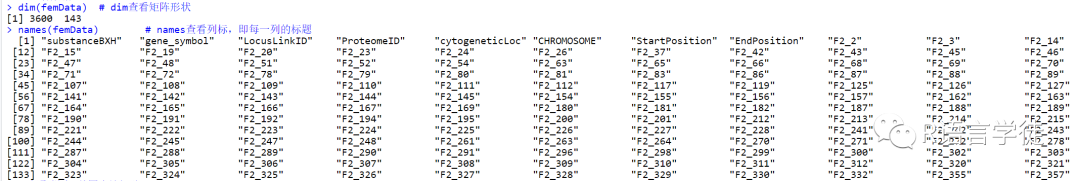
数据处理
接下来,我们将提取样本-基因表达矩阵:
datExpr0 = as.data.frame(t(femData[, -c(1:8)]))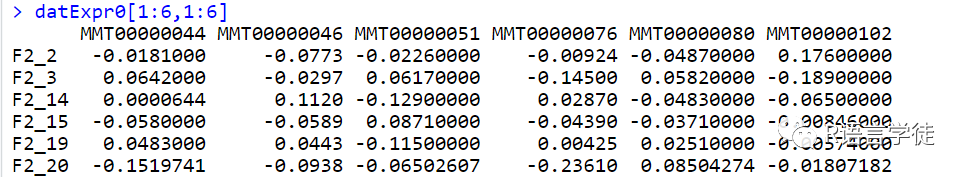
如果你不需要保留基因名作为列标,你可以删除下面这行代码:
rownames(datExpr0) = names(femData)[-c(1:8)]接下来,我们可以检查是否有太多缺失值的基因和样本:
egsg = goodSamplesGenes(datExpr0, verbose = 3)gsg$allOK
如果存在缺失值太多的基因或样本,你可以选择删除这些基因或样本。以下是一个可选步骤,它会打印被删除的基因和样本的名称,并删除不满足要求的基因和样本:
if (!gsg$allOK) { if (sum(!gsg$goodGenes) > 0) { printFlush(paste("Removing genes:", paste(names(datExpr0)[!gsg$goodGenes], collapse = ","))) } if (sum(!gsg$goodSamples) > 0) { printFlush(paste("Removing samples:", paste(rownames(datExpr0)[!gsg$goodSamples], collapse = ","))) } datExpr0 = datExpr0[gsg$goodSamples, gsg$goodGenes]}接下来,我们将使用聚类方法构建一个样本树:
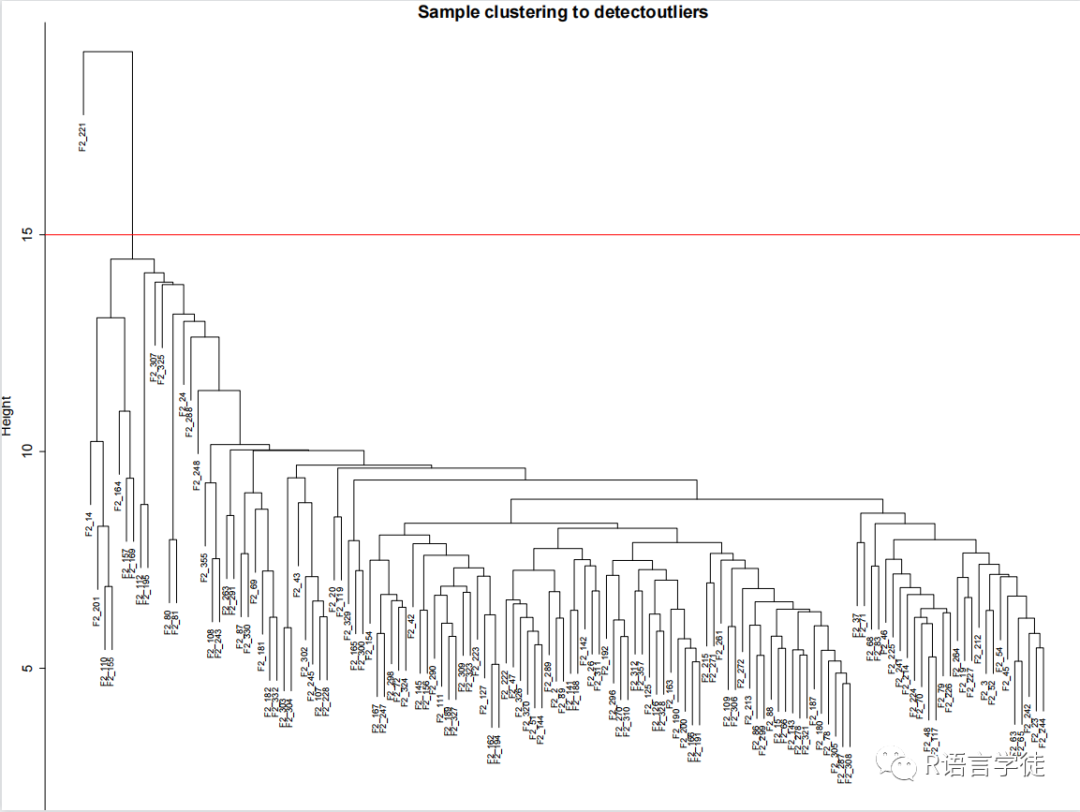
sampleTree = hclust(dist(datExpr0), method = "average")然后,我们可以绘制样本树。以下代码将打开一个图形输出窗口,并绘制样本树:
sizeGrWindow(12, 9)par(cex = 0.6)par(mar = c(0, 4, 2, 0))plot(sampleTree, main = "Sample clustering to detect outliers", sub = "", xlab = "", cex.lab = 1.5, cex.axis = 1.5, cex.main = 2)如果你想保存图形,你可以取消注释以下代码,并设置保存的文件路径:
我们一起来看一下绘制的图片是什么样吧!
接下来,我们可以绘制阈值切割线并确定切割后的集群:
abline(h = 15, col = "red")clust = cutreeStatic(sampleTree, cutHeight = 15, minSize = 10)table(clust)最后,我们可以根据切割后的集群选择保留的样本,并将其放入新的数据矩阵datExpr中:
keepSamples = (clust == 1)datExpr = datExpr0[keepSamples, ]这就是一个使用WGCNA包对基因表达数据进行分析的简单示例。大家可以根据自己的需求和数据进行相应的修改和扩展哦。希望本教程能够帮助你开始使用WGCNA包进行基因表达数据分析!快和小师妹一起学起来吧!

